
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 7,134,361 – 7,134,501 |
| Length | 140 |
| Max. P | 0.713194 |

| Location | 7,134,361 – 7,134,461 |
|---|---|
| Length | 100 |
| Sequences | 4 |
| Columns | 100 |
| Reading direction | reverse |
| Mean pairwise identity | 95.83 |
| Mean single sequence MFE | -26.43 |
| Consensus MFE | -22.24 |
| Energy contribution | -22.93 |
| Covariance contribution | 0.69 |
| Combinations/Pair | 1.09 |
| Mean z-score | -1.71 |
| Structure conservation index | 0.84 |
| SVM decision value | -0.05 |
| SVM RNA-class probability | 0.509519 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 7134361 100 - 23771897 UAUAUUGAUGUAUGUCCCCGUGUGCGUGUCUACGUUAUGAGGCAGUCGUGUGCUCAUCUAUGUAUAUAGGAUAUAUACCGAGAUAUACAAUCGAAGAAUA ....((((((((((((..((.(((..(((((((((.((((((((....))).)))))..)))).....)))))..))))).))))))).)))))...... ( -26.50) >DroSec_CAF1 49138 100 - 1 UAUAUUGAUGUGUGUACCCGUGUGCGUGUCUACGUUAUGAGGCAGUCGUGUGCUCAUGUAUGUAUAUAGGAUAUAUACCGAGAUAUACAAACGAAGAAUA ((((((...(((((((((..((((((((.......(((((((((....))).))))))))))))))..)).)))))))...))))))............. ( -24.71) >DroSim_CAF1 49535 100 - 1 UAUAUUGAUGUGUGUCCCCGUGUGCGUGUCUACGUUAUGAGGCAGUCGUGUGCUCAUGUAUGUAUAUAGGAUAUAUACCGAGAUAUACAAUCGAAGAAUA ....((((((((((((..((.(((..((((((((((((((((((....))).)))))).)))).....)))))..))))).))))))).)))))...... ( -27.80) >DroYak_CAF1 51267 100 - 1 UAUAUUUAUGUGUGUCCGUGUGUACGUGUCUACGUUAUGAGGCAGUCGUGUGCUCAUCUAUGUAUAUAGGAUAUAUACCGAGAUAUACAAUCGAAGAAUA ........(((((((((((((((((.(((.(((((.((((((((....))).)))))..))))).))).).)))))).)).))))))))........... ( -26.70) >consensus UAUAUUGAUGUGUGUCCCCGUGUGCGUGUCUACGUUAUGAGGCAGUCGUGUGCUCAUCUAUGUAUAUAGGAUAUAUACCGAGAUAUACAAUCGAAGAAUA ....((((((((((((..((.(((..((((((((((((((((((....))).)))))).)))).....)))))..))))).))))))).)))))...... (-22.24 = -22.93 + 0.69)



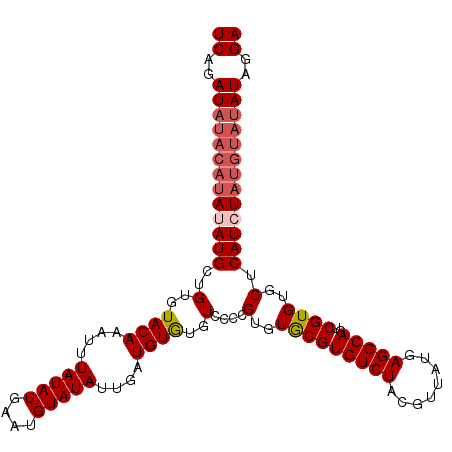
| Location | 7,134,390 – 7,134,501 |
|---|---|
| Length | 111 |
| Sequences | 4 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 93.99 |
| Mean single sequence MFE | -28.65 |
| Consensus MFE | -23.77 |
| Energy contribution | -25.40 |
| Covariance contribution | 1.63 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.85 |
| Structure conservation index | 0.83 |
| SVM decision value | 0.38 |
| SVM RNA-class probability | 0.713194 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 7134390 111 - 23771897 UCAGAUAUACAUAUAUGCUGUGUACAAAUUUAUAUGAAUGUAUAUUGAUGUAUGUCCCCGUGUGCGUGUCUACGUUAUGAGGCAGUCGUGUGCUCAUCUAUGUAUAUAGGA ((..(((((((((.(((..(..(((.....(((((.(((....))).)))))((.((.((((.((((....)))))))).))))...)))..).))).)))))))))..)) ( -27.10) >DroSec_CAF1 49167 111 - 1 UCAGAUAUACAUAUAUGCUGUGUACAAAUUUAUAUGAAUGUAUAUUGAUGUGUGUACCCGUGUGCGUGUCUACGUUAUGAGGCAGUCGUGUGCUCAUGUAUGUAUAUAGGA ((..(((((((((((((..(..(((.....((((((...((((((......)))))).)))))).((.((........)).))....)))..).)))))))))))))..)) ( -31.70) >DroSim_CAF1 49564 111 - 1 UCAGAUAUACAUAUAUGCUGUGUACAAAUUUAUAUGAAUGUAUAUUGAUGUGUGUCCCCGUGUGCGUGUCUACGUUAUGAGGCAGUCGUGUGCUCAUGUAUGUAUAUAGGA ((..(((((((((((((..(..(((.....(((((.(((....))).)))))((.((.((((.((((....)))))))).))))...)))..).)))))))))))))..)) ( -30.80) >DroYak_CAF1 51296 105 - 1 UCAGAU------AUAUGCUGUGUACAAAUUUAUAUGAAUGUAUAUUUAUGUGUGUCCGUGUGUACGUGUCUACGUUAUGAGGCAGUCGUGUGCUCAUCUAUGUAUAUAGGA ......------.....(((((((((....(((((((((....))))))))).....(.(..(((((((((........)))))..))))..).).....))))))))).. ( -25.00) >consensus UCAGAUAUACAUAUAUGCUGUGUACAAAUUUAUAUGAAUGUAUAUUGAUGUGUGUCCCCGUGUGCGUGUCUACGUUAUGAGGCAGUCGUGUGCUCAUCUAUGUAUAUAGGA ((..(((((((((((((..(..((((....(((((....)))))....))))..)....(..(((((((((........)))))..))))..).)))))))))))))..)) (-23.77 = -25.40 + 1.63)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:35:25 2006