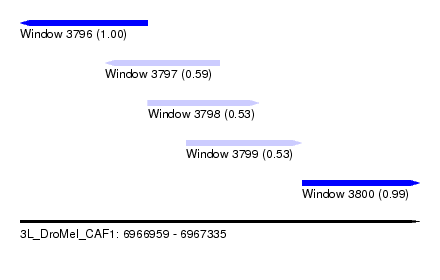
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 6,966,959 – 6,967,335 |
| Length | 376 |
| Max. P | 0.996538 |

| Location | 6,966,959 – 6,967,079 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.08 |
| Mean single sequence MFE | -51.32 |
| Consensus MFE | -45.48 |
| Energy contribution | -45.80 |
| Covariance contribution | 0.32 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.12 |
| Structure conservation index | 0.89 |
| SVM decision value | 2.71 |
| SVM RNA-class probability | 0.996538 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 6966959 120 - 23771897 UCCAGGGAUCGGCGAAUCCUUGACCACCAGCCGGCGAGACGCCAGUGGCUCCAGCUGCACUGUAAUCGUAGCUAGCAAAGGCAGGCGAGGCGUAAUAGUUGCCAUUGUCCUGGUGGGCAA ..(((((((......))))))).(((((((((((((...))))....(((..((((((.........)))))))))...))).(((((((((.......)))).))))).)))))).... ( -48.60) >DroSec_CAF1 36424 120 - 1 UCCAGGGAUCGGAGAAGCCUUGACCACCAGCCGGCGAGACGCCGCUGGCUCCAGCUGCACUAUAAUCGUAGCUAGCGAAGGCAGGCGAGGCGUAAUAGUUGCCAUUAUCCUGGUGGGAAA .((((((.........((((((.((...((((((((......))))))))..((((((.........)))))).((....)).))))))))(((.....))).....))))))....... ( -46.10) >DroSim_CAF1 37213 120 - 1 UCCAGGGAUCGGAGAAGCCUUGACCACCAGCCGGCGAGACGCCACUGGCUCCAGCUGCACUGUAAUCGUAGCUAGCGAAGGCGGGCGAGGCGUAAUAGUUGCCAUUGUCCUGGUGGGAAA ..(((((..........))))).(((((((((((((...))))....(((..((((((.........)))))))))...)))((((((((((.......)))).)))))))))))).... ( -48.90) >DroEre_CAF1 37363 120 - 1 UCCAGGGAUCGCCGAAUCCUUGACCGCCAGCCGGCGACACGCCAUUCGCUCCAGCCGCACUGUAAUCGUAGCUGGCGAAGGCGGGCGAGGCGUAAUAGUUGCCGUUGUCCUGGUGGCAAA ..(((((((......))))))).(((((((.((((((((((((..((((((((((..(.........)..))))((....)))))))))))))....))))))).....))))))).... ( -55.40) >DroYak_CAF1 38309 120 - 1 UCCAGGGAUCGGCGAAUCCUUGACCACCAGCUGGUGACACGCUGCUCGCUCCAGCUGCGCCGUAAUCGUAGCUGGCGAAGGCGGGCGAGGCGUAAUAGUUGCCAUUGUCCUGGUGGCAAA ..(((((((......))))))).(((((((.(((..(((((((((((((((((((((((.......)))))))))....)))))))..)))))....))..))).....))))))).... ( -57.60) >consensus UCCAGGGAUCGGCGAAUCCUUGACCACCAGCCGGCGAGACGCCACUGGCUCCAGCUGCACUGUAAUCGUAGCUAGCGAAGGCGGGCGAGGCGUAAUAGUUGCCAUUGUCCUGGUGGGAAA ..(((((((......))))))).(((((((((((((...))))....(((..((((((.........)))))))))...)))((((((((((.......)))).)))))))))))).... (-45.48 = -45.80 + 0.32)

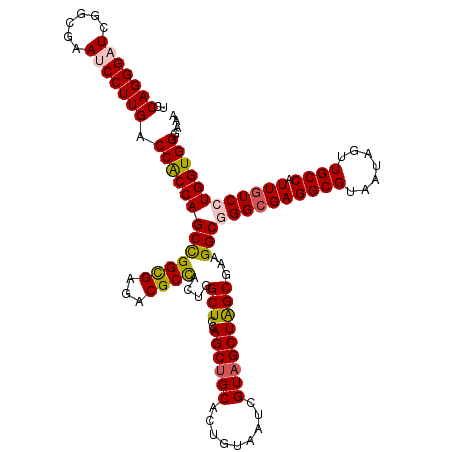
| Location | 6,967,039 – 6,967,147 |
|---|---|
| Length | 108 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 88.05 |
| Mean single sequence MFE | -54.90 |
| Consensus MFE | -42.83 |
| Energy contribution | -43.23 |
| Covariance contribution | 0.40 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.53 |
| Structure conservation index | 0.78 |
| SVM decision value | 0.12 |
| SVM RNA-class probability | 0.594233 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 6967039 108 - 23771897 CUGGGGCAGGGGA------CCCACGCCCAUGCCCACGC------CCACCGACGACGAGCCACUGUGGGCGGCAUGGGCGUUCCAGGGAUCGGCGAAUCCUUGACCACCAGCCGGCGAGAC ....(((.((((.------...(((((((((((...((------((((.(...........).)))))))))))))))))..(((((((......))))))).)).)).)))........ ( -49.90) >DroSec_CAF1 36504 108 - 1 CUGGGGCAGGGCG------CCCACGCCCAUGCCCACGC------CCACCGACGACGGGCCACUGUGGGCGGCAUGGGUGUUCCAGGGAUCGGAGAAGCCUUGACCACCAGCCGGCGAGAC (((((((((((((------((((.(((...((((((((------((.........))))....))))))))).))))).((((.......))))..)))))).)).)))).......... ( -51.40) >DroSim_CAF1 37293 108 - 1 CUGGGGCAGGGCG------CCCACGCCCAUGCCCACGC------CCACCGACGAGGAGCCACUGUGGGCGGCAUGGGCGUUCCAGGGAUCGGAGAAGCCUUGACCACCAGCCGGCGAGAC ((((((((((((.------((((((((((((((...((------((((.....((......)))))))))))))))))))....))).........)))))).)).)))).......... ( -53.30) >DroEre_CAF1 37443 120 - 1 CUGGGGCAGCGGGCUCACGCCCACACCCAUGCCCACGCCCAUGCCCACCGACGAGGCGCCACUGUGGGCGGCAUGGGCGUUCCAGGGAUCGCCGAAUCCUUGACCGCCAGCCGGCGACAC ...((((((.((((....)))).).....)))))(((((((((((..((.....)).(((......))))))))))))))..(((((((......)))))))..((((....)))).... ( -58.20) >DroYak_CAF1 38389 114 - 1 CUGGGGCAGUGGGCCCACGCCCACGCCCAUGCCCACGC------CCACCGUCGAGGAGCCACUGUGGGCGGCAUGGGCGUUCCAGGGAUCGGCGAAUCCUUGACCACCAGCUGGUGACAC (((((((.((((((....))))))(((((((((...((------((((.((.(.....).)).))))))))))))))))))))))((((......)))).....((((....)))).... ( -61.70) >consensus CUGGGGCAGGGGG______CCCACGCCCAUGCCCACGC______CCACCGACGAGGAGCCACUGUGGGCGGCAUGGGCGUUCCAGGGAUCGGCGAAUCCUUGACCACCAGCCGGCGAGAC ((((((((((((.......((((((((((((((..............((.....)).(((......))))))))))))))....))).........)))))).)).)))).......... (-42.83 = -43.23 + 0.40)

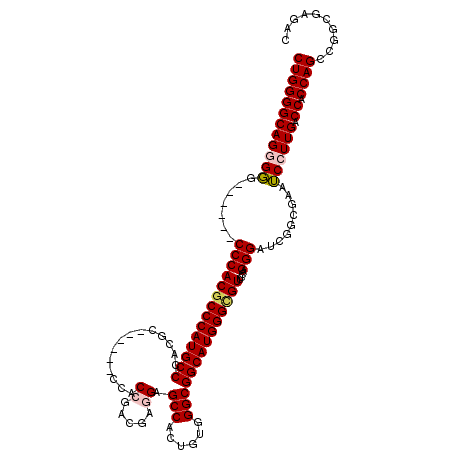
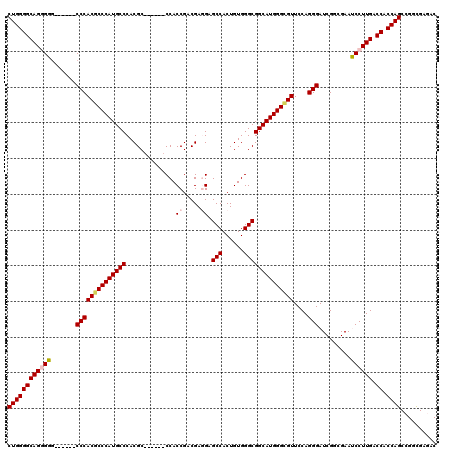
| Location | 6,967,079 – 6,967,184 |
|---|---|
| Length | 105 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 87.94 |
| Mean single sequence MFE | -54.60 |
| Consensus MFE | -37.55 |
| Energy contribution | -38.82 |
| Covariance contribution | 1.27 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.84 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.00 |
| SVM RNA-class probability | 0.533683 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 6967079 105 + 23771897 ACGCCCAUGCCGCCCACAGUGGCUCGUCGUCGGUGG------GCGUGGGCAUGGGCGUGGG------UCCCCUGCCCCAGUAUACGAACAUAUCCUGCCUGGCAG---CCGGAGGCAAUG (((((((((((((((((...(((.....))).))))------))...)))))))))))..(------(((((((((.(((.(((......))).)))...)))))---..)).))).... ( -50.20) >DroSec_CAF1 36544 105 + 1 ACACCCAUGCCGCCCACAGUGGCCCGUCGUCGGUGG------GCGUGGGCAUGGGCGUGGG------CGCCCUGCCCCAGUAUACGAACAUAUCCUGCCUGGCAG---CCGGCGGCAAUC ...((((((((((((((....((((..(...)..))------))))))))...))))))))------(((((((((.(((.(((......))).)))...)))))---..))))...... ( -49.50) >DroSim_CAF1 37333 105 + 1 ACGCCCAUGCCGCCCACAGUGGCUCCUCGUCGGUGG------GCGUGGGCAUGGGCGUGGG------CGCCCUGCCCCAGUAUACGAACAUAUCCUGCCUGGCAG---CCGGCGGCAAUC (((((((((((((((((...(((.....))).))))------))...))))))))))).(.------(((((((((.(((.(((......))).)))...)))))---..)))).).... ( -54.60) >DroEre_CAF1 37483 117 + 1 ACGCCCAUGCCGCCCACAGUGGCGCCUCGUCGGUGGGCAUGGGCGUGGGCAUGGGUGUGGGCGUGAGCCCGCUGCCCCAGUACACGAACAUAUCCUGCCUGGCAG---CCGGUGGCAAUC .((((..((((((((((...((((...)))).))))))(((..((((..(.((((.((((((....))))))...)))))..))))..))).........)))).---..))))...... ( -61.10) >DroYak_CAF1 38429 114 + 1 ACGCCCAUGCCGCCCACAGUGGCUCCUCGACGGUGG------GCGUGGGCAUGGGCGUGGGCGUGGGCCCACUGCCCCAGUACACGAACAUAUCCUGCCUGGCAGCAGCAGGCGGCAAUC ..(((((((((((((((.((.(.....).)).))))------))...)))))))))((((((....))))))((((...((......)).......(((((.......)))))))))... ( -57.60) >consensus ACGCCCAUGCCGCCCACAGUGGCUCCUCGUCGGUGG______GCGUGGGCAUGGGCGUGGG______CCCCCUGCCCCAGUAUACGAACAUAUCCUGCCUGGCAG___CCGGCGGCAAUC (((((((((((((((((...(((.....))).))))......))...)))))))))))((((....))))..((((((.((......)).....((((...)))).....)).))))... (-37.55 = -38.82 + 1.27)



| Location | 6,967,115 – 6,967,224 |
|---|---|
| Length | 109 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 88.80 |
| Mean single sequence MFE | -51.39 |
| Consensus MFE | -42.70 |
| Energy contribution | -41.98 |
| Covariance contribution | -0.72 |
| Combinations/Pair | 1.13 |
| Mean z-score | -1.35 |
| Structure conservation index | 0.83 |
| SVM decision value | 0.00 |
| SVM RNA-class probability | 0.533938 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 6967115 109 + 23771897 --GCGUGGGCAUGGGCGUGGG------UCCCCUGCCCCAGUAUACGAACAUAUCCUGCCUGGCAG---CCGGAGGCAAUGUGAAUGGCUCGGCUACCACGCCCCCGCUGGCCCACUCCGC --..((((((..((((((((.------......(((.(((.(((......))).)))...)))((---(((.((.((.......)).))))))).))))))))......))))))..... ( -46.70) >DroSec_CAF1 36580 109 + 1 --GCGUGGGCAUGGGCGUGGG------CGCCCUGCCCCAGUAUACGAACAUAUCCUGCCUGGCAG---CCGGCGGCAAUCUGAAUGGCUCGGCCACCACGCCUCCGUUGGCCCAUUCCGC --((((((((..((((((((.------(((((((((.(((.(((......))).)))...)))))---..))))..........((((...))))))))))))......)))))...))) ( -45.90) >DroSim_CAF1 37369 109 + 1 --GCGUGGGCAUGGGCGUGGG------CGCCCUGCCCCAGUAUACGAACAUAUCCUGCCUGGCAG---CCGGCGGCAAUCUGAAUGGCUCGGCCACCACGCCUCCGUUGGCCCACUCCGC --..((((((..((((((((.------(((((((((.(((.(((......))).)))...)))))---..))))..........((((...))))))))))))......))))))..... ( -47.50) >DroEre_CAF1 37523 117 + 1 GGGCGUGGGCAUGGGUGUGGGCGUGAGCCCGCUGCCCCAGUACACGAACAUAUCCUGCCUGGCAG---CCGGUGGCAAUCUGAAUGGCACGGCCACCACGCCCCCGCUGGCCCACUCCAC .((.((((((..((((((((((....))))((((((.(((..............)))...)))))---).((((((...((....))....))))))))))))......)))))).)).. ( -58.54) >DroYak_CAF1 38465 118 + 1 --GCGUGGGCAUGGGCGUGGGCGUGGGCCCACUGCCCCAGUACACGAACAUAUCCUGCCUGGCAGCAGCAGGCGGCAAUCUGAAUGGCUCGGCCACCACGCCCCCGCUGGCCCACUCCAC --..((((((...((((.(((((((((((..((((.((((..((.((.....)).)).))))..))))..)))(((...((....))....))).)))))))).)))).))))))..... ( -58.30) >consensus __GCGUGGGCAUGGGCGUGGG______CCCCCUGCCCCAGUAUACGAACAUAUCCUGCCUGGCAG___CCGGCGGCAAUCUGAAUGGCUCGGCCACCACGCCCCCGCUGGCCCACUCCGC ....((((((..((((((((............((((((.((......)).....((((...)))).....)).)))).......((((...))))))))))))......))))))..... (-42.70 = -41.98 + -0.72)

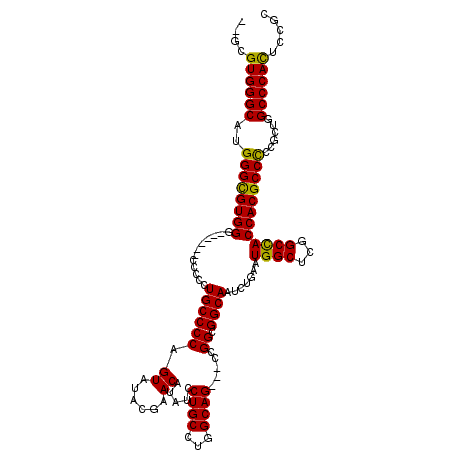
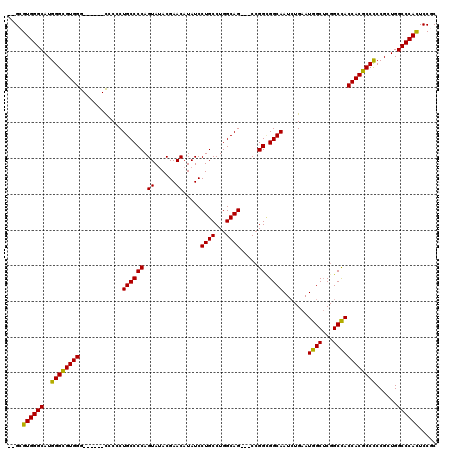
| Location | 6,967,224 – 6,967,335 |
|---|---|
| Length | 111 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 89.44 |
| Mean single sequence MFE | -51.06 |
| Consensus MFE | -40.66 |
| Energy contribution | -41.46 |
| Covariance contribution | 0.80 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.51 |
| Structure conservation index | 0.80 |
| SVM decision value | 2.03 |
| SVM RNA-class probability | 0.986249 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 6967224 111 + 23771897 ACUGGGAAUGGCCCCAUC---GGCAUCAUCCUCAAGUUCGCC------ACUGGGAGCGGCGGCGACGCUUCAAAGCGAUUAUGCGCCAACCGCGAGUCUCGUGGCCGCUGGCUAUUCCUA ..((((((((((((((..---(((...............)))------..))).((((((.((((.(((....)))((((.((((.....)))))))))))).))))))))))))))))) ( -49.46) >DroSec_CAF1 36689 111 + 1 CCUGGGAAUGGCCCCAUC---GGCAUCAUCCUCAAGUUCGCC------ACUGGGAGCGGCGGCGACGCUUCAAAGCGAUUAUGCGCCAACCGCGAGUCUCGUGGCCGCUGGCUAUUCCUA ..((((((((((((((..---(((...............)))------..))).((((((.((((.(((....)))((((.((((.....)))))))))))).))))))))))))))))) ( -49.46) >DroSim_CAF1 37478 111 + 1 CCUGGGAAUGGCCCCAUC---GGCAUCAUCCUCAAGUUCGCC------ACUGGGAGCGGCGGCGACGCUUCAAAGCGAUUAUGCGCCAACCGCGAGUCUCGUGGCCGCUGGCUAUUCCUA ..((((((((((((((..---(((...............)))------..))).((((((.((((.(((....)))((((.((((.....)))))))))))).))))))))))))))))) ( -49.46) >DroEre_CAF1 37640 114 + 1 CCUGGGCAUGGCCCCAUCAGCGGCCCCAACCUCCAGUUCGCC------CCUGGGAGCGGCGGCGACGCUUCAAAGCGACUAUGCGCCGAACGCGAGUCUCGUGGCCGCGGGCUAUUCCUA ..((((.(((((((..((((.(((...(((.....))).)))------.))))..(((((.((((.(((....)))((((.((((.....)))))))))))).)))))))))))).)))) ( -52.00) >DroYak_CAF1 38583 120 + 1 CCUGGGCAUGGCCCCAUCAUCGCUCCCACCCUCAAGUUCGCCAGUGAGCCUGGGAGCGGCGGCGUCGCUUCAAAGCGACUAUGCGCCAACCGCGAGUCUCGUGGCAGCGGGCUACUCCUA ..((((..((((((......((((((((..((((.(....)...))))..))))))))(((((((((((....)))))).....)))..(((((.....)))))..))))))))..)))) ( -54.90) >consensus CCUGGGAAUGGCCCCAUC___GGCAUCAUCCUCAAGUUCGCC______ACUGGGAGCGGCGGCGACGCUUCAAAGCGAUUAUGCGCCAACCGCGAGUCUCGUGGCCGCUGGCUAUUCCUA ..((((((((((((((.....(((...............)))........)))..(((((.((((.(((....)))((((.((((.....)))))))))))).))))).))))))))))) (-40.66 = -41.46 + 0.80)

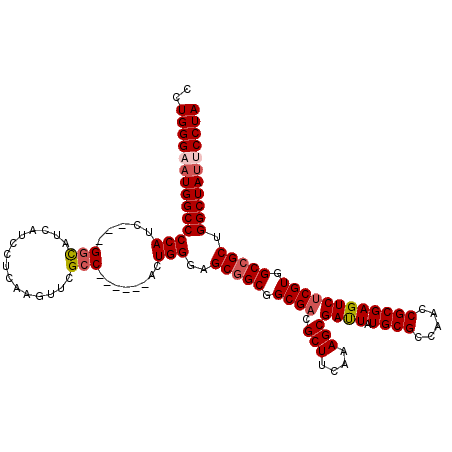
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:32:56 2006