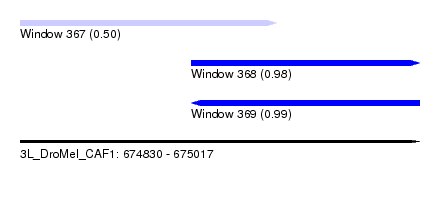
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 674,830 – 675,017 |
| Length | 187 |
| Max. P | 0.992084 |

| Location | 674,830 – 674,950 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.82 |
| Mean single sequence MFE | -39.52 |
| Consensus MFE | -25.35 |
| Energy contribution | -25.88 |
| Covariance contribution | 0.53 |
| Combinations/Pair | 1.23 |
| Mean z-score | -1.25 |
| Structure conservation index | 0.64 |
| SVM decision value | -0.08 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 674830 120 + 23771897 CAAUCGCUCCAAAAUGGAACUGCAGGCACUGGAGCACAAGAGCUUCCAGGUGGACACUGCGAGCAGCGGCGCGGCGACCAACGAGGAGGACGCCUCCUCCUGCGGCUGCUAGCAGUUGGG ((((.(((((.....)))..(((((.(.(((((((......)).)))))...)...)))))((((((.((.((........))(((((((....))))))))).)))))).)).)))).. ( -49.70) >DroVir_CAF1 37305 114 + 1 CAAUCGUUCCAAAAUGGAGCUGCAGGCACUCGAGCAACAGAGUUUUCAAAUAGACAAGGU---CAACGAGACAGAGAUUAACGAGGAG---GCCUCCUCGUGCAGCUGCUAGCAGUUGGG ........((((..(((((((((.....(((..........((((......))))...((---(.....))).)))....(((((((.---...))))))))))))).)))....)))). ( -35.30) >DroPse_CAF1 36894 114 + 1 CAAUCGCUCCAAAAUGGAGCUGCAGGCUCUCGAGCAGAAGAGCUUCAAAAUUGACACGGC---CAGUGACCCGACGGUCAACGAGGAG---GCCUCCUCGUGCAGUUGCUAGCAGUUGGG ((((.(((((.....)))))((.((((((((.....).)))))))))..)))).(((...---..))).((((((.....(((((((.---...)))))))((........)).)))))) ( -42.10) >DroMoj_CAF1 39475 114 + 1 CAAUCGUUCCAAAAUGGAGCUUCAGGCACUCGAACGACAAAGCUUUCAAGUGGACAAAAC---AAACCAGGCAGAGAUCAACGAGGAU---ACCUCCUCGUGCAGCUGCUAGCAGUUGGG .....(((((.....)))))......((((.(((.(......).))).)))).......(---((..(.(((((......(((((((.---...)))))))....))))).)...))).. ( -30.40) >DroAna_CAF1 31688 117 + 1 CAAUCGAUCCAAGAUGGAGUUGCAGGCUCUGGAGCAAAAAAGCUUCCAAGUGGAUUCAGC---CUCCGAUGCUGCUGUUAACGAGGAGGAUGCCUCUUCUUGUGGUUGCUAGCAGUUGGG .....((((((...(((((((....(((....))).....))).))))..))))))....---..(((..(((((((.(((((((((((...))))))).....)))).)))))))))). ( -37.50) >DroPer_CAF1 41316 114 + 1 CAAUCGCUCCAAAAUGGAGCUGCAGGCUCUCGAGCAGAAGAGCUUCAAAAUUGACACGGC---CAGUGACCCGACGGUCAACGAGGAG---GCCUCCUCGUGCAGUUGCUAGCAGUUGGG ((((.(((((.....)))))((.((((((((.....).)))))))))..)))).(((...---..))).((((((.....(((((((.---...)))))))((........)).)))))) ( -42.10) >consensus CAAUCGCUCCAAAAUGGAGCUGCAGGCACUCGAGCAAAAGAGCUUCCAAAUGGACACGGC___CAGCGACGCGGCGAUCAACGAGGAG___GCCUCCUCGUGCAGCUGCUAGCAGUUGGG ((((.(((((.....)))))(((.(((....((((......))))...........................(((.....((((((((.....))))))))...)))))).))))))).. (-25.35 = -25.88 + 0.53)

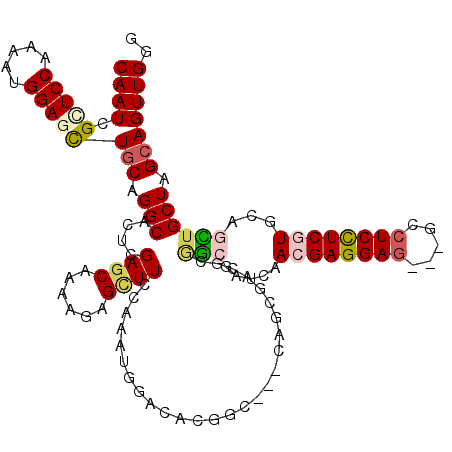
| Location | 674,910 – 675,017 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 110 |
| Reading direction | forward |
| Mean pairwise identity | 77.72 |
| Mean single sequence MFE | -42.07 |
| Consensus MFE | -32.94 |
| Energy contribution | -32.72 |
| Covariance contribution | -0.22 |
| Combinations/Pair | 1.21 |
| Mean z-score | -3.78 |
| Structure conservation index | 0.78 |
| SVM decision value | 1.76 |
| SVM RNA-class probability | 0.975726 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 674910 107 + 23771897 ACGAGGAGGACGCCUCCUCCUGCGGCUGCUAGCAGUUGGGGAACAUUGAGUUCGGCUAGGAACCC---UUAUUCCCGCCAGGCUCAAUGCUCACGCAGAGAUGCAUUUUU ..(((((((...)))))))(((((((((....))))....((.((((((((..(((..((((...---...)))).)))..)))))))).)).)))))............ ( -45.20) >DroVir_CAF1 37382 105 + 1 ACGAGGAG---GCCUCCUCGUGCAGCUGCUAGCAGUUGGGAAACAUUGAGUUUGGCUAAUUCGUUGGCG-CUCUCGGCUGAACUCAAUGU-AACGCAGCAAUGCAUUUGU (((((((.---...))))))).((((((....))))))....((((((((((..(((....((....))-.....)))..))))))))))-...(((....)))...... ( -43.70) >DroPse_CAF1 36971 101 + 1 ACGAGGAG---GCCUCCUCGUGCAGUUGCUAGCAGUUGGGGAACAUUGAGUUUGGCUAAAGACAU---CCAU-CAAGCCAGACUCAAUGC--GAGCAGAAAUGCAUUUUU (((((((.---...)))))))(((.(((((.((.(((....)))(((((((((((((...((...---...)-).)))))))))))))))--.)))))...)))...... ( -41.40) >DroGri_CAF1 44752 97 + 1 ACGAGGAA---GCCUCCGCUUGCAGCUGCUAGCAAUUGGGGAACAUUGAGUUUGGCUAAUA---------CUAAAUGCCAACCUCAAUGG-AAUGCAGAAAUGCAUUUAU ........---..(((((.((((........)))).)))))..(((((((.(((((.....---------......))))).)))))))(-((((((....))))))).. ( -35.80) >DroSec_CAF1 30536 107 + 1 ACGAGGAGGACGCCUCCUCCUGCGGCUGCUAGCAGUUGGGGAACAUUGAGUUCGGCUAGGAAGCG---UUAUCCCCGCCAGGCUCAAUGCUCAUGCAGAGAUGCAUUUUU ..(((((((...)))))))((.((((((....)))))).))..((((((((..(((..(((....---...)))..)))..))))))))...(((((....))))).... ( -45.10) >DroPer_CAF1 41393 100 + 1 ACGAGGAG---GCCUCCUCGUGCAGUUGCUAGCAGUUGGGGAACAUUGAGUUUGGCUAAAGACAU---CCCA-C-AGCCAGACUCAAUGC--GAGCAGAAAUGCAUUUUU (((((((.---...)))))))(((.(((((.((.(((....)))(((((((((((((........---....-.-)))))))))))))))--.)))))...)))...... ( -41.22) >consensus ACGAGGAG___GCCUCCUCGUGCAGCUGCUAGCAGUUGGGGAACAUUGAGUUUGGCUAAAAACAU___C_AU_CCAGCCAGACUCAAUGC__AAGCAGAAAUGCAUUUUU ..((((((.....)))))).((((.((((...(....).....(((((((((((((....................))))))))))))).....))))...))))..... (-32.94 = -32.72 + -0.22)

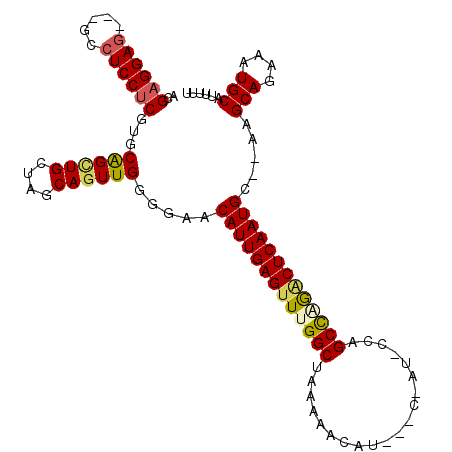
| Location | 674,910 – 675,017 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 110 |
| Reading direction | reverse |
| Mean pairwise identity | 77.72 |
| Mean single sequence MFE | -38.72 |
| Consensus MFE | -25.11 |
| Energy contribution | -26.03 |
| Covariance contribution | 0.92 |
| Combinations/Pair | 1.12 |
| Mean z-score | -3.51 |
| Structure conservation index | 0.65 |
| SVM decision value | 2.31 |
| SVM RNA-class probability | 0.992084 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 674910 107 - 23771897 AAAAAUGCAUCUCUGCGUGAGCAUUGAGCCUGGCGGGAAUAA---GGGUUCCUAGCCGAACUCAAUGUUCCCCAACUGCUAGCAGCCGCAGGAGGAGGCGUCCUCCUCGU ............(((((.((((((((((..((((((((((..---..)))))).))))..)))))))))).....(((....))).)))))(((((((...))))))).. ( -47.30) >DroVir_CAF1 37382 105 - 1 ACAAAUGCAUUGCUGCGUU-ACAUUGAGUUCAGCCGAGAG-CGCCAACGAAUUAGCCAAACUCAAUGUUUCCCAACUGCUAGCAGCUGCACGAGGAGGC---CUCCUCGU ......(((((((((((((-((((((((((..((..(...-((....))..)..))..)))))))))).....))).)).))))).)))(((((((...---.))))))) ( -37.50) >DroPse_CAF1 36971 101 - 1 AAAAAUGCAUUUCUGCUC--GCAUUGAGUCUGGCUUG-AUGG---AUGUCUUUAGCCAAACUCAAUGUUCCCCAACUGCUAGCAACUGCACGAGGAGGC---CUCCUCGU ......(((....((((.--(((((((((.(((((.(-((..---..)))...))))).)))))))(((....))).)).))))..)))(((((((...---.))))))) ( -36.20) >DroGri_CAF1 44752 97 - 1 AUAAAUGCAUUUCUGCAUU-CCAUUGAGGUUGGCAUUUAG---------UAUUAGCCAAACUCAAUGUUCCCCAAUUGCUAGCAGCUGCAAGCGGAGGC---UUCCUCGU ...((((((....))))))-.(((((((.(((((......---------.....))))).))))))).........((((.((....)).))))((((.---..)))).. ( -29.00) >DroSec_CAF1 30536 107 - 1 AAAAAUGCAUCUCUGCAUGAGCAUUGAGCCUGGCGGGGAUAA---CGCUUCCUAGCCGAACUCAAUGUUCCCCAACUGCUAGCAGCCGCAGGAGGAGGCGUCCUCCUCGU ....(((((....)))))((((((((((..((((.((((...---....)))).))))..)))))))))).....((((........))))(((((((...))))))).. ( -46.00) >DroPer_CAF1 41393 100 - 1 AAAAAUGCAUUUCUGCUC--GCAUUGAGUCUGGCU-G-UGGG---AUGUCUUUAGCCAAACUCAAUGUUCCCCAACUGCUAGCAACUGCACGAGGAGGC---CUCCUCGU ......(((....((((.--(((((((((.(((((-(-.((.---....)).)))))).)))))))(((....))).)).))))..)))(((((((...---.))))))) ( -36.30) >consensus AAAAAUGCAUUUCUGCAU__GCAUUGAGUCUGGCGGG_AG_A___AGGUUAUUAGCCAAACUCAAUGUUCCCCAACUGCUAGCAGCUGCACGAGGAGGC___CUCCUCGU .....((((...((((....((((((((..((((....................))))..)))))))).............)))).)))).((((((.....)))))).. (-25.11 = -26.03 + 0.92)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:37:23 2006