
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 5,985,206 – 5,985,317 |
| Length | 111 |
| Max. P | 0.804652 |

| Location | 5,985,206 – 5,985,317 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 112 |
| Reading direction | forward |
| Mean pairwise identity | 82.51 |
| Mean single sequence MFE | -34.53 |
| Consensus MFE | -25.78 |
| Energy contribution | -27.12 |
| Covariance contribution | 1.34 |
| Combinations/Pair | 1.26 |
| Mean z-score | -2.13 |
| Structure conservation index | 0.75 |
| SVM decision value | 0.63 |
| SVM RNA-class probability | 0.804652 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
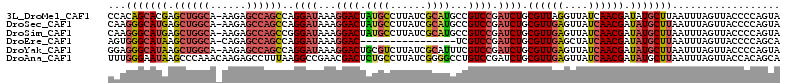
>3L_DroMel_CAF1 5985206 111 + 23771897 CCACAGCACGAGCUGGCA-AAGAGCCAGCCAGGAUAAAGGACUAUGCCUUAUCGCAUGCCGUCCGAUCUGCGUUAGGUUAUCAACGAUAUGCUUAAUUUAGUUACCCCAGUA .....((..(.((((((.-....))))))).((((...(((((((((......)))))..)))).))))))(((((((((((...)))).)))))))............... ( -31.30) >DroSec_CAF1 40679 111 + 1 CAAGGGCAUGAGCUGGCA-AAGAGCCAGCCAGGAUAAAGGACUAUGCCUUAUCGCAUGCCGUCCGAUCUGCGUUGAGUUAUCAACGAUAUGCUUAAUUUAGUUACCCCAGUA ...(((((((.((((((.-....))))))..((((...(((((((((......)))))..)))).)))).((((((....)))))).))))))).................. ( -36.90) >DroSim_CAF1 42081 111 + 1 CAAGGGCAUGAGCUGGCA-AAGAGCCAGCCGGGAUAAAGGACUAUGCCUUAUCGCAUGCCGUCCGAUCUGCGUUGAGUUAUCAACGAUAUGCUUAAUUUAGUUACCCCAGUA ...(((((((.((((((.-....))))))..((((...(((((((((......)))))..)))).)))).((((((....)))))).))))))).................. ( -37.00) >DroEre_CAF1 40708 95 + 1 AGUGGGCAUAAGCUGGCA-CAGAGCCAGCCAGGAUAAAGGAC----------------UCGUCCGAUCUGCGUUGAGCUAUCAACGAUAUGCUUAAUUUAGUUACCCCAGCA ..((((((((.((((((.-....))))))..((((...(((.----------------...))).)))).((((((....)))))).))))))))................. ( -32.10) >DroYak_CAF1 42690 111 + 1 GGAGGGCAUAAGCUGGCA-AAGAGCCAGCCAGGAUAAAGGACUGCGUCUUAUCGCAUUUCGUCCGAUCUGCGUUGAGUUAUCAACGAUAUGCUUAAUUUAGUUACCCCAGUA ((.(((((((.((((((.-....))))))..((((...((((((((......))))....)))).)))).((((((....)))))).))))).((((...)))))))).... ( -38.00) >DroAna_CAF1 40002 112 + 1 UUUGGGAAUAAGCCCAAACAAGAGCCUUUAAGGCCGAACGACUCUGCCUUAUCGGGGCCUGUCCGAUCUGCGUUGAGUUAUCAACGAUAUGCUUAAUUUAGUUACCACAGCA ((((((......)))))).....(((((((((((.((.....)).))))))..)))))((((.....(((.(((((((((((...)))).))))))).))).....)))).. ( -31.90) >consensus CAAGGGCAUAAGCUGGCA_AAGAGCCAGCCAGGAUAAAGGACUAUGCCUUAUCGCAUGCCGUCCGAUCUGCGUUGAGUUAUCAACGAUAUGCUUAAUUUAGUUACCCCAGUA ...(((((((.((((((......))))))..((((...((((.((((......))))...)))).)))).((((((....)))))).))))))).................. (-25.78 = -27.12 + 1.34)


| Location | 5,985,206 – 5,985,317 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 112 |
| Reading direction | reverse |
| Mean pairwise identity | 82.51 |
| Mean single sequence MFE | -33.18 |
| Consensus MFE | -22.75 |
| Energy contribution | -24.62 |
| Covariance contribution | 1.86 |
| Combinations/Pair | 1.21 |
| Mean z-score | -1.65 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.10 |
| SVM RNA-class probability | 0.581515 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 5985206 111 - 23771897 UACUGGGGUAACUAAAUUAAGCAUAUCGUUGAUAACCUAACGCAGAUCGGACGGCAUGCGAUAAGGCAUAGUCCUUUAUCCUGGCUGGCUCUU-UGCCAGCUCGUGCUGUGG ..(((.((((..........((((..(((((((...(....)...))).))))..))))(((((((.......)))))))..(((((((....-.)))))))..)))).))) ( -33.00) >DroSec_CAF1 40679 111 - 1 UACUGGGGUAACUAAAUUAAGCAUAUCGUUGAUAACUCAACGCAGAUCGGACGGCAUGCGAUAAGGCAUAGUCCUUUAUCCUGGCUGGCUCUU-UGCCAGCUCAUGCCCUUG ....((((((................((((((....))))))......((((...((((......)))).))))........(((((((....-.)))))))..)))))).. ( -35.30) >DroSim_CAF1 42081 111 - 1 UACUGGGGUAACUAAAUUAAGCAUAUCGUUGAUAACUCAACGCAGAUCGGACGGCAUGCGAUAAGGCAUAGUCCUUUAUCCCGGCUGGCUCUU-UGCCAGCUCAUGCCCUUG ....(((((((...............((((((....))))))......((((...((((......)))).)))).)))))))(((((((....-.))))))).......... ( -36.80) >DroEre_CAF1 40708 95 - 1 UGCUGGGGUAACUAAAUUAAGCAUAUCGUUGAUAGCUCAACGCAGAUCGGACGA----------------GUCCUUUAUCCUGGCUGGCUCUG-UGCCAGCUUAUGCCCACU .....(((((................((((((....))))))......(((...----------------.)))........(((((((....-.)))))))..)))))... ( -28.30) >DroYak_CAF1 42690 111 - 1 UACUGGGGUAACUAAAUUAAGCAUAUCGUUGAUAACUCAACGCAGAUCGGACGAAAUGCGAUAAGACGCAGUCCUUUAUCCUGGCUGGCUCUU-UGCCAGCUUAUGCCCUCC ....((((((................((((((....))))))......((((....((((......))))))))........(((((((....-.)))))))..)))))).. ( -34.30) >DroAna_CAF1 40002 112 - 1 UGCUGUGGUAACUAAAUUAAGCAUAUCGUUGAUAACUCAACGCAGAUCGGACAGGCCCCGAUAAGGCAGAGUCGUUCGGCCUUAAAGGCUCUUGUUUGGGCUUAUUCCCAAA ...((.((.........(((((....((((((....))))))....(((((((((..((..((((((.(((...))).))))))..))..))))))))))))))..)))).. ( -31.40) >consensus UACUGGGGUAACUAAAUUAAGCAUAUCGUUGAUAACUCAACGCAGAUCGGACGGCAUGCGAUAAGGCAUAGUCCUUUAUCCUGGCUGGCUCUU_UGCCAGCUCAUGCCCUCG ....(((((((...............((((((....))))))......((((...((((......)))).)))).)))))))(((((((......))))))).......... (-22.75 = -24.62 + 1.86)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:26:23 2006