
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 5,905,336 – 5,905,456 |
| Length | 120 |
| Max. P | 0.832543 |

| Location | 5,905,336 – 5,905,456 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 79.07 |
| Mean single sequence MFE | -42.93 |
| Consensus MFE | -24.94 |
| Energy contribution | -24.25 |
| Covariance contribution | -0.69 |
| Combinations/Pair | 1.39 |
| Mean z-score | -1.62 |
| Structure conservation index | 0.58 |
| SVM decision value | 0.04 |
| SVM RNA-class probability | 0.553530 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
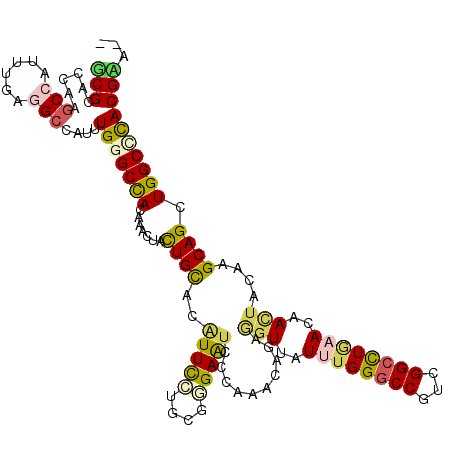
>3L_DroMel_CAF1 5905336 120 + 23771897 GUCUCGUGGGCCAGCUGCUUGAAGUUGUUAAGCCCUGUGGCCCAGAUGACCUGUGUCUGUGACUCGCGCAUGAAUGUGCAGUAGUUGGUGGCCCAGAUGGCCUCAAAAGGUUUGGCCCGC .....(((((((((((..((((.((..(......(((.(((((((((.......)))))..(((.(((((....)))))))).......))))))))..)).))))..)).))))))))) ( -44.70) >DroVir_CAF1 42279 118 + 1 --UGCGUGCGCCAGCUGCUUAUAAUUGUUUAGGCCAACGGCCCAAAUGACCUCAGUCUGCGACUCCCGCAGGAAUGUGCAGUAGUUGGUAGCCCAAAUUGCCUCAAAAGGCUUGGCGCGC --.((((((((((((((((((((.(..(((.((((...)))).)))..)......((((((.....))))))..)))).))))))))))..........((((....))))...)))))) ( -46.60) >DroPse_CAF1 32557 119 + 1 G-UUCGUAAGCCAGCUGCUUGAAGUUGUUCAAGCCGACGGCCCAAAUAACUUCUGUUUGGGAUUCCCGAAGGAACGUACAGUAGUUUGUAGCCCAUAUGGCCUCGAAUGGCUUGGUUCGA .-.(((((((((.((((((((((....)))))))..(((.((((((((.....)))))))).((((....)))))))..))).(((((..(((.....)))..)))))))))))...))) ( -38.20) >DroGri_CAF1 35087 118 + 1 --GACGUGGGCCAGCUGCUUGUAGUUGUUCAGGCCCACGGCCCAAAUAAUGCCAGUCUGCGAUUCGCGCACAAAAGUGCAAUAGUUUGUGGCCCAAAUCGCUUCAAAUGGCUUGGUGCGC --..(((((((((((((....))))).....))))))))((((((.....((((....((((((.((.((((((..........))))))))...))))))......)))))))).)).. ( -43.90) >DroMoj_CAF1 37763 118 + 1 --UGCGUGCGCCAGCUGCUUGUAGUUGUUCAAGCCCACGGCCCAGAUCACCCCCGUUUGGGAUUCACGCAAAAAUGUGCAAUAAUUUGUGGCCCAAAUGGCUUCAAAGGGCUUGGCGCGC --.((((((..((((((....))))))..(((((((....(((((((.......)))))))......(((......))).....((((.((((.....)))).))))))))))))))))) ( -46.00) >DroPer_CAF1 30214 119 + 1 G-UUCGUAAGCCAGCUGCUUGAAGUUGUUCAAGCCGACGGCCCAAAUAACUUCUGUUUGGGAUUCCCGAAGGAACGUACAGUAGUUUGUAGCCCAUAUGGCCUCGAAUGGCUUGGUUCGA .-.(((((((((.((((((((((....)))))))..(((.((((((((.....)))))))).((((....)))))))..))).(((((..(((.....)))..)))))))))))...))) ( -38.20) >consensus __UUCGUGAGCCAGCUGCUUGAAGUUGUUCAAGCCCACGGCCCAAAUAACCUCUGUCUGGGAUUCCCGCAGGAAUGUGCAGUAGUUUGUAGCCCAAAUGGCCUCAAAUGGCUUGGCGCGC .....(((((((((((((.....((((((...(((...)))...)))))).........(((((...(((......)))...)))))))))).......(((......))).)))))))) (-24.94 = -24.25 + -0.69)


| Location | 5,905,336 – 5,905,456 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.07 |
| Mean single sequence MFE | -41.84 |
| Consensus MFE | -22.96 |
| Energy contribution | -22.63 |
| Covariance contribution | -0.33 |
| Combinations/Pair | 1.45 |
| Mean z-score | -2.18 |
| Structure conservation index | 0.55 |
| SVM decision value | 0.72 |
| SVM RNA-class probability | 0.832543 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 5905336 120 - 23771897 GCGGGCCAAACCUUUUGAGGCCAUCUGGGCCACCAACUACUGCACAUUCAUGCGCGAGUCACAGACACAGGUCAUCUGGGCCACAGGGCUUAACAACUUCAAGCAGCUGGCCCACGAGAC (.((((((..........((((.....))))........((((........((....))....(((....)))...((((((....))))))..........)))).)))))).)..... ( -36.60) >DroVir_CAF1 42279 118 - 1 GCGCGCCAAGCCUUUUGAGGCAAUUUGGGCUACCAACUACUGCACAUUCCUGCGGGAGUCGCAGACUGAGGUCAUUUGGGCCGUUGGCCUAAACAAUUAUAAGCAGCUGGCGCACGCA-- ((((((((.((((....))))..(((((((((.(.....((((..(((((....))))).)))).....((((.....))))).)))))))))..............))))))..)).-- ( -44.70) >DroPse_CAF1 32557 119 - 1 UCGAACCAAGCCAUUCGAGGCCAUAUGGGCUACAAACUACUGUACGUUCCUUCGGGAAUCCCAAACAGAAGUUAUUUGGGCCGUCGGCUUGAACAACUUCAAGCAGCUGGCUUACGAA-C (((....((((((..(((((((...((((.((((......)))).(((((....)))))))))..((((.....)))))))).)))(((((((....)))))))...)))))).))).-. ( -41.80) >DroGri_CAF1 35087 118 - 1 GCGCACCAAGCCAUUUGAAGCGAUUUGGGCCACAAACUAUUGCACUUUUGUGCGCGAAUCGCAGACUGGCAUUAUUUGGGCCGUGGGCCUGAACAACUACAAGCAGCUGGCCCACGUC-- ..((.((((((((.((...((((((((.((.(((((..........))))))).)))))))).)).)))).....))))))((((((((...................))))))))..-- ( -43.71) >DroMoj_CAF1 37763 118 - 1 GCGCGCCAAGCCCUUUGAAGCCAUUUGGGCCACAAAUUAUUGCACAUUUUUGCGUGAAUCCCAAACGGGGGUGAUCUGGGCCGUGGGCUUGAACAACUACAAGCAGCUGGCGCACGCA-- ((((((((.((.........((((...((((.((.(((((((((......)))......(((....))))))))).))))))))))(((((........))))).))))))))..)).-- ( -42.40) >DroPer_CAF1 30214 119 - 1 UCGAACCAAGCCAUUCGAGGCCAUAUGGGCUACAAACUACUGUACGUUCCUUCGGGAAUCCCAAACAGAAGUUAUUUGGGCCGUCGGCUUGAACAACUUCAAGCAGCUGGCUUACGAA-C (((....((((((..(((((((...((((.((((......)))).(((((....)))))))))..((((.....)))))))).)))(((((((....)))))))...)))))).))).-. ( -41.80) >consensus GCGCACCAAGCCAUUUGAGGCCAUUUGGGCCACAAACUACUGCACAUUCCUGCGGGAAUCCCAAACAGAGGUUAUUUGGGCCGUCGGCCUGAACAACUACAAGCAGCUGGCCCACGAA__ (((......(((......)))....(((((((.......((((..(((((....)))))..........(((..((((((((...))))))))..)))....)))).))))))))))... (-22.96 = -22.63 + -0.33)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:25:58 2006