
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
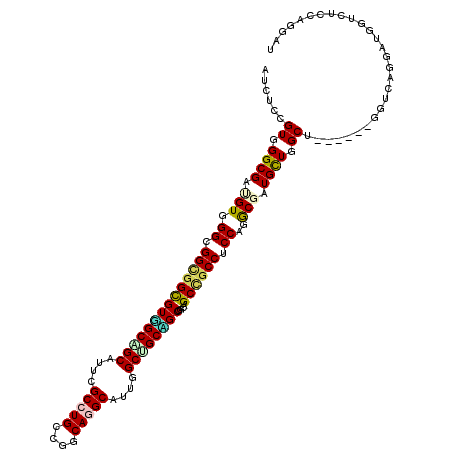
| Location | 5,620,989 – 5,621,142 |
| Length | 153 |
| Max. P | 0.819298 |

| Location | 5,620,989 – 5,621,102 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 81.97 |
| Mean single sequence MFE | -56.38 |
| Consensus MFE | -40.44 |
| Energy contribution | -40.30 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.37 |
| Mean z-score | -2.15 |
| Structure conservation index | 0.72 |
| SVM decision value | 0.68 |
| SVM RNA-class probability | 0.819298 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
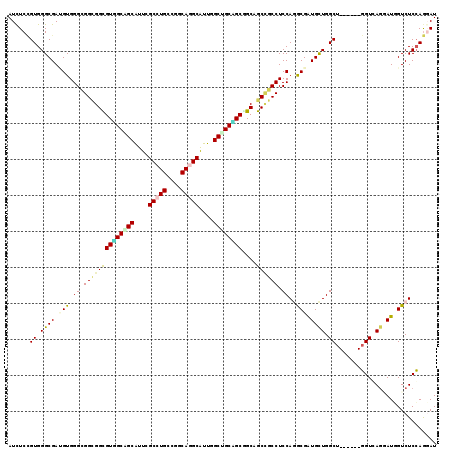
>3L_DroMel_CAF1 5620989 113 - 23771897 AUCUCCGUGGGCGAUGUGGGCGGUGGUGUGGCGGCAUUGGCCUGUCGGCAGGCAUUUGCCGCAGCAGCUGCCGCCUCCAAGCGAUGUUGGCU------GGUGAGGAUAGUCUCCAGGAU ...(((.(((((..((..(((((..((((.((((((...(((((....)))))...)))))).))).)..)))))..)).))......((((------(.......))))).)))))). ( -56.60) >DroVir_CAF1 8287 113 - 1 AUCUCCGUGGGCGAGGUGGGCGGCGGCGUUGCGGCGUUCGCCUGCCUGCAAGCAUUAGCUGCGGCUGCAGCGGCCUCCAGGCGAUGCUGGCU------GGUCAGUAUGGUCUCCAGUAU ......(..(((.(((((((((.((......)).))))))))))))..).((((((.(((((....))))).(((....))))))))).(((------((..((......))))))).. ( -52.90) >DroGri_CAF1 2438 119 - 1 AUCUCUGUGGGCGAUGUGGGUGGUGGUGUUGCAGCAUUUGCCUGCCUACAGGCAUUUGCUGCAGCGGCAGCUGCCUCCAAACGAUGCUGGCUGUGAGUGGUGAGUAUCGUCUCCAAGAU ((((.((..(((..((..((..(((.((((((((((..((((((....))))))..)))))))))).)).)..))..))...((((((.(((......))).)))))))))..)))))) ( -59.80) >DroMoj_CAF1 2351 113 - 1 AUCUCCGUGGGCGAUGUGGGCGGCGGCGUGGCUGCAUUCGCCUGCCGGCAGGCGUUCGCUGCCGCGGCAGCCGCCUCCAGGCGAUGCUGGCU------GGUCAGAAUGGUCUCCAGUAU ..........((..((..(((((((.((((((.((...((((((....))))))...)).)))))).).))))))..)).)).(((((((..------(..(.....)..).))))))) ( -59.30) >DroAna_CAF1 2841 113 - 1 AUUUCCGUGGGCGAGGUGGGCGGGGGCGUGGCAGCGUUGGCCUGACGGCAAGCAUUGGCUGCUGCCGCAGCUGCCUCCAGACGGUGCUGGCU------GGUGAGGAUGGUCUCCAGGAU ...(((((.((((..((.((.((.(((((((((((....(((....))).(((....))))))))))).))).)).))..))..)))).))(------((.((......)).)))))). ( -51.00) >DroPer_CAF1 2597 113 - 1 AUCUCAGUGGGCGACGUGGGGGGCGGUGUGGCAGCGUUCGCCUGGCGGCAGGCAUUGGCCGCUGCGGCAGCCGCCUCCAGGCGGUGCUGGCU------CGUCAAGAUCGUCUCGAGGAU .(((((((.((((.(((.(((((((((((.((((((..((((((....)))))...)..)))))).))).))))))))..))).)))).)))------......((.....)))))).. ( -58.70) >consensus AUCUCCGUGGGCGAUGUGGGCGGCGGCGUGGCAGCAUUCGCCUGCCGGCAGGCAUUGGCUGCAGCGGCAGCCGCCUCCAGGCGAUGCUGGCU______GGUCAGGAUGGUCUCCAGGAU ......((.((((.(((.((.((((((((((((((....(((((....)))))....))))))))....)))))).))..))).)))).))............................ (-40.44 = -40.30 + -0.14)

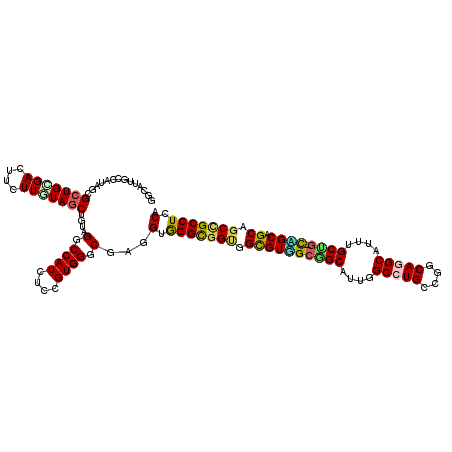
| Location | 5,621,023 – 5,621,142 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 84.13 |
| Mean single sequence MFE | -57.35 |
| Consensus MFE | -39.72 |
| Energy contribution | -39.45 |
| Covariance contribution | -0.27 |
| Combinations/Pair | 1.37 |
| Mean z-score | -2.15 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.05 |
| SVM RNA-class probability | 0.560649 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
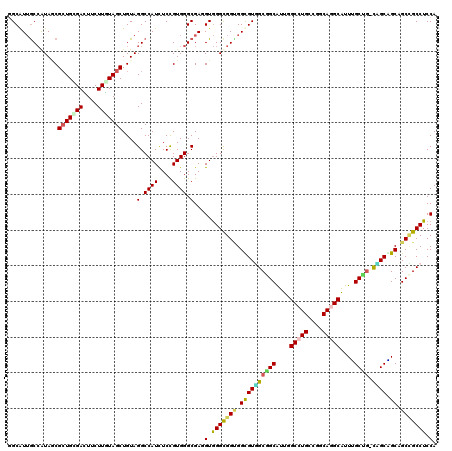
>3L_DroMel_CAF1 5621023 119 - 23771897 GGCAUUACCGUACCGCUGGGAUUUCUUGUAGCUGUAGGCCAUCUCCGUGGGCGAUGUGGGCGGUGGUGUGGCGGCAUUGGCCUGUCGGCAGGCAUUUGCCG-CAGCAGCUGCCGCCUCCA ........(((.((((.(((((..((((......))))..))))).))))))).((..(((((..((((.((((((...(((((....)))))...)))))-).))).)..)))))..)) ( -58.10) >DroVir_CAF1 8321 119 - 1 GGCAUUGCCAUAGCGCUGCGACUUCUUGUAGCUGUAGGCCAUCUCCGUGGGCGAGGUGGGCGGCGGCGUUGCGGCGUUCGCCUGCCUGCAAGCAUUAGCUG-CGGCUGCAGCGGCCUCCA (((...))).....(((((((....))))))).(.(((((......(..(((.(((((((((.((......)).))))))))))))..)........((((-(....)))))))))).). ( -57.10) >DroGri_CAF1 2478 119 - 1 UGCAUUGCCAUAGCGCUGUGAUUUCUUAUAGCUGUAGGCCAUCUCUGUGGGCGAUGUGGGUGGUGGUGUUGCAGCAUUUGCCUGCCUACAGGCAUUUGCUG-CAGCGGCAGCUGCCUCCA .((((((((...(((((((((....)))))))...(((.....))))).))))))))(((..(((.((((((((((..((((((....))))))..)))))-))))).)).)..)))... ( -57.60) >DroSec_CAF1 4950 120 - 1 GGCAUGGCGAUAUCGAUGGGACUUCUUGUAGCUGUAGGCCAUCUCCGUGGGCGAGGUAGGCGGUGGUGUGGCGGCAUUGGCCUGUCGGCAGGCAUUUGCCUUUUGCAGCUGCCGCCUCCA ((...((((........(((....)))(((((((((((((((((((((..((...))..)))).)).)))))((((...(((((....)))))...))))..)))))))))))))).)). ( -52.60) >DroMoj_CAF1 2385 119 - 1 CGCGUUGCCGUAGCGCUGUGACUUCUUGUAGCUGUAGGCCAUCUCCGUGGGCGAUGUGGGCGGCGGCGUGGCUGCAUUCGCCUGCCGGCAGGCGUUCGCUG-CCGCGGCAGCCGCCUCCA (((((((((...((((((..(....)..))))....((......)))).)))))))))(((((((.((((((.((...((((((....))))))...)).)-))))).).)))))).... ( -62.90) >DroAna_CAF1 2875 119 - 1 GGCAUUGCCGUAACGCUGCGACUUCUUGUAGCUGUAGGCCAUUUCCGUGGGCGAGGUGGGCGGGGGCGUGGCAGCGUUGGCCUGACGGCAAGCAUUGGCUG-CUGCCGCAGCUGCCUCCA ((((..((((..(((((((.(((((((((.(((...(.((((....)))).)..)))..))))))).)).)))))))))))(((.(((((.(((.....))-))))))))).)))).... ( -55.80) >consensus GGCAUUGCCAUAGCGCUGCGACUUCUUGUAGCUGUAGGCCAUCUCCGUGGGCGAGGUGGGCGGUGGCGUGGCGGCAUUGGCCUGCCGGCAGGCAUUUGCUG_CAGCAGCAGCCGCCUCCA ..............(((((((....)))))))....(.((((....)))).)...(.(((((((.((((((((((....(((((....)))))....)))).)))).)).))))))).). (-39.72 = -39.45 + -0.27)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:23:05 2006