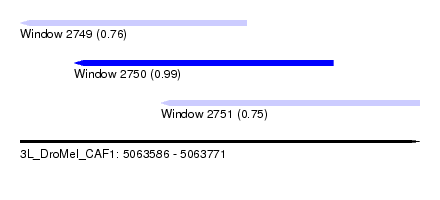
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 5,063,586 – 5,063,771 |
| Length | 185 |
| Max. P | 0.993682 |

| Location | 5,063,586 – 5,063,691 |
|---|---|
| Length | 105 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.64 |
| Mean single sequence MFE | -27.40 |
| Consensus MFE | -14.50 |
| Energy contribution | -14.70 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.08 |
| Structure conservation index | 0.53 |
| SVM decision value | 0.51 |
| SVM RNA-class probability | 0.764944 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 5063586 105 - 23771897 AGGUCCACCUCCGCAACUGUGGUGUGAGCUCAGCUAAUUUCUGGUCAAGUUCAGCUAUUUACCAUCUUAUACCAAGUUAUUUGUUAGCUU---------------GGAGAAGAAAUCAAU .(((....(.((((....)))).)((((((..((((.....))))..)))))).......))).((((...((((((((.....))))))---------------))..))))....... ( -28.40) >DroSec_CAF1 270108 104 - 1 AGCUCCACCUCACCGACUGUGGUGUGAGCUCAGCUAAUUUCUGGUCAAGUUCAGCUAUUUACCGUCUUAUACCAAGUUAUUUGUUAGCUU---------------GG-GAAGAAAUUAAU (((((((((.((.....)).)))).)))))....(((((((((((.((((......)))))))........((((((((.....))))))---------------))-..)))))))).. ( -32.70) >DroSim_CAF1 291411 104 - 1 AGCUCCACCUCACCGACUGUGGUGUGAGCUCAGCUAAUUUCUGGUCAAGUUCAGCUAUUUACCAUCUUAUACCAAGUUAUUUGUUAGCUU---------------GG-GAAGAAAUUAAU (((((((((.((.....)).)))).)))))....(((((((((((.((((......)))))))........((((((((.....))))))---------------))-..)))))))).. ( -32.70) >DroEre_CAF1 276973 93 - 1 AGCUCCACCCC-CCAACUGAGGUGUGAGCUCAGCUAAUUUCUGGUCAAGUUCAGCUAUUUACC---UUAUACUUGGUUAUUUGUCAA-UUGUU----------------------UCACU (((((((((.(-......).)))).))))).(((((((((......))))).))))....(((---........)))..........-.....----------------------..... ( -17.60) >DroYak_CAF1 280745 118 - 1 AGCUCCACCCC-CCAACUGUGGUGUGAGCUCAGCUAAUUUCUGGUCAAGUUCAGCUACUCACCUUCUUAUACCUGGUUAC-CGUCAACUUGUUUACGAUAUUCGUGGAGAUGAAGUCACU ..((((((...-......(((((.((((((..((((.....))))..)))))))))))......((.((.((..((((..-....)))).)).)).)).....))))))........... ( -25.60) >consensus AGCUCCACCUC_CCAACUGUGGUGUGAGCUCAGCUAAUUUCUGGUCAAGUUCAGCUAUUUACCAUCUUAUACCAAGUUAUUUGUUAGCUU_______________GG_GAAGAAAUCAAU (((((((((.(.......).)))).))))).(((((((((......))))).))))................................................................ (-14.50 = -14.70 + 0.20)



| Location | 5,063,611 – 5,063,731 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.24 |
| Mean single sequence MFE | -25.95 |
| Consensus MFE | -23.74 |
| Energy contribution | -24.18 |
| Covariance contribution | 0.44 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.81 |
| Structure conservation index | 0.91 |
| SVM decision value | 2.42 |
| SVM RNA-class probability | 0.993682 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 5063611 120 - 23771897 UUUCUCUGCAUGGAAUAUAAUUAAUUUGGUAUUUACCCACAGGUCCACCUCCGCAACUGUGGUGUGAGCUCAGCUAAUUUCUGGUCAAGUUCAGCUAUUUACCAUCUUAUACCAAGUUAU .((((......))))......(((((((((((..((((((((..............)))))).))(((((..((((.....))))..)))))................))))))))))). ( -28.24) >DroSec_CAF1 270132 120 - 1 UUUCUCUGCAUGGAAUAUAAUUAAUUUGGUAUUUACCCACAGCUCCACCUCACCGACUGUGGUGUGAGCUCAGCUAAUUUCUGGUCAAGUUCAGCUAUUUACCGUCUUAUACCAAGUUAU .((((......))))......(((((((((((..((....(((((((((.((.....)).)))).))))).(((((((((......))))).)))).......))...))))))))))). ( -29.50) >DroSim_CAF1 291435 120 - 1 UUUCUCUGCAUGGAAUAUAAUUAAUUUGGUAUUUACCCACAGCUCCACCUCACCGACUGUGGUGUGAGCUCAGCUAAUUUCUGGUCAAGUUCAGCUAUUUACCAUCUUAUACCAAGUUAU .((((......))))......(((((((((((........(((((((((.((.....)).)))).))))).(((((((((......))))).))))............))))))))))). ( -29.00) >DroEre_CAF1 276990 116 - 1 UUUCUCUGCAUGGAAUAUAAUUAAUUUGGUAUUUACCCACAGCUCCACCCC-CCAACUGAGGUGUGAGCUCAGCUAAUUUCUGGUCAAGUUCAGCUAUUUACC---UUAUACUUGGUUAU .((((......)))).((((((((...((((.........(((((((((.(-......).)))).))))).(((((((((......))))).))))...))))---......)))))))) ( -21.90) >DroYak_CAF1 280784 119 - 1 UUUCUCUGCAUGGAAUAUAAUUAAUUUCGUAUUUACCCACAGCUCCACCCC-CCAACUGUGGUGUGAGCUCAGCUAAUUUCUGGUCAAGUUCAGCUACUCACCUUCUUAUACCUGGUUAC .......(((((((((.......)))))))....((((((((.........-....)))))).))(((((..((((.....))))..))))).)).....(((...........)))... ( -21.12) >consensus UUUCUCUGCAUGGAAUAUAAUUAAUUUGGUAUUUACCCACAGCUCCACCUC_CCAACUGUGGUGUGAGCUCAGCUAAUUUCUGGUCAAGUUCAGCUAUUUACCAUCUUAUACCAAGUUAU .((((......))))......(((((((((((..((((((((..............)))))).))(((((..((((.....))))..)))))................))))))))))). (-23.74 = -24.18 + 0.44)



| Location | 5,063,651 – 5,063,771 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.64 |
| Mean single sequence MFE | -33.74 |
| Consensus MFE | -28.24 |
| Energy contribution | -28.84 |
| Covariance contribution | 0.60 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.55 |
| Structure conservation index | 0.84 |
| SVM decision value | 0.48 |
| SVM RNA-class probability | 0.754652 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 5063651 120 - 23771897 GUAGUUGGGUUUCGGUUGGGCCAACGCUACGCUUGAUUAAUUUCUCUGCAUGGAAUAUAAUUAAUUUGGUAUUUACCCACAGGUCCACCUCCGCAACUGUGGUGUGAGCUCAGCUAAUUU .(((((((((((.(((.(((((..........((((((((((((.......))))).)))))))...(((....)))....)))))))).((((....))))...))))))))))).... ( -35.50) >DroSec_CAF1 270172 119 - 1 GUAGU-GGGUUUUGGUUGGGCCAACGCUACGCUUGAUUAAUUUCUCUGCAUGGAAUAUAAUUAAUUUGGUAUUUACCCACAGCUCCACCUCACCGACUGUGGUGUGAGCUCAGCUAAUUU ...((-((((.((((.....)))).((((...((((((((((((.......))))).)))))))..))))....))))))(((((((((.((.....)).)))).))))).......... ( -34.20) >DroSim_CAF1 291475 119 - 1 GUAGU-GGGUUUUGGUUGGGCCAACGCUACGCUUGAUUAAUUUCUCUGCAUGGAAUAUAAUUAAUUUGGUAUUUACCCACAGCUCCACCUCACCGACUGUGGUGUGAGCUCAGCUAAUUU ...((-((((.((((.....)))).((((...((((((((((((.......))))).)))))))..))))....))))))(((((((((.((.....)).)))).))))).......... ( -34.20) >DroEre_CAF1 277027 118 - 1 GUAGU-GGGUUUUGGUUGGGCCAACGCUACGCUUGAUUAAUUUCUCUGCAUGGAAUAUAAUUAAUUUGGUAUUUACCCACAGCUCCACCCC-CCAACUGAGGUGUGAGCUCAGCUAAUUU ...((-((((.((((.....)))).((((...((((((((((((.......))))).)))))))..))))....))))))(((((((((.(-......).)))).))))).......... ( -32.30) >DroYak_CAF1 280824 118 - 1 GUAGC-GGGUUUUGGUUGGGCCGACGCUACGCUUGAUUAAUUUCUCUGCAUGGAAUAUAAUUAAUUUCGUAUUUACCCACAGCUCCACCCC-CCAACUGUGGUGUGAGCUCAGCUAAUUU .((((-((((.((((.....))))...((((.((((((((((((.......))))).)))))))...))))...))))..(((((((((.(-......).)))).)))))..)))).... ( -32.50) >consensus GUAGU_GGGUUUUGGUUGGGCCAACGCUACGCUUGAUUAAUUUCUCUGCAUGGAAUAUAAUUAAUUUGGUAUUUACCCACAGCUCCACCUC_CCAACUGUGGUGUGAGCUCAGCUAAUUU ...........((((((((((.........(((.((((((((..(((....)))....)))))))).)))..((((((((((..............)))))).))))))))))))))... (-28.24 = -28.84 + 0.60)

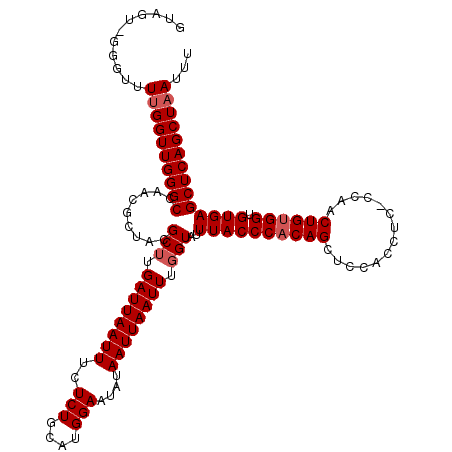
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:16:04 2006