
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 5,062,037 – 5,062,231 |
| Length | 194 |
| Max. P | 0.997657 |

| Location | 5,062,037 – 5,062,151 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 116 |
| Reading direction | forward |
| Mean pairwise identity | 97.30 |
| Mean single sequence MFE | -36.98 |
| Consensus MFE | -33.12 |
| Energy contribution | -33.24 |
| Covariance contribution | 0.12 |
| Combinations/Pair | 1.08 |
| Mean z-score | -1.81 |
| Structure conservation index | 0.90 |
| SVM decision value | 1.69 |
| SVM RNA-class probability | 0.972054 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 5062037 114 + 23771897 UUUUGACUCUCGAUCCCUGGCCGCUUGCAGAAAUGCUAAUUAGGCGUAAUUCGCAUAAAAUUCGCAAACAUUUGCUGUCACUGG--AGUCCGGGACUCGGGAAUCGGGAUUCGCUU ....((.(((((((.(((((((....(((....)))......)))..................((((....)))).(((.((((--...))))))).)))).))))))).)).... ( -35.10) >DroSec_CAF1 268500 114 + 1 UUUUGACUCUUGAUCCCCGGCCGCUUUCAGAAAUGCUAAUUAGGCGUAAUUCGCAUAAAAUUCGCAAACAUUUGCUGUCACUGG--AGUCCGGGACUCGGGAAUCGGGAUUCGCUU ((((((.((((((..(((((..((((.(((..((((.(((((....))))).)))).......((((....)))).....))))--))))))))..)))))).))))))....... ( -33.00) >DroSim_CAF1 289808 114 + 1 UUUUGACUCUCGAUCCCCGGCCGCUUCCAGAAAUGCUAAUUAGGCGUAAUUCGCAUAAAAUUCGCAAACAUUUGCUGUCACUGG--AGUCCGGGACUCGGGAAUCGGGAUUCGCUU ((((((.((((((..(((((..(((.((((..((((.(((((....))))).)))).......((((....)))).....))))--))))))))..)))))).))))))....... ( -37.90) >DroEre_CAF1 275341 114 + 1 UUUUGACUCUCGAUCCCCGGCCGCUUGCAGAAAUGCUAAUUAGGCGUAAUUCGCAUAAAAUUCGCAAACAUUUGCUGUCACUGG--AGUCCGGGGCUCGGGAAUCGGGAUACGCUU ((((((.((((((.((((((..(((((((....)))....((((((.....))).........((((....)))).....))))--))))))))).)))))).))))))....... ( -39.40) >DroYak_CAF1 279097 116 + 1 UUUUGACUCUCGAUCCCCGGCCGCUUGCAGAAAUGCUAAUUAGGCGUAAUUCGCAUAAAAUUCGCAAACAUUUGCUGUCACUGGAGAGUCCGGGACUCGGGAAUCGGGAUUCGCUU ....((.(((((((.(((((((....(((....)))......)))..................((((....)))).(((.((((.....))))))).)))).))))))).)).... ( -39.50) >consensus UUUUGACUCUCGAUCCCCGGCCGCUUGCAGAAAUGCUAAUUAGGCGUAAUUCGCAUAAAAUUCGCAAACAUUUGCUGUCACUGG__AGUCCGGGACUCGGGAAUCGGGAUUCGCUU ....((.(((((((.(((((......(((((..(((.((((..(((.....)))....)))).)))....))))).(((.((((.....)))))))))))).))))))).)).... (-33.12 = -33.24 + 0.12)

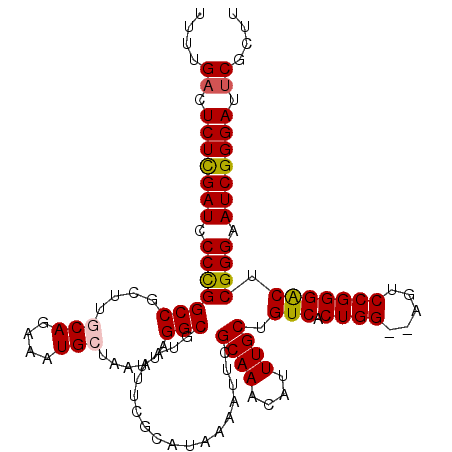

| Location | 5,062,037 – 5,062,151 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 116 |
| Reading direction | reverse |
| Mean pairwise identity | 97.30 |
| Mean single sequence MFE | -34.70 |
| Consensus MFE | -33.04 |
| Energy contribution | -33.48 |
| Covariance contribution | 0.44 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.53 |
| Structure conservation index | 0.95 |
| SVM decision value | 1.51 |
| SVM RNA-class probability | 0.960124 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 5062037 114 - 23771897 AAGCGAAUCCCGAUUCCCGAGUCCCGGACU--CCAGUGACAGCAAAUGUUUGCGAAUUUUAUGCGAAUUACGCCUAAUUAGCAUUUCUGCAAGCGGCCAGGGAUCGAGAGUCAAAA ...........(((((.(((.((((((...--((.((....))....(((((((((....((((.(((((....))))).))))))).)))))))))).))))))).))))).... ( -31.40) >DroSec_CAF1 268500 114 - 1 AAGCGAAUCCCGAUUCCCGAGUCCCGGACU--CCAGUGACAGCAAAUGUUUGCGAAUUUUAUGCGAAUUACGCCUAAUUAGCAUUUCUGAAAGCGGCCGGGGAUCAAGAGUCAAAA ...........(((((..((..(((((...--((.((..(((.(((((((.((((((((.....))))).)))......))))))))))...)))))))))..))..))))).... ( -30.70) >DroSim_CAF1 289808 114 - 1 AAGCGAAUCCCGAUUCCCGAGUCCCGGACU--CCAGUGACAGCAAAUGUUUGCGAAUUUUAUGCGAAUUACGCCUAAUUAGCAUUUCUGGAAGCGGCCGGGGAUCGAGAGUCAAAA ....((.((.(((((((((.(((.(....(--((((.....((((....)))).......((((.(((((....))))).))))..))))).).)))))))))))).)).)).... ( -35.70) >DroEre_CAF1 275341 114 - 1 AAGCGUAUCCCGAUUCCCGAGCCCCGGACU--CCAGUGACAGCAAAUGUUUGCGAAUUUUAUGCGAAUUACGCCUAAUUAGCAUUUCUGCAAGCGGCCGGGGAUCGAGAGUCAAAA ...........(((((.(((.((((((...--((.((....))....(((((((((....((((.(((((....))))).))))))).)))))))))))))).))).))))).... ( -37.70) >DroYak_CAF1 279097 116 - 1 AAGCGAAUCCCGAUUCCCGAGUCCCGGACUCUCCAGUGACAGCAAAUGUUUGCGAAUUUUAUGCGAAUUACGCCUAAUUAGCAUUUCUGCAAGCGGCCGGGGAUCGAGAGUCAAAA ....((.((.(((((((((.((((.((.....)).).))).((....(((((((((....((((.(((((....))))).))))))).)))))).))))))))))).)).)).... ( -38.00) >consensus AAGCGAAUCCCGAUUCCCGAGUCCCGGACU__CCAGUGACAGCAAAUGUUUGCGAAUUUUAUGCGAAUUACGCCUAAUUAGCAUUUCUGCAAGCGGCCGGGGAUCGAGAGUCAAAA ....((.((.(((((((((.(((..((.....)).((..(((.(((((((.((((((((.....))))).)))......))))))))))...)))))))))))))).)).)).... (-33.04 = -33.48 + 0.44)

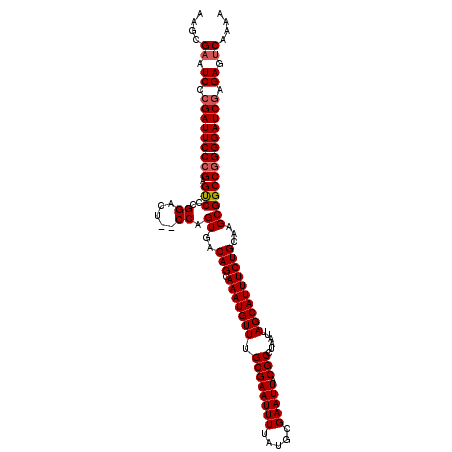

| Location | 5,062,073 – 5,062,191 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.46 |
| Mean single sequence MFE | -42.34 |
| Consensus MFE | -36.82 |
| Energy contribution | -37.10 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.42 |
| Structure conservation index | 0.87 |
| SVM decision value | 2.73 |
| SVM RNA-class probability | 0.996647 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 5062073 118 + 23771897 UAAUUAGGCGUAAUUCGCAUAAAAUUCGCAAACAUUUGCUGUCACUGG--AGUCCGGGACUCGGGAAUCGGGAUUCGCUUGGCGGCAGUCAGCUUUUGAUUUACGAGUUCCAGGGCUCCG .......(((.....))).........((((....)))).......((--(((((((((((((..((((((((...(((.(((....))))))))))))))..)))))))).))))))). ( -46.30) >DroSec_CAF1 268536 118 + 1 UAAUUAGGCGUAAUUCGCAUAAAAUUCGCAAACAUUUGCUGUCACUGG--AGUCCGGGACUCGGGAAUCGGGAUUCGCUUGGCGGCAGUCAGCUUUUGAUUUACGAGUUCCAGGGCUCCG .......(((.....))).........((((....)))).......((--(((((((((((((..((((((((...(((.(((....))))))))))))))..)))))))).))))))). ( -46.30) >DroSim_CAF1 289844 118 + 1 UAAUUAGGCGUAAUUCGCAUAAAAUUCGCAAACAUUUGCUGUCACUGG--AGUCCGGGACUCGGGAAUCGGGAUUCGCUUGGCGGCAGUCAGCUUUUGAUUUACGAGUUCCAGGGCUCCG .......(((.....))).........((((....)))).......((--(((((((((((((..((((((((...(((.(((....))))))))))))))..)))))))).))))))). ( -46.30) >DroEre_CAF1 275377 115 + 1 UAAUUAGGCGUAAUUCGCAUAAAAUUCGCAAACAUUUGCUGUCACUGG--AGUCCGGGGCUCGGGAAUCGGGAUACGCUUGG---CAGUCAGCUUUUGAUUUACGAGUUCCAGAGCUCCG .......(((.....))).........((((....)))).......((--((..(((((((((..((((((((...(((...---.....)))))))))))..)))))))).)..)))). ( -36.10) >DroYak_CAF1 279133 117 + 1 UAAUUAGGCGUAAUUCGCAUAAAAUUCGCAAACAUUUGCUGUCACUGGAGAGUCCGGGACUCGGGAAUCGGGAUUCGCUUGG---CAGUCAGCUUUUGAUUUACGAGUUUCAGGGCUCCG ...(((((((.....))).........((((....)))).....)))).(((((((..(((((..((((((((...(((...---.....)))))))))))..)))))..).)))))).. ( -36.70) >consensus UAAUUAGGCGUAAUUCGCAUAAAAUUCGCAAACAUUUGCUGUCACUGG__AGUCCGGGACUCGGGAAUCGGGAUUCGCUUGGCGGCAGUCAGCUUUUGAUUUACGAGUUCCAGGGCUCCG .......(((.....))).........((((....)))).......((..(((((((((((((..((((((((...(((.(((....))))))))))))))..)))))))).))))))). (-36.82 = -37.10 + 0.28)

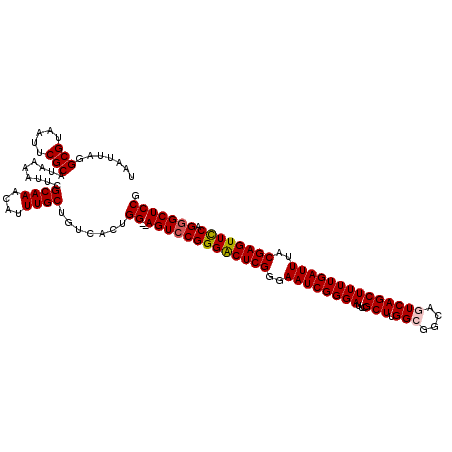

| Location | 5,062,113 – 5,062,231 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 90.99 |
| Mean single sequence MFE | -49.20 |
| Consensus MFE | -40.22 |
| Energy contribution | -40.50 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.88 |
| Structure conservation index | 0.82 |
| SVM decision value | 2.90 |
| SVM RNA-class probability | 0.997657 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 5062113 118 + 23771897 GUCACUGG--AGUCCGGGACUCGGGAAUCGGGAUUCGCUUGGCGGCAGUCAGCUUUUGAUUUACGAGUUCCAGGGCUCCGGGAUGCCAAUCUGGCCAAUCUGGCCUAUCAACGGCUCAUC (((.((((--(((((((((((((..((((((((...(((.(((....))))))))))))))..)))))))).))))))))))))(((.....((((.....)))).......)))..... ( -56.70) >DroSec_CAF1 268576 109 + 1 GUCACUGG--AGUCCGGGACUCGGGAAUCGGGAUUCGCUUGGCGGCAGUCAGCUUUUGAUUUACGAGUUCCAGGGCUCCGGGAUGCCAAU---------CUGGCCUAUCAACGGCUCAUC (((.((((--(((((((((((((..((((((((...(((.(((....))))))))))))))..)))))))).)))))))))((((((...---------..)))..)))...)))..... ( -51.30) >DroSim_CAF1 289884 109 + 1 GUCACUGG--AGUCCGGGACUCGGGAAUCGGGAUUCGCUUGGCGGCAGUCAGCUUUUGAUUUACGAGUUCCAGGGCUCCGGGAUGCCAAU---------CUGGCCUAUCAACGGCUCAUC (((.((((--(((((((((((((..((((((((...(((.(((....))))))))))))))..)))))))).)))))))))((((((...---------..)))..)))...)))..... ( -51.30) >DroEre_CAF1 275417 115 + 1 GUCACUGG--AGUCCGGGGCUCGGGAAUCGGGAUACGCUUGG---CAGUCAGCUUUUGAUUUACGAGUUCCAGAGCUCCGGGAUGCCAAUCUGGCCAAUCUGGCCUAUCAACGGCUCAUC (((.((((--((..(((((((((..((((((((...(((...---.....)))))))))))..)))))))).)..)))))))))(((.....((((.....)))).......)))..... ( -46.50) >DroYak_CAF1 279173 117 + 1 GUCACUGGAGAGUCCGGGACUCGGGAAUCGGGAUUCGCUUGG---CAGUCAGCUUUUGAUUUACGAGUUUCAGGGCUCCGGCAUGCCAAUCUGACCAAUCUGGCCUAUCAAAGGCUCAUC ((((...(.(((((((..(((((..((((((((...(((...---.....)))))))))))..)))))..).)))))))((....))....))))......(((((.....))))).... ( -40.20) >consensus GUCACUGG__AGUCCGGGACUCGGGAAUCGGGAUUCGCUUGGCGGCAGUCAGCUUUUGAUUUACGAGUUCCAGGGCUCCGGGAUGCCAAUCUG_CCAAUCUGGCCUAUCAACGGCUCAUC (((.((((..(((((((((((((..((((((((...(((.(((....))))))))))))))..)))))))).)))))))))...((((............))))........)))..... (-40.22 = -40.50 + 0.28)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:16:00 2006