
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 5,053,517 – 5,053,626 |
| Length | 109 |
| Max. P | 0.999717 |

| Location | 5,053,517 – 5,053,626 |
|---|---|
| Length | 109 |
| Sequences | 5 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 93.11 |
| Mean single sequence MFE | -38.88 |
| Consensus MFE | -32.96 |
| Energy contribution | -33.76 |
| Covariance contribution | 0.80 |
| Combinations/Pair | 1.00 |
| Mean z-score | -3.71 |
| Structure conservation index | 0.85 |
| SVM decision value | 3.94 |
| SVM RNA-class probability | 0.999717 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
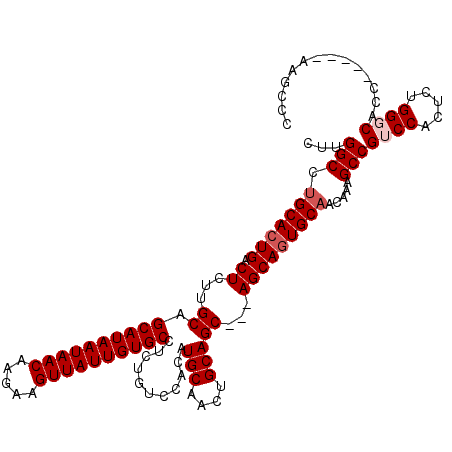
>3L_DroMel_CAF1 5053517 109 + 23771897 CUUGGCCUGCACUGACUCUUGCAGCAUAAUAACAAGAAGUUAUUGUGCCUCUGUCCACAUGCAACUGCAGC---AGCAAUGCAACAAAGCCGUCCACUC-GGCCACC-----AAGCCC (((((..((((.((.((.(((((((((((((((.....))))))))))...(((......)))..))))).---)))).)))).....((((......)-)))..))-----)))... ( -34.50) >DroSec_CAF1 260016 110 + 1 CUUGGCCUGCACUGACUCUUGCAGCAUAAUAACAAGAAGUUAUUGUGCCUCUGUCCACAUGCAACUGCAGC---AGCAGUGCAACAAAGCCGUCCACUCUGGGCACC-----AAGCCC (((((..(((((((.((.(((((((((((((((.....))))))))))...(((......)))..))))).---)))))))))........((((.....)))).))-----)))... ( -39.80) >DroSim_CAF1 281302 110 + 1 CUUGGCCUGCACUGACUCUUGCAGCAUAAUAACAAGAAGUUAUUGUGCCUCUGUCCACAUGCAACUGCAGC---AGCAGUGCAACAAAGCCGUCCACUCUGGGCACC-----AAGCCC (((((..(((((((.((.(((((((((((((((.....))))))))))...(((......)))..))))).---)))))))))........((((.....)))).))-----)))... ( -39.80) >DroEre_CAF1 266737 115 + 1 CUUGGCCCGCACUGACUCUUGCAGCAUAAUAACAAGAAGUUAUUGUGCCUCUGUCCACAUGCAACUGCAGC---AGCAGUGCAACAAAGCCGGCCACUCUGGGCUCUGUGCUCCGUGC ..(((((.((...(((....(..((((((((((.....))))))))))..).)))....((((.((((...---.)))))))).....)).)))))....((((.....)).)).... ( -39.60) >DroYak_CAF1 270038 113 + 1 CUUGGCCUGCACUGACUCUUGCAGCAUAAUAACAAGAAGUUAUUGUGCCUCUGUCCACAUGCAACUGCAGCAGCAGCAGUGCAACAAAGCCGUCCACUCUGGGCACC-----CAGCCC ...(((.(((((((.((.((((.((((((((((.....))))))))))...........(((....))))))).)))))))))........((((.....))))...-----..))). ( -40.70) >consensus CUUGGCCUGCACUGACUCUUGCAGCAUAAUAACAAGAAGUUAUUGUGCCUCUGUCCACAUGCAACUGCAGC___AGCAGUGCAACAAAGCCGUCCACUCUGGGCACC_____AAGCCC ...(((.(((((((.((...((.((((((((((.....))))))))))...........(((....)))))...))))))))).....)))((((.....)))).............. (-32.96 = -33.76 + 0.80)


| Location | 5,053,517 – 5,053,626 |
|---|---|
| Length | 109 |
| Sequences | 5 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 93.11 |
| Mean single sequence MFE | -41.54 |
| Consensus MFE | -33.64 |
| Energy contribution | -33.92 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.87 |
| Structure conservation index | 0.81 |
| SVM decision value | 1.09 |
| SVM RNA-class probability | 0.913528 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
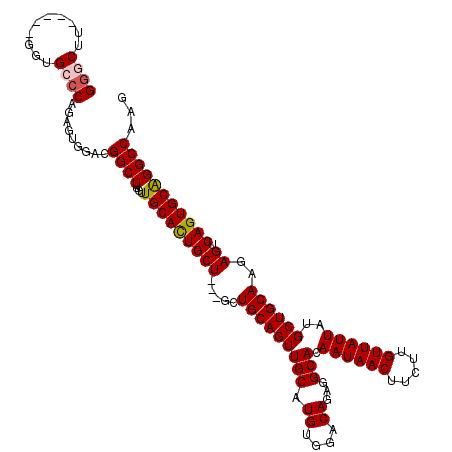
>3L_DroMel_CAF1 5053517 109 - 23771897 GGGCUU-----GGUGGCC-GAGUGGACGGCUUUGUUGCAUUGCU---GCUGCAGUUGCAUGUGGACAGAGGCACAAUAACUUCUUGUUAUUAUGCUGCAAGAGUCAGUGCAGGCCAAG ...(((-----(((((((-(......)))))...((((((((((---..(((((((((.((....))...))).((((((.....))))))..))))))..)).)))))))))))))) ( -39.90) >DroSec_CAF1 260016 110 - 1 GGGCUU-----GGUGCCCAGAGUGGACGGCUUUGUUGCACUGCU---GCUGCAGUUGCAUGUGGACAGAGGCACAAUAACUUCUUGUUAUUAUGCUGCAAGAGUCAGUGCAGGCCAAG ((((..-----...)))).........((((....(((((((((---..(((((((((.((....))...))).((((((.....))))))..))))))..)).)))))))))))... ( -41.60) >DroSim_CAF1 281302 110 - 1 GGGCUU-----GGUGCCCAGAGUGGACGGCUUUGUUGCACUGCU---GCUGCAGUUGCAUGUGGACAGAGGCACAAUAACUUCUUGUUAUUAUGCUGCAAGAGUCAGUGCAGGCCAAG ((((..-----...)))).........((((....(((((((((---..(((((((((.((....))...))).((((((.....))))))..))))))..)).)))))))))))... ( -41.60) >DroEre_CAF1 266737 115 - 1 GCACGGAGCACAGAGCCCAGAGUGGCCGGCUUUGUUGCACUGCU---GCUGCAGUUGCAUGUGGACAGAGGCACAAUAACUUCUUGUUAUUAUGCUGCAAGAGUCAGUGCGGGCCAAG ....((.((.....))))....(((((.((.((.((((.((((.---.((((....))).)..).))).((((.((((((.....)))))).)))))))).)).....)).))))).. ( -41.70) >DroYak_CAF1 270038 113 - 1 GGGCUG-----GGUGCCCAGAGUGGACGGCUUUGUUGCACUGCUGCUGCUGCAGUUGCAUGUGGACAGAGGCACAAUAACUUCUUGUUAUUAUGCUGCAAGAGUCAGUGCAGGCCAAG ((((..-----...)))).........((((....(((((((((((..((((....))).)..).))).((((.((((((.....)))))).))))........)))))))))))... ( -42.90) >consensus GGGCUU_____GGUGCCCAGAGUGGACGGCUUUGUUGCACUGCU___GCUGCAGUUGCAUGUGGACAGAGGCACAAUAACUUCUUGUUAUUAUGCUGCAAGAGUCAGUGCAGGCCAAG ((((..........)))).........((((....(((((((((.....(((((((((.((....))...))).((((((.....))))))..))))))..)).)))))))))))... (-33.64 = -33.92 + 0.28)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:15:35 2006