
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 4,772,600 – 4,772,800 |
| Length | 200 |
| Max. P | 0.999324 |

| Location | 4,772,600 – 4,772,720 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.83 |
| Mean single sequence MFE | -49.62 |
| Consensus MFE | -45.12 |
| Energy contribution | -45.20 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.26 |
| Structure conservation index | 0.91 |
| SVM decision value | 2.80 |
| SVM RNA-class probability | 0.997139 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 4772600 120 + 23771897 GGAGGAAAAGUACGGCAGGGAUCUGCAGAUCAUACUGGAGGAGUUCUGCCACCCCAUGCUGGUGGCCGGCCUGCUCACCCAGGAGCAGCUCUCUGCGAUCUUCCUGAAUACGGAAGAUCU ..............(((((((.((((........((((..((((.((((((((.......))))).)))...))))..))))..)))).)))))))((((((((.......)))))))). ( -50.70) >DroSec_CAF1 35357 120 + 1 GGAGGAGAAGUACGGCAGGGAUCUACAGAUCAUACUGGAGGAGUUCUGCCACCCCAUGCUGGUGGCCGGCCUGCUCACCCAGGAGCAGCUCUCCGCGAUCUUCCUGAACACGGAAGAUCU ...(((((.((...((...((((....))))...((((..((((.((((((((.......))))).)))...))))..))))..)).)))))))..((((((((.......)))))))). ( -50.10) >DroSim_CAF1 34296 120 + 1 GGAGGAGAAGUACGGCAGGGAUCUACAGAUCAUACUGGAGGAGUUCUGCCACCCCAUGCUGGUGGCCGGCCUGCUCACCCAGGAGCAGCUCUCCGCGAUCUUCCUGAACACGGAAGAUCU ...(((((.((...((...((((....))))...((((..((((.((((((((.......))))).)))...))))..))))..)).)))))))..((((((((.......)))))))). ( -50.10) >DroEre_CAF1 24651 120 + 1 GGAGGAGAAGUACGGCAGGGAUCUGCAGAUCAUACUGGAGGAGUUCUGCCACCCCAUGCUGGUGGCCGGCCUGCUCACCCAGGAGCAGCUCUCCGCGAUCUUCCUGAACACGGAAGAUCU ...(((((.((...((...((((....))))...((((..((((.((((((((.......))))).)))...))))..))))..)).)))))))..((((((((.......)))))))). ( -50.10) >DroYak_CAF1 31853 120 + 1 GGAGGAGAAGUACGGCAGGGAUCUGCAGAUCAUACUGGAGGAGUUCUGCCACCCAAUGCUGGUGGCCGGCCUGCUCACCCAGGAGCAGCUCUCCGCGAUCUUCCUGAACACGGAAGAUCU ...(((((.((...((...((((....))))...((((..((((.((((((((.......))))).)))...))))..))))..)).)))))))..((((((((.......)))))))). ( -50.10) >DroAna_CAF1 25593 120 + 1 GGAGGAAAAGUACGGACGGGACUUGCAGAUCAUUCUCGAGGAAUUCUGCCACCCGAUGCUGGUGGCCGGCCUGCUCACCCAAGAGCAGCUCUCCGCGAUCUUCCUGAACACGGAAGAUCU (((((...........((((....(((((..((((.....)))))))))..))))..(((((...)))))((((((......)))))).)))))..((((((((.......)))))))). ( -46.60) >consensus GGAGGAGAAGUACGGCAGGGAUCUGCAGAUCAUACUGGAGGAGUUCUGCCACCCCAUGCUGGUGGCCGGCCUGCUCACCCAGGAGCAGCUCUCCGCGAUCUUCCUGAACACGGAAGAUCU (((((........(((((((.(((.(((......))).)))..))))))).......(((((...)))))((((((......)))))).)))))..((((((((.......)))))))). (-45.12 = -45.20 + 0.08)



| Location | 4,772,600 – 4,772,720 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.83 |
| Mean single sequence MFE | -47.67 |
| Consensus MFE | -44.55 |
| Energy contribution | -44.30 |
| Covariance contribution | -0.25 |
| Combinations/Pair | 1.09 |
| Mean z-score | -1.70 |
| Structure conservation index | 0.93 |
| SVM decision value | 2.23 |
| SVM RNA-class probability | 0.990794 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 4772600 120 - 23771897 AGAUCUUCCGUAUUCAGGAAGAUCGCAGAGAGCUGCUCCUGGGUGAGCAGGCCGGCCACCAGCAUGGGGUGGCAGAACUCCUCCAGUAUGAUCUGCAGAUCCCUGCCGUACUUUUCCUCC .((((((((.......))))))))((((.((.(((((((((((.(((...((((.((.((.....))))))))....))).)))))...))...)))).)).)))).............. ( -45.50) >DroSec_CAF1 35357 120 - 1 AGAUCUUCCGUGUUCAGGAAGAUCGCGGAGAGCUGCUCCUGGGUGAGCAGGCCGGCCACCAGCAUGGGGUGGCAGAACUCCUCCAGUAUGAUCUGUAGAUCCCUGCCGUACUUCUCCUCC .((((((((.......))))))))..(((((((..((((..((((.((......)))))).....))))..))...........((((((....((((....)))))))))))))))... ( -47.80) >DroSim_CAF1 34296 120 - 1 AGAUCUUCCGUGUUCAGGAAGAUCGCGGAGAGCUGCUCCUGGGUGAGCAGGCCGGCCACCAGCAUGGGGUGGCAGAACUCCUCCAGUAUGAUCUGUAGAUCCCUGCCGUACUUCUCCUCC .((((((((.......))))))))..(((((((..((((..((((.((......)))))).....))))..))...........((((((....((((....)))))))))))))))... ( -47.80) >DroEre_CAF1 24651 120 - 1 AGAUCUUCCGUGUUCAGGAAGAUCGCGGAGAGCUGCUCCUGGGUGAGCAGGCCGGCCACCAGCAUGGGGUGGCAGAACUCCUCCAGUAUGAUCUGCAGAUCCCUGCCGUACUUCUCCUCC .((((((((.......))))))))..(((((((..((((..((((.((......)))))).....))))..))...........((((((....((((....)))))))))))))))... ( -49.80) >DroYak_CAF1 31853 120 - 1 AGAUCUUCCGUGUUCAGGAAGAUCGCGGAGAGCUGCUCCUGGGUGAGCAGGCCGGCCACCAGCAUUGGGUGGCAGAACUCCUCCAGUAUGAUCUGCAGAUCCCUGCCGUACUUCUCCUCC .((((((((.......))))))))..(((((.((((((......))))))....((((((.......))))))...........((((((....((((....)))))))))))))))... ( -48.70) >DroAna_CAF1 25593 120 - 1 AGAUCUUCCGUGUUCAGGAAGAUCGCGGAGAGCUGCUCUUGGGUGAGCAGGCCGGCCACCAGCAUCGGGUGGCAGAAUUCCUCGAGAAUGAUCUGCAAGUCCCGUCCGUACUUUUCCUCC .((((((((.......))))))))(((((...((((((......))))))((.((...)).))..((((..(((((((((.....))))..)))))....)))))))))........... ( -46.40) >consensus AGAUCUUCCGUGUUCAGGAAGAUCGCGGAGAGCUGCUCCUGGGUGAGCAGGCCGGCCACCAGCAUGGGGUGGCAGAACUCCUCCAGUAUGAUCUGCAGAUCCCUGCCGUACUUCUCCUCC .((((((((.......))))))))..((((((.(((....(((...((((..((((((((.......))))))...(((.....))).))..))))....)))....))).))))))... (-44.55 = -44.30 + -0.25)

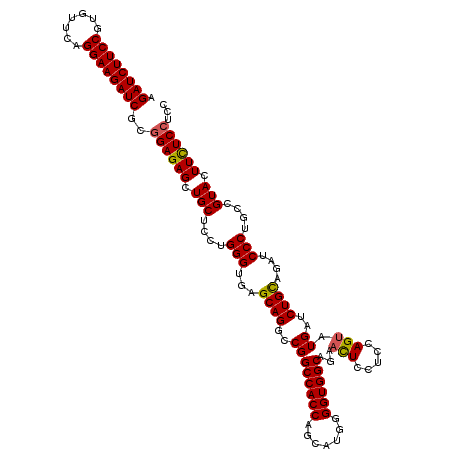
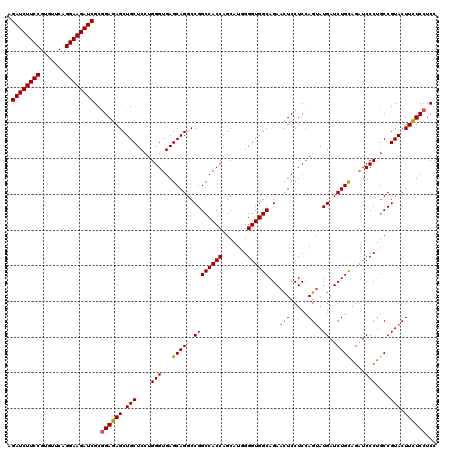
| Location | 4,772,640 – 4,772,760 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.22 |
| Mean single sequence MFE | -50.22 |
| Consensus MFE | -47.78 |
| Energy contribution | -47.95 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.13 |
| Structure conservation index | 0.95 |
| SVM decision value | 3.51 |
| SVM RNA-class probability | 0.999324 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 4772640 120 + 23771897 GAGUUCUGCCACCCCAUGCUGGUGGCCGGCCUGCUCACCCAGGAGCAGCUCUCUGCGAUCUUCCUGAAUACGGAAGAUCUGCUCGAGAACAACCAGACGCUGGCCGAGCGCAUGCGCGAC ..((((.((((........))))(((((((((((((......)))))).((((.((((((((((.......)))))))).))..))))..........)))))))))))((....))... ( -50.10) >DroSec_CAF1 35397 120 + 1 GAGUUCUGCCACCCCAUGCUGGUGGCCGGCCUGCUCACCCAGGAGCAGCUCUCCGCGAUCUUCCUGAACACGGAAGAUCUGCUCGAGAACAACCAGACGCUGGCCGAGCGUAUGCGCGAC .......((((((.......))))))((((((((((......)))))(.((((.((((((((((.......)))))))).))..)))).)...........))))).(((....)))... ( -49.00) >DroSim_CAF1 34336 120 + 1 GAGUUCUGCCACCCCAUGCUGGUGGCCGGCCUGCUCACCCAGGAGCAGCUCUCCGCGAUCUUCCUGAACACGGAAGAUCUGCUCGAGAACAACCAGACGCUGGCCGAGCGUAUGCGCGAC .......((((((.......))))))((((((((((......)))))(.((((.((((((((((.......)))))))).))..)))).)...........))))).(((....)))... ( -49.00) >DroEre_CAF1 24691 120 + 1 GAGUUCUGCCACCCCAUGCUGGUGGCCGGCCUGCUCACCCAGGAGCAGCUCUCCGCGAUCUUCCUGAACACGGAAGAUCUGCUAGAGAACAACCAGACGCUGGCCGAGCGCAUGCGCGAC ..((((.((((........))))(((((((((((((......))))))((((..((((((((((.......)))))))).)).))))...........)))))))))))((....))... ( -49.40) >DroYak_CAF1 31893 120 + 1 GAGUUCUGCCACCCAAUGCUGGUGGCCGGCCUGCUCACCCAGGAGCAGCUCUCCGCGAUCUUCCUGAACACGGAAGAUCUGCUCGAGAACAACCAGACGCUGGCCGAGCGCAUGCGCGAC ..((((.((((........))))(((((((((((((......)))))).((((.((((((((((.......)))))))).))..))))..........)))))))))))((....))... ( -49.90) >DroAna_CAF1 25633 120 + 1 GAAUUCUGCCACCCGAUGCUGGUGGCCGGCCUGCUCACCCAAGAGCAGCUCUCCGCGAUCUUCCUGAACACGGAAGAUCUGCUCGAGAACAACCAGACGCUGGCCGAGCGGAUGCGGGAC ...........((((...(((.((((((((((((((......)))))).((((.((((((((((.......)))))))).))..))))..........))))))))..)))...)))).. ( -53.90) >consensus GAGUUCUGCCACCCCAUGCUGGUGGCCGGCCUGCUCACCCAGGAGCAGCUCUCCGCGAUCUUCCUGAACACGGAAGAUCUGCUCGAGAACAACCAGACGCUGGCCGAGCGCAUGCGCGAC .......((((((.......))))))((((((((((......)))))(.((((.((((((((((.......)))))))).))..)))).)...........))))).(((....)))... (-47.78 = -47.95 + 0.17)



| Location | 4,772,640 – 4,772,760 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.22 |
| Mean single sequence MFE | -56.17 |
| Consensus MFE | -53.65 |
| Energy contribution | -53.82 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.03 |
| Structure conservation index | 0.96 |
| SVM decision value | 3.39 |
| SVM RNA-class probability | 0.999129 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 4772640 120 - 23771897 GUCGCGCAUGCGCUCGGCCAGCGUCUGGUUGUUCUCGAGCAGAUCUUCCGUAUUCAGGAAGAUCGCAGAGAGCUGCUCCUGGGUGAGCAGGCCGGCCACCAGCAUGGGGUGGCAGAACUC (((((.((((((((((.((((.(...(((.((((((..((.((((((((.......)))))))))).)))))).)))))))).))))).((....))....)))).).)))))....... ( -56.50) >DroSec_CAF1 35397 120 - 1 GUCGCGCAUACGCUCGGCCAGCGUCUGGUUGUUCUCGAGCAGAUCUUCCGUGUUCAGGAAGAUCGCGGAGAGCUGCUCCUGGGUGAGCAGGCCGGCCACCAGCAUGGGGUGGCAGAACUC ..((.((....(((((.((((.(...(((.((((((..((.((((((((.......)))))))))).)))))).)))))))).)))))..))))((((((.......))))))....... ( -55.50) >DroSim_CAF1 34336 120 - 1 GUCGCGCAUACGCUCGGCCAGCGUCUGGUUGUUCUCGAGCAGAUCUUCCGUGUUCAGGAAGAUCGCGGAGAGCUGCUCCUGGGUGAGCAGGCCGGCCACCAGCAUGGGGUGGCAGAACUC ..((.((....(((((.((((.(...(((.((((((..((.((((((((.......)))))))))).)))))).)))))))).)))))..))))((((((.......))))))....... ( -55.50) >DroEre_CAF1 24691 120 - 1 GUCGCGCAUGCGCUCGGCCAGCGUCUGGUUGUUCUCUAGCAGAUCUUCCGUGUUCAGGAAGAUCGCGGAGAGCUGCUCCUGGGUGAGCAGGCCGGCCACCAGCAUGGGGUGGCAGAACUC (((((.((((((((((.((((.(...(((.(((((((.((.((((((((.......))))))))))))))))).)))))))).))))).((....))....)))).).)))))....... ( -57.20) >DroYak_CAF1 31893 120 - 1 GUCGCGCAUGCGCUCGGCCAGCGUCUGGUUGUUCUCGAGCAGAUCUUCCGUGUUCAGGAAGAUCGCGGAGAGCUGCUCCUGGGUGAGCAGGCCGGCCACCAGCAUUGGGUGGCAGAACUC (((((.(((((....((((.(.(((((.(..(((..((((((.(((.(((((.((.....)).)))))))).))))))..)))..).))))))))))....))).)).)))))....... ( -57.20) >DroAna_CAF1 25633 120 - 1 GUCCCGCAUCCGCUCGGCCAGCGUCUGGUUGUUCUCGAGCAGAUCUUCCGUGUUCAGGAAGAUCGCGGAGAGCUGCUCUUGGGUGAGCAGGCCGGCCACCAGCAUCGGGUGGCAGAAUUC .((((((....((((((((((...)))))......))))).((((((((.......)))))))))))).)).((((((......))))))....((((((.......))))))....... ( -55.10) >consensus GUCGCGCAUGCGCUCGGCCAGCGUCUGGUUGUUCUCGAGCAGAUCUUCCGUGUUCAGGAAGAUCGCGGAGAGCUGCUCCUGGGUGAGCAGGCCGGCCACCAGCAUGGGGUGGCAGAACUC ...(((....))).(((((...........((((((..((.((((((((.......)))))))))).))))))(((((......))))))))))((((((.......))))))....... (-53.65 = -53.82 + 0.17)

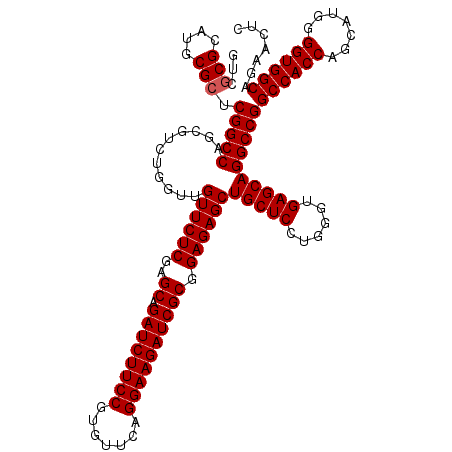

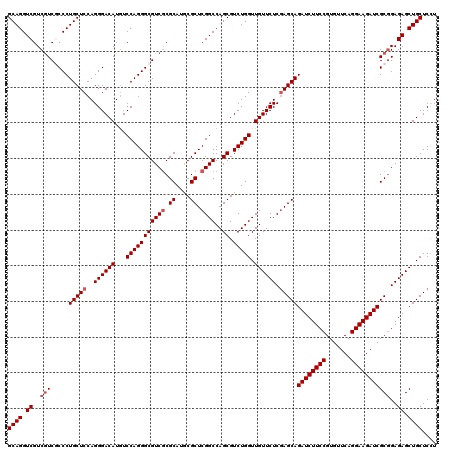
| Location | 4,772,680 – 4,772,800 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.94 |
| Mean single sequence MFE | -54.35 |
| Consensus MFE | -50.63 |
| Energy contribution | -51.30 |
| Covariance contribution | 0.67 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.00 |
| Structure conservation index | 0.93 |
| SVM decision value | 2.81 |
| SVM RNA-class probability | 0.997178 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 4772680 120 - 23771897 GCAGGUCGUCGUCGCCCUGCUCCAGGGACAUGUCCAGGGCGUCGCGCAUGCGCUCGGCCAGCGUCUGGUUGUUCUCGAGCAGAUCUUCCGUAUUCAGGAAGAUCGCAGAGAGCUGCUCCU ((((.((((((.(((((((...((......))..))))))).)).)).(((((((((((((...)))))......))))).((((((((.......)))))))))))..)).)))).... ( -54.90) >DroSec_CAF1 35437 120 - 1 GCAGGUCGUCGUCGCCCUGCUCCAGGGACAUGUCCAGGGCGUCGCGCAUACGCUCGGCCAGCGUCUGGUUGUUCUCGAGCAGAUCUUCCGUGUUCAGGAAGAUCGCGGAGAGCUGCUCCU ((((.((....((((..(((((..((((((...(((((((((((.((....)).))))..)).))))).)))))).)))))((((((((.......)))))))))))).)).)))).... ( -55.20) >DroSim_CAF1 34376 120 - 1 GCAGGUCGUCAUCUCCCUGCUCCAGGGACAUGUCCAGGGCGUCGCGCAUACGCUCGGCCAGCGUCUGGUUGUUCUCGAGCAGAUCUUCCGUGUUCAGGAAGAUCGCGGAGAGCUGCUCCU ((.(((((.(((.((((((...)))))).))).)..((((((.......)))))))))).))....(((.((((((..((.((((((((.......)))))))))).)))))).)))... ( -52.50) >DroEre_CAF1 24731 120 - 1 GCAGGUCGUCGUCGCCCUGCUCCAGGGACAUGUCCAGGGCGUCGCGCAUGCGCUCGGCCAGCGUCUGGUUGUUCUCUAGCAGAUCUUCCGUGUUCAGGAAGAUCGCGGAGAGCUGCUCCU ((.(((((..(.(((((((...((......))..))))))).)(((....))).))))).))....(((.(((((((.((.((((((((.......))))))))))))))))).)))... ( -54.60) >DroYak_CAF1 31933 120 - 1 GCAGGUCGUCGUCGCCCUGCUCCAGGGACAUGUCCAGGGCGUCGCGCAUGCGCUCGGCCAGCGUCUGGUUGUUCUCGAGCAGAUCUUCCGUGUUCAGGAAGAUCGCGGAGAGCUGCUCCU ((((.((....((((..(((((..((((((...(((((((((((.((....)).))))..)).))))).)))))).)))))((((((((.......)))))))))))).)).)))).... ( -55.20) >DroAna_CAF1 25673 120 - 1 GCAGGUCGUCGUCCCCCUGCUCCAGGGACAUAUCCAGGGCGUCCCGCAUCCGCUCGGCCAGCGUCUGGUUGUUCUCGAGCAGAUCUUCCGUGUUCAGGAAGAUCGCGGAGAGCUGCUCUU ((((.(((.((((((((((...))))).........))))).)((((....((((((((((...)))))......))))).((((((((.......)))))))))))).)).)))).... ( -53.70) >consensus GCAGGUCGUCGUCGCCCUGCUCCAGGGACAUGUCCAGGGCGUCGCGCAUGCGCUCGGCCAGCGUCUGGUUGUUCUCGAGCAGAUCUUCCGUGUUCAGGAAGAUCGCGGAGAGCUGCUCCU ((((.((..(((.....(((((..((((((...(((((((((((.((....)).))))..)).))))).)))))).)))))((((((((.......)))))))))))..)).)))).... (-50.63 = -51.30 + 0.67)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:13:00 2006