
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 4,658,821 – 4,658,941 |
| Length | 120 |
| Max. P | 0.976384 |

| Location | 4,658,821 – 4,658,941 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 85.44 |
| Mean single sequence MFE | -42.90 |
| Consensus MFE | -28.53 |
| Energy contribution | -28.45 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.19 |
| Mean z-score | -3.30 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.22 |
| SVM RNA-class probability | 0.641539 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
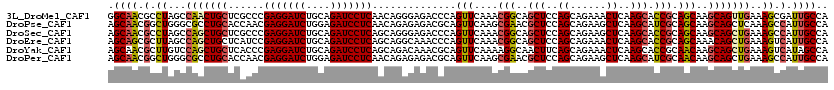
>3L_DroMel_CAF1 4658821 120 + 23771897 GGCAACGCCUAGCCAACUGCUCGCCCGAGGAUCUGCAGAUCCUCAACAGGGAGACCCAGUUCAAACGGCAGCUCCAGCAGAAACUCAAGCACCGCAGCAAGCAGUUGAAAGCGAUUGCCA ((((((((.....((((((((.((..(((((((....)))))))....((....)).........(((..(((..((......))..))).)))..)).))))))))...))).))))). ( -46.90) >DroPse_CAF1 36609 120 + 1 AGCAACGGCUGGGCGCCUGCACCAACGAGGAUCUGGAGAUCCUCAACAGAGAGACGCAGUUCAAGCGAACGCUCCAGCAGAAGCUCAAGCAUCGCAGCAAGCAGCUCAAAGCCAUUGCCA .((((.((((((((((.(((......(((((((....)))))))...........((.((((....))))(((..(((....)))..)))...)).))).)).))))..)))).)))).. ( -47.00) >DroSec_CAF1 31083 120 + 1 AGCAACGCCUAGCCAGCUGCUCGCCCGAGGAUCUGCAGAUCCUCAGCAGGGAGACCCAGUUCAAACGGCAGCUCCAGCAGAAGCUCAAGCACCGCAGCAAGCAGCUGAAAGCCAUUGCCA .((((.(.((...((((((((.((..(((((((....))))))).((.((....))..........((..(((..(((....)))..))).)))).)).))))))))..)).).)))).. ( -47.40) >DroEre_CAF1 31338 120 + 1 AGCAGCGCUUAGCCAGCUGCUCAUCCGAGGAUCUGCAGAUCCUCAGCAGGCAAACCCAGUUCAAACGGCAGCUCCAGCAGAAACUCAAGCACCGCAGCAAACAGCUGAAAGUCAUUGCCA ((((((.........)))))).....(((((((....)))))))....(((((............(((..(((..((......))..))).)))((((.....)))).......))))). ( -38.20) >DroYak_CAF1 35171 120 + 1 AGCAACGCUUGUCCAGCUGCUCACCCGAGGAUCUGCAGAUCCUCAGCAGACAAACGCAGUUCAAAAGGCAACUUCAGCAGAAACUCAAGCACCGCAACAAGCAGCUGAAAGUCAUAGCCA (((...(((((.....(((((.....(((((((....))))))).((........)).......(((....))).))))).....)))))...((.....)).))).............. ( -33.90) >DroPer_CAF1 36715 120 + 1 AGCAACGGCUGGGCGCCUGCACCAACGAGGAUCUGGAGAUCCUCAACAGAGAGACGCAGUUCAAGCGAACGCUCCAGCAGAAGCUCAAGCAUCGCAACAAGCAGCUGAAAGCCAUUGCCA .((((.((((...((.((((......(((((((....)))))))...........((.((((....))))(((..(((....)))..)))...)).....)))).))..)))).)))).. ( -44.00) >consensus AGCAACGCCUAGCCAGCUGCUCACCCGAGGAUCUGCAGAUCCUCAACAGAGAGACCCAGUUCAAACGGCAGCUCCAGCAGAAACUCAAGCACCGCAGCAAGCAGCUGAAAGCCAUUGCCA .((((.(.((...(((((((......(((((((....)))))))..............(((....(((..(((..((......))..))).))).)))..)))))))..)).).)))).. (-28.53 = -28.45 + -0.08)

| Location | 4,658,821 – 4,658,941 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 85.44 |
| Mean single sequence MFE | -57.67 |
| Consensus MFE | -44.28 |
| Energy contribution | -45.40 |
| Covariance contribution | 1.12 |
| Combinations/Pair | 1.23 |
| Mean z-score | -4.59 |
| Structure conservation index | 0.77 |
| SVM decision value | 1.77 |
| SVM RNA-class probability | 0.976384 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
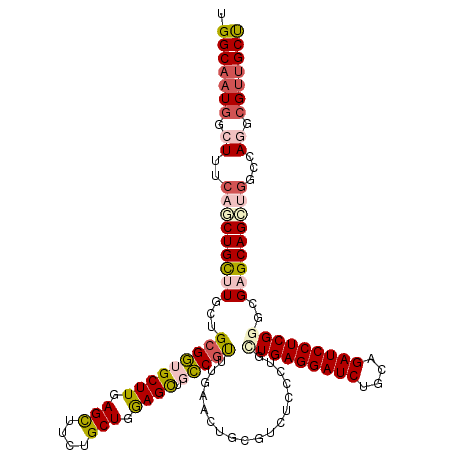
>3L_DroMel_CAF1 4658821 120 - 23771897 UGGCAAUCGCUUUCAACUGCUUGCUGCGGUGCUUGAGUUUCUGCUGGAGCUGCCGUUUGAACUGGGUCUCCCUGUUGAGGAUCUGCAGAUCCUCGGGCGAGCAGUUGGCUAGGCGUUGCC .(((((.(((((((((((((((((((((((((((.(((....))).)))).)))))...(((.(((...))).)))(((((((....))))))).)))))))))))))..)))))))))) ( -63.80) >DroPse_CAF1 36609 120 - 1 UGGCAAUGGCUUUGAGCUGCUUGCUGCGAUGCUUGAGCUUCUGCUGGAGCGUUCGCUUGAACUGCGUCUCUCUGUUGAGGAUCUCCAGAUCCUCGUUGGUGCAGGCGCCCAGCCGUUGCU .(((((((((((..(((.((.....))(((((((.(((....))).))))))).)))..)...(((((((.((..((((((((....))))))))..)).).))))))..)))))))))) ( -58.50) >DroSec_CAF1 31083 120 - 1 UGGCAAUGGCUUUCAGCUGCUUGCUGCGGUGCUUGAGCUUCUGCUGGAGCUGCCGUUUGAACUGGGUCUCCCUGCUGAGGAUCUGCAGAUCCUCGGGCGAGCAGCUGGCUAGGCGUUGCU .(((((((.((.((((((((((((.(((((((((.(((....))).)))).))))).......(((...)))..(((((((((....)))))))))))))))))))))..)).))))))) ( -67.60) >DroEre_CAF1 31338 120 - 1 UGGCAAUGACUUUCAGCUGUUUGCUGCGGUGCUUGAGUUUCUGCUGGAGCUGCCGUUUGAACUGGGUUUGCCUGCUGAGGAUCUGCAGAUCCUCGGAUGAGCAGCUGGCUAAGCGCUGCU .((((.((.(((((((((((((...(((((((((.(((....))).)))).))))).......(((....))).(((((((((....)))))))))..))))))))))..))))).)))) ( -51.00) >DroYak_CAF1 35171 120 - 1 UGGCUAUGACUUUCAGCUGCUUGUUGCGGUGCUUGAGUUUCUGCUGAAGUUGCCUUUUGAACUGCGUUUGUCUGCUGAGGAUCUGCAGAUCCUCGGGUGAGCAGCUGGACAAGCGUUGCU .(((.(((.((((((((((((..(.(((((((((.(((....))).))))..........))))).........(((((((((....))))))))))..))))))))))..))))).))) ( -48.20) >DroPer_CAF1 36715 120 - 1 UGGCAAUGGCUUUCAGCUGCUUGUUGCGAUGCUUGAGCUUCUGCUGGAGCGUUCGCUUGAACUGCGUCUCUCUGUUGAGGAUCUCCAGAUCCUCGUUGGUGCAGGCGCCCAGCCGUUGCU .((((((((((((((((.((.....))(((((((.(((....))).))))))).)).))))..(((((((.((..((((((((....))))))))..)).).))))))..)))))))))) ( -56.90) >consensus UGGCAAUGGCUUUCAGCUGCUUGCUGCGGUGCUUGAGCUUCUGCUGGAGCUGCCGUUUGAACUGCGUCUCCCUGCUGAGGAUCUGCAGAUCCUCGGGCGAGCAGCUGGCCAGGCGUUGCU .(((((((.((..(((((((((...(((((((((.(((....))).)))).)))))..................(((((((((....)))))))))..)))))))))...)).))))))) (-44.28 = -45.40 + 1.12)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:11:29 2006