
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 4,648,150 – 4,648,256 |
| Length | 106 |
| Max. P | 0.824933 |

| Location | 4,648,150 – 4,648,256 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 84.34 |
| Mean single sequence MFE | -30.71 |
| Consensus MFE | -23.01 |
| Energy contribution | -25.85 |
| Covariance contribution | 2.84 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.84 |
| Structure conservation index | 0.75 |
| SVM decision value | 0.69 |
| SVM RNA-class probability | 0.824933 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 4648150 106 + 23771897 AAUUUGUGGACUU-AGCAAUUUGUGGCG-------------AUGUGUUUGUGUUUGGGUUAAGCAAACUAAAACACAGACACCAAAUUGCUGGGGUUUAUCGAGUGUGACUUAUUAACGA .((((((((((((-(((((((((.....-------------..((((((((((((.((((.....)))).))))))))))))))))))))).))))))).)))))............... ( -34.51) >DroSec_CAF1 20574 106 + 1 AAUUUGUGGACUU-AGCAAUUUGUGGCG-------------AUGUGUUUGUGUUUGGGUUUAGCAAACUAAAACACGGACACCAAAUUGCUUGGGUUUAUCGAGUGUGACUUAUUAACGA .((((((((((((-(((((((((.....-------------..((((((((((((.((((.....)))).)))))))))))))))))))).)))))))).)))))............... ( -33.51) >DroSim_CAF1 21149 106 + 1 AAUUUGUGGACUU-AGCAAUUUGUGGCG-------------AUGUGUUUGUGUUUGGGUUAAGCAAACUAAAACACAGACACCAAAUUGCUUGGGUUUAUCGAGUGUGACUUAUUAACGA .((((((((((((-(((((((((.....-------------..((((((((((((.((((.....)))).)))))))))))))))))))).)))))))).)))))............... ( -34.51) >DroEre_CAF1 20796 106 + 1 AAUUUGUGGACUU-AGCAAUUUGUGGCG-------------AUGUGUUUGUGUUUGGGCUAAGCAAACUAAAACACAGACACCAAAUUUCUUGGGUUUAUCGAGUGUGACUUAUUAACGA .((((((((((((-((.((((((.....-------------..((((((((((((((..........)).))))))))))))))))))..))))))))).)))))............... ( -24.71) >DroYak_CAF1 24035 106 + 1 AAUUUGUGGACUU-AGCAAUUUGUGGCG-------------AUGUGUUUGUGUUUGGGUUAAGCAAACUAAAACACAGACAGCAAAUUGCUUGGGUUUAUUGAGUGUGACUUAUUAACAA .....((((((((-((((((((((....-------------...(((((((((((.((((.....)))).)))))))))))))))))))).)))))))))((((.....))))....... ( -32.41) >DroAna_CAF1 20730 107 + 1 AAUUUGUGGACUCAAGCAAUUUGUGGGUGUAUGUUUGGCGUGUGUGUUUAUAUUUGAGCUAAACAAACCA-AACACAGAC-----------UGAGUGUAA-AAGUUUGAUUUAUUAAUGG (((((.((.((((..((.....)).(((.(.(((((((..(((..(((((....)))))...)))..)))-)))).).))-----------))))).)).-))))).............. ( -24.60) >consensus AAUUUGUGGACUU_AGCAAUUUGUGGCG_____________AUGUGUUUGUGUUUGGGUUAAGCAAACUAAAACACAGACACCAAAUUGCUUGGGUUUAUCGAGUGUGACUUAUUAACGA .((((((((((((.(((((((((....................((((((((((((.((((.....)))).))))))))))))))))))))).))))))).)))))............... (-23.01 = -25.85 + 2.84)

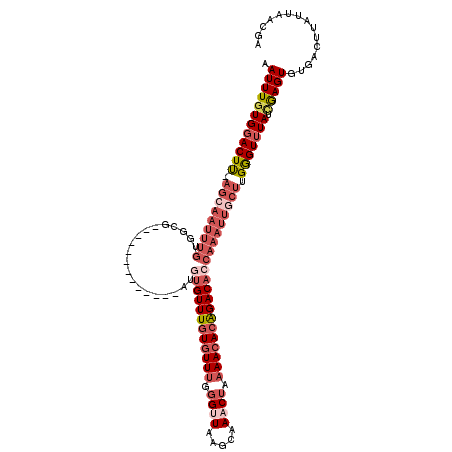
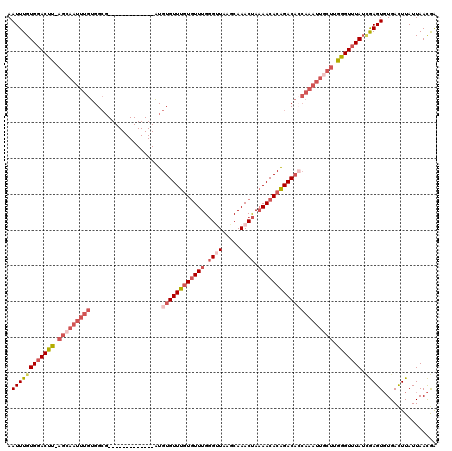
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:11:23 2006