
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 4,647,203 – 4,647,358 |
| Length | 155 |
| Max. P | 0.979649 |

| Location | 4,647,203 – 4,647,318 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 115 |
| Reading direction | reverse |
| Mean pairwise identity | 73.51 |
| Mean single sequence MFE | -45.08 |
| Consensus MFE | -19.49 |
| Energy contribution | -18.92 |
| Covariance contribution | -0.57 |
| Combinations/Pair | 1.57 |
| Mean z-score | -1.79 |
| Structure conservation index | 0.43 |
| SVM decision value | -0.05 |
| SVM RNA-class probability | 0.507038 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
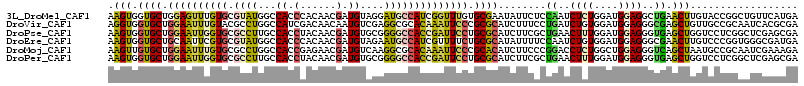
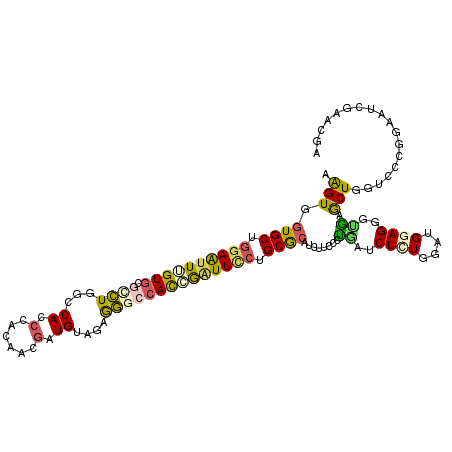
>3L_DroMel_CAF1 4647203 115 - 23771897 AAGUGGUGCUGGAGUUUGUGCGUAUGGCCACCCACAACGAUGUAGGAUGCCAUCGGUUUGUGCGAAUAUUCUCCAAUCUCUGGAUGGAGGCUGAACUUGUACCGGCUGUUCAUGA ..((((.(((((.(((((..((.(((((..((.(((....))).))..))))).....))..)))))..((((((.((....))))))))...........)))))...)))).. ( -34.40) >DroVir_CAF1 34591 115 - 1 AGGUGGUGCUGGAAUUUGUACGCCUGGCCAUCGACAACAAUGUCGAGGCGCACAAAUUCCCGCGCAUCUUUCCUGAUCUGUGGAUGGAGGGCGAGCUGUUGCCGCAAUCACGCGA ..(((((((.(((((((((.((((......((((((....)))))))))).))))))))).))((.(((.(((........))).))).(((((....))))))).))))).... ( -46.60) >DroPse_CAF1 24687 115 - 1 AAGUGGUGCUGGAAUUGGUGCGCCUUGCCACCUACAACGAUGUGCGGGGCCACCGAUUCCUGCGCAUCUUCGCUGAACUUUGGAUGGAGGGUGAGCUGGUCCUCGGCUCGAGCGA .....((((.((((((((((.(((((((((.(......).)).))))))))))))))))).))))....(((((..(((((......)))))((((((.....)))))).))))) ( -52.60) >DroEre_CAF1 19851 115 - 1 AAGUGGUGCUGCAAUUCGUGCGUAUGGCCACCCACAACGAUGUAGAAUGCCAUCGUUUUCUGCGCAUAUUUUCCAAUCUGUGGAUGGAGGGCGAACUUGUCCCGGUGGGCGAUGA ..((.((((.(((.....))))))).))(.(((((...(.(((((((.((....)).))))))))......((((.((....))))))(((((....)))))..))))).).... ( -32.70) >DroMoj_CAF1 29928 115 - 1 AAGUUGUGCUGGAAUUUGUGCGCCUGGCCACCGAGAACGAUGUCAAGGCGCACAAAUUCCCGCACAUCUUCCCGGACCUCUGGCUGGAGGGUCAGCUAAUGCCGCAAUCGAAAGA (((.(((((.((((((((((((((((((...((....))..))).))))))))))))))).))))).)))..(((.....(((((((....)))))))...)))...((....)) ( -51.60) >DroPer_CAF1 24859 115 - 1 AAGUGGUGCUGGAAUUGGUGCGCCUUGCCACCUACAACGAUGUGCGGGGCCACCGAUUCCUGCGCAUCUUCGCUGAACUUUGGAUGGAGGGUGAGCUGGUCCUCGGCUCGAGCGA .....((((.((((((((((.(((((((((.(......).)).))))))))))))))))).))))....(((((..(((((......)))))((((((.....)))))).))))) ( -52.60) >consensus AAGUGGUGCUGGAAUUUGUGCGCCUGGCCACCCACAACGAUGUAGAGGGCCACCGAUUCCUGCGCAUCUUCCCUGAUCUCUGGAUGGAGGGUGAGCUGGUCCCGGAAUCGAACGA .(((.((((.((((((((((.((((...((.(......).))....)))))))))))))).))))........((..((((....))))..)).))).................. (-19.49 = -18.92 + -0.57)
| Location | 4,647,241 – 4,647,358 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 77.15 |
| Mean single sequence MFE | -45.42 |
| Consensus MFE | -16.54 |
| Energy contribution | -18.18 |
| Covariance contribution | 1.64 |
| Combinations/Pair | 1.31 |
| Mean z-score | -2.34 |
| Structure conservation index | 0.36 |
| SVM decision value | 0.08 |
| SVM RNA-class probability | 0.572358 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
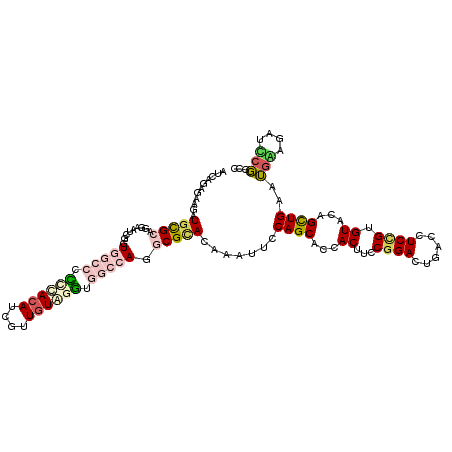
>3L_DroMel_CAF1 4647241 117 + 23771897 AUUGGAGAAUAUUCGCACAAACCGAUGGCAUCCUACAUCGUUGUGGGUGGCCAUACGCACAAACUCCAGCACCACUUCCGGACUCAGUUCCGUGUAGAGUUGUAGGAGUAAUCUGCC ...(((((..............(((((((..((((((....))))))..))))).)).....(((((.(((.(.((.(((((......)))).).)).).))).)))))..))).)) ( -35.20) >DroVir_CAF1 34629 117 + 1 AUCAGGAAAGAUGCGCGGGAAUUUGUGCGCCUCGACAUUGUUGUCGAUGGCCAGGCGUACAAAUUCCAGCACCACCUCCGGACUGACCUCUGUGUAGAGCUGAAUGCAGAUGCGGCC ....(((..(.(((...(((((((((((((((.(.(((((....))))).).))))))))))))))).))).)...)))((.(((.(.(((((((........))))))).)))))) ( -47.60) >DroPse_CAF1 24725 117 + 1 UUCAGCGAAGAUGCGCAGGAAUCGGUGGCCCCGCACAUCGUUGUAGGUGGCAAGGCGCACCAAUUCCAGCACCACUUCCGGACUGACCUCCGUGUACAGCUGAAUGACGAUCCGGCC ((((((((((..(.((.(((((.(((((((..((.((((......))))))..))).)))).))))).)).)..))))((((......))))......))))))............. ( -42.90) >DroEre_CAF1 19889 117 + 1 AUUGGAAAAUAUGCGCAGAAAACGAUGGCAUUCUACAUCGUUGUGGGUGGCCAUACGCACGAAUUGCAGCACCACUUCCGGACUGAGUUCCGUGUAGAGCUGCAGCAGUAAUCUGCC ..............(((((...(((((((..((((((....))))))..))))).)).((...(((((((.(.((...((((......)))).)).).)))))))..))..))))). ( -37.60) >DroMoj_CAF1 29966 117 + 1 GUCCGGGAAGAUGUGCGGGAAUUUGUGCGCCUUGACAUCGUUCUCGGUGGCCAGGCGCACAAAUUCCAGCACAACUUCCGGACUGACCUCGGUGUACAGCUGAAUGCAAAUGCGGCC ((((((.(((.(((((.(((((((((((((((.(.(((((....))))).).))))))))))))))).))))).)))))))))...(((((((.....)))))..((....)))).. ( -63.40) >DroPer_CAF1 24897 117 + 1 UUCAGCGAAGAUGCGCAGGAAUCGGUGGCCCCGCACAUCGUUGUAGGUGGCAAGGCGCACCAAUUCCAGCACCACUUCCGGACUGACCUCCGUGUACAGCUGAAUGGCGAUCCGGCC ((((((((((..(.((.(((((.(((((((..((.((((......))))))..))).)))).))))).)).)..))))((((......))))......)))))).(((......))) ( -45.80) >consensus AUCAGAGAAGAUGCGCAGGAAUCGGUGGCCCCCCACAUCGUUGUAGGUGGCCAGGCGCACAAAUUCCAGCACCACUUCCGGACUGACCUCCGUGUACAGCUGAAUGAAGAUCCGGCC ...........(((((.........(((((.((((((....)))))).))))).))))).......((((...((...((((......)))).))...))))............... (-16.54 = -18.18 + 1.64)


| Location | 4,647,241 – 4,647,358 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 77.15 |
| Mean single sequence MFE | -48.37 |
| Consensus MFE | -21.93 |
| Energy contribution | -22.30 |
| Covariance contribution | 0.37 |
| Combinations/Pair | 1.41 |
| Mean z-score | -2.89 |
| Structure conservation index | 0.45 |
| SVM decision value | 1.84 |
| SVM RNA-class probability | 0.979649 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 4647241 117 - 23771897 GGCAGAUUACUCCUACAACUCUACACGGAACUGAGUCCGGAAGUGGUGCUGGAGUUUGUGCGUAUGGCCACCCACAACGAUGUAGGAUGCCAUCGGUUUGUGCGAAUAUUCUCCAAU ((.(((..(((((..((...((((.((((......))))...))))))..)))))(((..((.(((((..((.(((....))).))..))))).....))..)))....)))))... ( -32.30) >DroVir_CAF1 34629 117 - 1 GGCCGCAUCUGCAUUCAGCUCUACACAGAGGUCAGUCCGGAGGUGGUGCUGGAAUUUGUACGCCUGGCCAUCGACAACAAUGUCGAGGCGCACAAAUUCCCGCGCAUCUUUCCUGAU ((((((....)).......(((....))))))).(((.(((((..((((.(((((((((.((((......((((((....)))))))))).))))))))).))))...))))).))) ( -45.60) >DroPse_CAF1 24725 117 - 1 GGCCGGAUCGUCAUUCAGCUGUACACGGAGGUCAGUCCGGAAGUGGUGCUGGAAUUGGUGCGCCUUGCCACCUACAACGAUGUGCGGGGCCACCGAUUCCUGCGCAUCUUCGCUGAA (((......))).((((((......((((......))))((((..((((.((((((((((.(((((((((.(......).)).))))))))))))))))).))))..)))))))))) ( -54.70) >DroEre_CAF1 19889 117 - 1 GGCAGAUUACUGCUGCAGCUCUACACGGAACUCAGUCCGGAAGUGGUGCUGCAAUUCGUGCGUAUGGCCACCCACAACGAUGUAGAAUGCCAUCGUUUUCUGCGCAUAUUUUCCAAU (((((....)))))(((((.((((.((((......))))...)))).))))).....((((((((((....))).(((((((........)))))))...))))))).......... ( -41.20) >DroMoj_CAF1 29966 117 - 1 GGCCGCAUUUGCAUUCAGCUGUACACCGAGGUCAGUCCGGAAGUUGUGCUGGAAUUUGUGCGCCUGGCCACCGAGAACGAUGUCAAGGCGCACAAAUUCCCGCACAUCUUCCCGGAC ((((.....((((......))))......)))).(((((((((.(((((.((((((((((((((((((...((....))..))).))))))))))))))).))))).))).)))))) ( -59.00) >DroPer_CAF1 24897 117 - 1 GGCCGGAUCGCCAUUCAGCUGUACACGGAGGUCAGUCCGGAAGUGGUGCUGGAAUUGGUGCGCCUUGCCACCUACAACGAUGUGCGGGGCCACCGAUUCCUGCGCAUCUUCGCUGAA (((......))).((((((......((((......))))((((..((((.((((((((((.(((((((((.(......).)).))))))))))))))))).))))..)))))))))) ( -57.40) >consensus GGCCGAAUCCUCAUUCAGCUCUACACGGAGGUCAGUCCGGAAGUGGUGCUGGAAUUUGUGCGCCUGGCCACCCACAACGAUGUAGAGGGCCACCGAUUCCUGCGCAUCUUCCCUGAU .........................((((......))))((((..((((.((((((((((.((((...((.(......).))....)))))))))))))).))))..))))...... (-21.93 = -22.30 + 0.37)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:11:19 2006