
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 4,567,478 – 4,567,638 |
| Length | 160 |
| Max. P | 0.981852 |

| Location | 4,567,478 – 4,567,598 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.67 |
| Mean single sequence MFE | -34.87 |
| Consensus MFE | -31.58 |
| Energy contribution | -31.63 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.87 |
| Structure conservation index | 0.91 |
| SVM decision value | 1.90 |
| SVM RNA-class probability | 0.981852 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
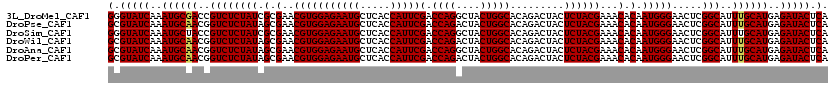
>3L_DroMel_CAF1 4567478 120 + 23771897 GGGUAUCAAAUGCGACCGUCUCUAUCGCGAACGUGGAGAAUGCUCACCAUUCGACCAGGCUACUGGCACAGACUACUCUACGAAACACAAUGGGAACUCGGCAUUUGCAUGAGAUACUCA (((((((..(((((((((((((.........(((((((((((.....)))))(.((((....))))).........)))))).........))))...)))...))))))..))))))). ( -37.77) >DroPse_CAF1 159651 120 + 1 GCGUAUCAAAUGCAACGGUCUCUAUAGCGAACGUGGAGAAUGCUCACCAUUCGACCAGACUACUGGCACAGACUACUCUACGAAACACAAUGGGAACUCGGCAUUUGCAUGAGAUACUCA (.(((((..((((((((((..((((.(.(..(((((((((((.....)))))(.((((....))))).........))))))...).).))))..)).))....))))))..))))).). ( -33.60) >DroSim_CAF1 144646 120 + 1 GGGUAUCAAAUGCUACCGUCUCUAUCGCGAACGUGGAGAAUGCUCACCAUUCGACCAGGCUACUGGCACAGACUACUCUACGAAACACAAUGGGAACUCGGCAUUUGCAUGAGAUACUCA (((((((..((((..(((((((.........(((((((((((.....)))))(.((((....))))).........)))))).........))))...))).....))))..))))))). ( -35.77) >DroWil_CAF1 128836 120 + 1 GCGUAUCAAAUGCAACGGUCUCUAUAGCGAACGUGGAGAAUGCUCACCAUUCGACCAGACUACUGGCACAGACUACUCUACGAAACACAAUGGGAACUCGGCAUUUGCAUGAGAUACUCA (.(((((..((((((((((..((((.(.(..(((((((((((.....)))))(.((((....))))).........))))))...).).))))..)).))....))))))..))))).). ( -33.60) >DroAna_CAF1 147203 120 + 1 GCGUAUCAAAUGCAACGGUCUCUAUAGCGAACGUGGAGAAUGCUCACCAUUCGACCAGGCUACUGGCACAGACUACUCUACGAAACACAAUGGGAACUCGGCAUUUGCAUGAGAUACUCA (.(((((..((((((((((..((((.(.(..(((((((((((.....)))))(.((((....))))).........))))))...).).))))..)).))....))))))..))))).). ( -34.90) >DroPer_CAF1 161027 120 + 1 GCGUAUCAAAUGCAACGGUCUCUAUAGCGAACGUGGAGAAUGCUCACCAUUCGACCAGACUACUGGCACAGACUACUCUACGAAACACAAUGGGAACUCGGCAUUUGCAUGAGAUACUCA (.(((((..((((((((((..((((.(.(..(((((((((((.....)))))(.((((....))))).........))))))...).).))))..)).))....))))))..))))).). ( -33.60) >consensus GCGUAUCAAAUGCAACGGUCUCUAUAGCGAACGUGGAGAAUGCUCACCAUUCGACCAGACUACUGGCACAGACUACUCUACGAAACACAAUGGGAACUCGGCAUUUGCAUGAGAUACUCA (.(((((..((((((..((((((((.(.(..(((((((((((.....)))))(.((((....))))).........))))))...).).))))).....)))..))))))..))))).). (-31.58 = -31.63 + 0.06)

| Location | 4,567,518 – 4,567,638 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 98.44 |
| Mean single sequence MFE | -35.22 |
| Consensus MFE | -35.12 |
| Energy contribution | -35.12 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.15 |
| Structure conservation index | 1.00 |
| SVM decision value | 1.78 |
| SVM RNA-class probability | 0.976681 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
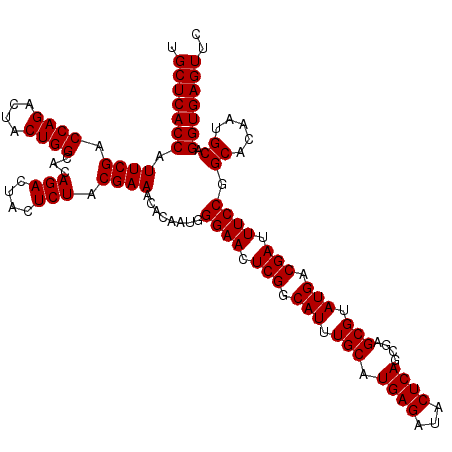
>3L_DroMel_CAF1 4567518 120 + 23771897 UGCUCACCAUUCGACCAGGCUACUGGCACAGACUACUCUACGAAACACAAUGGGAACUCGGCAUUUGCAUGAGAUACUCAGCGAGCGUAUGACGAUUUCCGGCACAAUGCAGGUGAGUUC .(((((((.((((.((((....))))...(((....))).))))........((((.(((.(((.(((.((((...))))....))).))).))).)))).((.....)).))))))).. ( -36.00) >DroVir_CAF1 167598 119 + 1 UGCUCACCAUUCGACCAGACUACUGGCACAGACUACUCUACGAAACACAAUGGGAACUCGGCAUUUGCAUGAGAUACUCAGCGAGCGUAUGACGAUUUCCGGCACCAUGCAGGUGAGUG- .(((((((.((((.((((....))))...(((....))).))))........((((.(((.(((.(((.((((...))))....))).))).))).)))).((.....)).))))))).- ( -35.30) >DroGri_CAF1 164329 120 + 1 UGCUCACCAUUCGACCAGACUACUGGCACAGACUACUCUACGAAACACAAUGGGAACUCGGCAUUUGCAUGAGAUACUCAGCGAGCGUAUGACGAUUUCCGGCACCAUGCAGGUGAGUUC .(((((((.((((.((((....))))...(((....))).))))........((((.(((.(((.(((.((((...))))....))).))).))).)))).((.....)).))))))).. ( -34.70) >DroWil_CAF1 128876 120 + 1 UGCUCACCAUUCGACCAGACUACUGGCACAGACUACUCUACGAAACACAAUGGGAACUCGGCAUUUGCAUGAGAUACUCAGCGAGCGUAUGACGAUUUCCGGCACAAUGCAGGUGAGUUC .(((((((.((((.((((....))))...(((....))).))))........((((.(((.(((.(((.((((...))))....))).))).))).)))).((.....)).))))))).. ( -34.70) >DroMoj_CAF1 200728 120 + 1 UGCUCACCAUUCGACCAGACUACUGGCACAGACUACUCUACGAAACACAAUGGGAACUCGGCAUUUGCAUGAGAUACUCAGCGAGCGUAUGACGAUUUCCGGCACCAUGCAGGUGAGUAC ((((((((.((((.((((....))))...(((....))).))))........((((.(((.(((.(((.((((...))))....))).))).))).)))).((.....)).)))))))). ( -35.90) >DroPer_CAF1 161067 120 + 1 UGCUCACCAUUCGACCAGACUACUGGCACAGACUACUCUACGAAACACAAUGGGAACUCGGCAUUUGCAUGAGAUACUCAGCGAGCGUAUGACGAUUUCCGGCACAAUGCAGGUGAGUUC .(((((((.((((.((((....))))...(((....))).))))........((((.(((.(((.(((.((((...))))....))).))).))).)))).((.....)).))))))).. ( -34.70) >consensus UGCUCACCAUUCGACCAGACUACUGGCACAGACUACUCUACGAAACACAAUGGGAACUCGGCAUUUGCAUGAGAUACUCAGCGAGCGUAUGACGAUUUCCGGCACAAUGCAGGUGAGUUC .(((((((.((((.((((....))))...(((....))).))))........((((.(((.(((.(((.((((...))))....))).))).))).)))).((.....)).))))))).. (-35.12 = -35.12 + 0.00)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:10:48 2006