
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 4,024,985 – 4,025,105 |
| Length | 120 |
| Max. P | 0.680724 |

| Location | 4,024,985 – 4,025,105 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.33 |
| Mean single sequence MFE | -37.56 |
| Consensus MFE | -25.34 |
| Energy contribution | -24.68 |
| Covariance contribution | -0.66 |
| Combinations/Pair | 1.38 |
| Mean z-score | -1.92 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.29 |
| SVM RNA-class probability | 0.671570 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
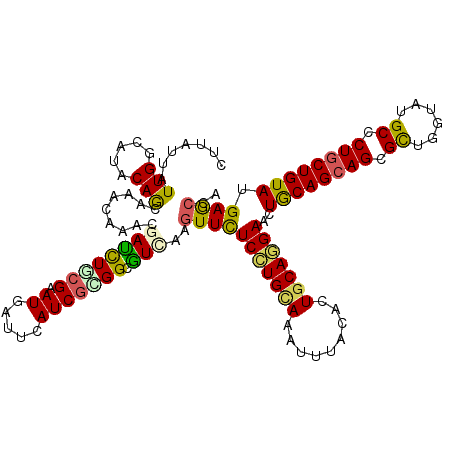
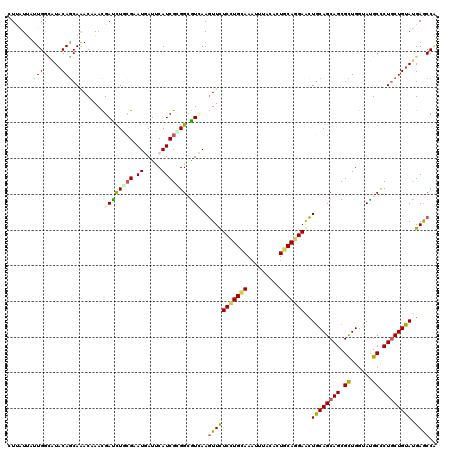
>3L_DroMel_CAF1 4024985 120 + 23771897 CUCAUAAUUGGCAUCCAGCAGACCAACGAUCUGCGGAUAAUCCAUCGCGGCGUUAAGUUUUCCUGCAAGUUUACUCUGCAGGAACUACAGCAGCGCUGGUAUGCUCUGCUGUAUGAGCCU ........(((.((((.(((((.......)))))))))...)))....(((........((((((((((....)).)))))))).((((((((.((......)).))))))))...))). ( -44.90) >DroVir_CAF1 5044 120 + 1 CUGGUUAUUGGCAUACAGCAAACAAACGAUCUACGCAUGAUUCAUCGCGGCAUCAAGUUCUCCUGCAAAUUUACCCUGCAGGAGCUGCAGCAGCGCUGGUAUGCCCUACUGUAUGAACCA ..((((...((((((((((........(((...((((((...))).)))..)))..((.((((((((.........))))))))..))......))).)))))))...(.....))))). ( -36.60) >DroGri_CAF1 4806 120 + 1 CUCGUUAUUGGCAUACAGCAAACAAACGAUUUGCGAAUGAUUCAUCGUGGCGUCAAGUUCUCCUGUAAAUUUACGCUGCAGGAGCUGCAGCAGCGUUGGUACGCGCUGCUGUACGAACCA .((((..(((((.(((.((((((....).)))))((.....))...)))..)))))...((((((((.........))))))))..((((((((((......)))))))))))))).... ( -39.40) >DroWil_CAF1 4386 120 + 1 CUUAUUAUUGGUGUACAACAAACAAAUGAUUUGCGUAUGAUUCAUCGGGGAGUGAAGUUCUCUUGCAAGUUUACAUUGCAAGAACUGCAGCAGCGUUGGUAUGCCCUGCUGUACGAGCCA .(((((.((((((.........(((.....))).........))))))..))))).((((((((((((.......))))))))..((((((((.((......)).)))))))).)))).. ( -30.67) >DroMoj_CAF1 5546 120 + 1 CUUGUUAUUGGUAUACAGCAAACCAACGACCUCCGCAUGAUACAUCGGGGCGUCAAAUUCUCCUGUAAAUUUACUCUGCAAGAGUUGCAGCAGCGCUGGUACGCCCUGCUGUAUGAGCCA ...((..(((((.........)))))..))....((.(.(((((.(((((((((.....((.(((((.....((((.....))))))))).)).....).)))))))).))))).))).. ( -36.50) >DroAna_CAF1 5664 120 + 1 CUUAUAAUUGGAAUACAGCAGACCAACGACUUGAGAAUCAUCCAUCGCGGAGUGAAGUUCUCCUGCAAGUUUACAAUUCAGGAACUGCAGCAGCGCUGGUAUGCUCUGCUGUAUGAGCCG .........((.(((((((((((((.((.((.(((((((((((.....))).)))..)))))(((((.((((.........))))))))).)))).))).....))))))))))...)). ( -37.30) >consensus CUUAUUAUUGGCAUACAGCAAACAAACGAUCUGCGAAUGAUUCAUCGCGGCGUCAAGUUCUCCUGCAAAUUUACACUGCAGGAACUGCAGCAGCGCUGGUAUGCCCUGCUGUAUGAGCCA .......(((.....))).........((((((((.((.....))))))).)))..(((((((((((.........)))))))..((((((((.((......)).)))))))).)))).. (-25.34 = -24.68 + -0.66)

| Location | 4,024,985 – 4,025,105 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.33 |
| Mean single sequence MFE | -39.61 |
| Consensus MFE | -24.36 |
| Energy contribution | -23.75 |
| Covariance contribution | -0.60 |
| Combinations/Pair | 1.52 |
| Mean z-score | -2.29 |
| Structure conservation index | 0.61 |
| SVM decision value | 0.31 |
| SVM RNA-class probability | 0.680724 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
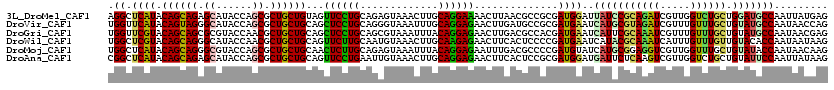
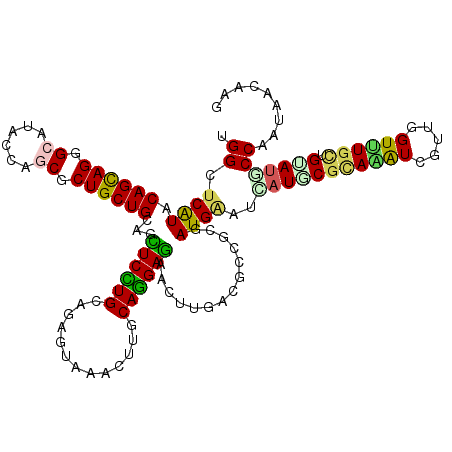
>3L_DroMel_CAF1 4024985 120 - 23771897 AGGCUCAUACAGCAGAGCAUACCAGCGCUGCUGUAGUUCCUGCAGAGUAAACUUGCAGGAAAACUUAACGCCGCGAUGGAUUAUCCGCAGAUCGUUGGUCUGCUGGAUGCCAAUUAUGAG .(((...((((((((.((......)).)))))))).((((((((.((....))))))))))........)))....(((..(((((((((((.....)))))).))))))))........ ( -49.90) >DroVir_CAF1 5044 120 - 1 UGGUUCAUACAGUAGGGCAUACCAGCGCUGCUGCAGCUCCUGCAGGGUAAAUUUGCAGGAGAACUUGAUGCCGCGAUGAAUCAUGCGUAGAUCGUUUGUUUGCUGUAUGCCAAUAACCAG ((((((((((((((((((((..((((...))))...((((((((((.....))))))))))......)))))(((((.............)))))....)))))))))).....))))). ( -42.82) >DroGri_CAF1 4806 120 - 1 UGGUUCGUACAGCAGCGCGUACCAACGCUGCUGCAGCUCCUGCAGCGUAAAUUUACAGGAGAACUUGACGCCACGAUGAAUCAUUCGCAAAUCGUUUGUUUGCUGUAUGCCAAUAACGAG ((((..((((((((((((((....(((((((..........))))))).......((((....)))))))).(((((.............)))))..).)))))))))))))........ ( -36.62) >DroWil_CAF1 4386 120 - 1 UGGCUCGUACAGCAGGGCAUACCAACGCUGCUGCAGUUCUUGCAAUGUAAACUUGCAAGAGAACUUCACUCCCCGAUGAAUCAUACGCAAAUCAUUUGUUUGUUGUACACCAAUAAUAAG (((...(((((((((((.........((....))..(((((((((.(....))))))))))..........)))(((((............)))))....)))))))).)))........ ( -28.30) >DroMoj_CAF1 5546 120 - 1 UGGCUCAUACAGCAGGGCGUACCAGCGCUGCUGCAACUCUUGCAGAGUAAAUUUACAGGAGAAUUUGACGCCCCGAUGUAUCAUGCGGAGGUCGUUGGUUUGCUGUAUACCAAUAACAAG (((...(((((((((.....(((((((((.(((((.((((((.(((.....))).))))))....(((..(......)..)))))))).)).)))))))))))))))).)))........ ( -39.60) >DroAna_CAF1 5664 120 - 1 CGGCUCAUACAGCAGAGCAUACCAGCGCUGCUGCAGUUCCUGAAUUGUAAACUUGCAGGAGAACUUCACUCCGCGAUGGAUGAUUCUCAAGUCGUUGGUCUGCUGUAUUCCAAUUAUAAG .((...((((((((((.....(((((((((((((((((....))))))).....))))(((((..(((.(((.....)))))))))))....)))))))))))))))).))......... ( -40.40) >consensus UGGCUCAUACAGCAGGGCAUACCAGCGCUGCUGCAGCUCCUGCAGAGUAAACUUGCAGGAGAACUUGACGCCGCGAUGAAUCAUGCGCAAAUCGUUGGUUUGCUGUAUGCCAAUAACAAG .((.((((.((((((.((......)).))))))...((((((.............))))))..............))))..(((((((((((.....)))))).)))))))......... (-24.36 = -23.75 + -0.60)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:06:12 2006