
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 4,020,961 – 4,021,072 |
| Length | 111 |
| Max. P | 0.628772 |

| Location | 4,020,961 – 4,021,072 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 113 |
| Reading direction | forward |
| Mean pairwise identity | 76.27 |
| Mean single sequence MFE | -30.77 |
| Consensus MFE | -21.35 |
| Energy contribution | -19.72 |
| Covariance contribution | -1.63 |
| Combinations/Pair | 1.43 |
| Mean z-score | -1.07 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.20 |
| SVM RNA-class probability | 0.628772 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 4020961 111 + 23771897 GAUAUCCACUUA-CUUAGGUGGCUGGAACCAUCGUCACGUACGAACUUGUUCUCAUCCAAUUUCAUGAGGACAACGACCUGUGGGACUGCGAUCAGAGCUAUUACUCAUAGG- ....((((.(((-(....)))).))))....((((.....))))..((((((((((........))))))))))...((((((((...((.......)).....))))))))- ( -30.00) >DroPse_CAF1 519 111 + 1 AAUUUUUCUUUG-UGAAGGUGGCAGGCACCAUUGUUACCUAUGAGCUGGUGCUUAUCCAGUUCCACGAGGAUAACGAUUUGUGGGAUUGCAAUCAGAGCUAUUACUCUUAAG- ......((((((-((.(((((((((......)))))))))..(((((((.......)))))))))))))))....((((.((......))))))((((......))))....- ( -33.60) >DroYak_CAF1 440 107 + 1 UGUAUCCAU-----UUAGGUUGCUGGAACCAUCGUCACCUACGAACUGGUGCUCAUCCAAUUUCACGAGGACAACGACCUGUGGGACUGCGAUCAGAGCUAUUACUCAUAGG- .((((((..-----.(((((((.((...((.((((((((........)))).............)))))).)).)))))))..))).))).....(((......))).....- ( -26.71) >DroMoj_CAF1 1312 112 + 1 GAUAUGUUCUU-CAUCAGGUGGCUGGCACCAUCGUCACAUAUGAGUUGGUGCUGAUUCAGUUCCACGAGGAUCAGGAUCUGUGGACUUGCGACCAGAGCACCUAUUCGUAGAA ...........-..((..(((((((....))..))))).(((((((.(((((((((((...(((....)))...)))))..(((........))).)))))).))))))))). ( -32.00) >DroAna_CAF1 521 107 + 1 UAUAUCAUA-----ACAGGUUGCUGGUACUAUUGUAACAUAUGAGCUGGUGCUCAUCCAAUUCCACGAGGACAACGACCUGUGGGACUGCGAUCAGAGCUACUACUCCUAGU- .........-----.......((((((((....)))......(((.(((((((((((((.((((((.(((.......))))))))).)).)))..)))).))))))))))))- ( -28.70) >DroPer_CAF1 519 111 + 1 AAUUUUUCUUUG-UGAAGGUGGCAGGCACCAUUGUUACCUAUGAGCUGGUGCUUAUCCAGUUCCACGAGGAUAACGAUUUGUGGGAUUGCAAUCAGAGCUAUUACUCUUAAG- ......((((((-((.(((((((((......)))))))))..(((((((.......)))))))))))))))....((((.((......))))))((((......))))....- ( -33.60) >consensus AAUAUCUAUUU___UAAGGUGGCUGGCACCAUCGUCACCUAUGAGCUGGUGCUCAUCCAAUUCCACGAGGACAACGACCUGUGGGACUGCGAUCAGAGCUAUUACUCAUAGG_ .................(((.((.(((((((((((.....))))..))))))).......((((((.(((.......)))))))))..)).))).(((......)))...... (-21.35 = -19.72 + -1.63)

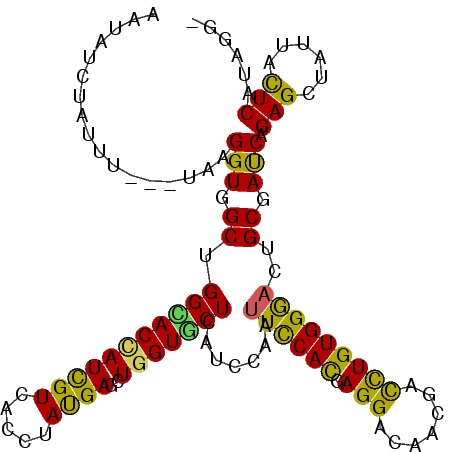
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:06:05 2006