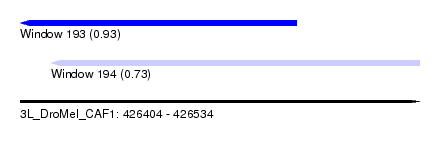
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 426,404 – 426,534 |
| Length | 130 |
| Max. P | 0.930103 |

| Location | 426,404 – 426,494 |
|---|---|
| Length | 90 |
| Sequences | 6 |
| Columns | 101 |
| Reading direction | reverse |
| Mean pairwise identity | 72.38 |
| Mean single sequence MFE | -41.40 |
| Consensus MFE | -19.39 |
| Energy contribution | -19.17 |
| Covariance contribution | -0.22 |
| Combinations/Pair | 1.26 |
| Mean z-score | -2.06 |
| Structure conservation index | 0.47 |
| SVM decision value | 1.20 |
| SVM RNA-class probability | 0.930103 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 426404 90 - 23771897 ACGCUGAGGUCGCUCUACGAGCUGGUAGACCGGCCCGUGGGUGGACGCCUCGGUCGGUUCUUUGACGGGCUGCGGGGACUGUUUUAAACA----------- .((((((((((((.(((((.(((((....))))).))))))))...))))))).))(((....(((((.((....)).)))))...))).----------- ( -36.00) >DroVir_CAF1 30061 96 - 1 ACGCUGCGGUCGCUCUAUGCGCUGGUCAAUCGGCCCAUGG---GGCGGCUGGAGCGUCUCUUUGCCGGCGUUCGGGGUUUGCUCUGAAAC--AGUGCAACA (((((.(((((((((((((.(((((....))))).)))))---)))))))).)))))....(((((....(((((((....)))))))..--.).)))).. ( -48.40) >DroPse_CAF1 59134 87 - 1 ACUCUCAGGUCGCUCUAUGCGCUGGUCAACCGGCCCAUGG---GGCCUCUCGAGCGGCUCUUUGGGGGCCUUAGGGGUCUGCUAUGAGCA----------- .(.(((..(..((((((((.(((((....))))).)))))---)))..)..))).)((((...(.(((((.....))))).)...)))).----------- ( -42.40) >DroYak_CAF1 7839 90 - 1 ACGCUGAGGUCGCUCUACGAGCUGGUAGACCGGCCCGUGGGCGGACGCCUCGGUCGGUUCUUUGUCGGCUUGCGGGGACUGUUUUAAACA----------- ..(((((((((((.(((((.(((((....))))).))))))))...))))))))((((.((((((......)))))))))).........----------- ( -38.30) >DroMoj_CAF1 15366 98 - 1 ACGCUGCGGACGCUCUAUGCGCUGGUCAAUCGGCCCAUGG---GGCGGCUGGAGCGUCUCUUCGCUGGGUUUCGGGGGCUGCUCUAAGAGCUAAGAAAUGA (((((.(((.(((((((((.(((((....))))).)))))---)))).))).)))))...(((..(((.(((..(((....)))..))).))).))).... ( -40.90) >DroPer_CAF1 56731 87 - 1 ACUCUCAGGUCGCUCUAUGCGCUGGUCAACCGGCCCAUGG---GGCCUCUCGAGCGGCUCUUUGGGGGCCUUAGGGGUCUGCUAUGAGCA----------- .(.(((..(..((((((((.(((((....))))).)))))---)))..)..))).)((((...(.(((((.....))))).)...)))).----------- ( -42.40) >consensus ACGCUGAGGUCGCUCUAUGCGCUGGUCAACCGGCCCAUGG___GGCGCCUCGAGCGGCUCUUUGCCGGCCUUCGGGGUCUGCUAUAAACA___________ ........(((...(((((.(((((....))))).)))))...))).....((((((((((............)))).))))))................. (-19.39 = -19.17 + -0.22)

| Location | 426,414 – 426,534 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.92 |
| Mean single sequence MFE | -56.07 |
| Consensus MFE | -30.79 |
| Energy contribution | -31.38 |
| Covariance contribution | 0.59 |
| Combinations/Pair | 1.29 |
| Mean z-score | -2.65 |
| Structure conservation index | 0.55 |
| SVM decision value | 0.43 |
| SVM RNA-class probability | 0.732153 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 426414 120 - 23771897 UAUGCUGGAGCACUUUGCGCUCUACCGAUUCCUGGCGGGAACGCUGAGGUCGCUCUACGAGCUGGUAGACCGGCCCGUGGGUGGACGCCUCGGUCGGUUCUUUGACGGGCUGCGGGGACU .....((((((.......))))))(((...((((..(((((((((((((((((.(((((.(((((....))))).))))))))...))))))))..))))))...))))...)))..... ( -52.90) >DroVir_CAF1 30080 117 - 1 UAUGCUGGAGCACUUUACGCUCUAUAGAUUCCUCGCCGGGACGCUGCGGUCGCUCUAUGCGCUGGUCAAUCGGCCCAUGG---GGCGGCUGGAGCGUCUCUUUGCCGGCGUUCGGGGUUU .....((((((.......)))))).(((((((.((((((((((((.(((((((((((((.(((((....))))).)))))---)))))))).))))))......))))))...))))))) ( -62.80) >DroPse_CAF1 59144 117 - 1 UCUGCUGGAGCACUUUGCGCUCUACAGGUUCCUGGCCGGGACUCUCAGGUCGCUCUAUGCGCUGGUCAACCGGCCCAUGG---GGCCUCUCGAGCGGCUCUUUGGGGGCCUUAGGGGUCU ((((.((((((.......))))))))))....(((((((((((....))))((.....)).)))))))...(((((.(((---(((((((.(((.....))).)))))))))).))))). ( -52.70) >DroGri_CAF1 15321 117 - 1 UCUGCUGGAGCAUUUUGCGCUCUAUAGAUUCCUGGCCGGCACGUUGCGGUCGCUUUAUGCGCUCGUCAAUCGGCCCAUGG---GGCGGCUGGAGCGUUUAUUUGCCGGCUUUCGAGGGUU ((((.((((((.......)))))))))).((((((((((((((((.(((((((((((((.((.((.....)))).)))))---)))))))).)))))......)))))))....)))).. ( -55.40) >DroMoj_CAF1 15387 117 - 1 UUUGCUGGAGCACUUCACGCUCUAUCGAUUUCUGGCCGGGACGCUGCGGACGCUCUAUGCGCUGGUCAAUCGGCCCAUGG---GGCGGCUGGAGCGUCUCUUCGCUGGGUUUCGGGGGCU ...((((((((.......))))).((((..((..((.((((((((.(((.(((((((((.(((((....))))).)))))---)))).))).))))))))...))..))..)))).))). ( -59.90) >DroPer_CAF1 56741 117 - 1 UCUGCUGGAGCACUUUGCGCUCUACAGGUUCCUGGCCGGGACUCUCAGGUCGCUCUAUGCGCUGGUCAACCGGCCCAUGG---GGCCUCUCGAGCGGCUCUUUGGGGGCCUUAGGGGUCU ((((.((((((.......))))))))))....(((((((((((....))))((.....)).)))))))...(((((.(((---(((((((.(((.....))).)))))))))).))))). ( -52.70) >consensus UCUGCUGGAGCACUUUGCGCUCUACAGAUUCCUGGCCGGGACGCUGAGGUCGCUCUAUGCGCUGGUCAACCGGCCCAUGG___GGCGGCUCGAGCGGCUCUUUGCCGGCCUUCGGGGUCU ((((.((((((.......))))))))))((((.(((((((.((((..((.(((((((((.(((((....))))).)))))...)))).))..)))).)))......))))...))))... (-30.79 = -31.38 + 0.59)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:33:33 2006