
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 3,860,530 – 3,860,643 |
| Length | 113 |
| Max. P | 0.971114 |

| Location | 3,860,530 – 3,860,631 |
|---|---|
| Length | 101 |
| Sequences | 6 |
| Columns | 101 |
| Reading direction | forward |
| Mean pairwise identity | 84.95 |
| Mean single sequence MFE | -22.32 |
| Consensus MFE | -15.79 |
| Energy contribution | -17.18 |
| Covariance contribution | 1.39 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.45 |
| Structure conservation index | 0.71 |
| SVM decision value | 0.08 |
| SVM RNA-class probability | 0.572448 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 3860530 101 + 23771897 ACAAACGCACCCGUGCACUGCGUGUAUCUAUGUCGUUGUAUCUGUGUUUGUAUAUCAUUACUUAUCAGAUACUUAUUAUGAGCAUACGAUAUCGUAUAUUU ......(((....)))..((((.(((((((((((((.(((((((.((..(((......)))..))))))))).....)))).)))).)))))))))..... ( -25.70) >DroGri_CAF1 66297 89 + 1 ----------ACAUGCUCUGAGUAUAUGUGAGUA--UCUGUGUGUGUUUGUAUAUCAUUACUUAUCAGAUACUUAUUAUGAGCACACGAUAUCGUAUAUUU ----------...(((((.........(((((((--((((...(((..((.....)).)))....)))))))))))...))))).(((....)))...... ( -21.10) >DroSec_CAF1 59781 96 + 1 -----GGCAACCGUGCACUGCGUGUAUCUAUGUCGUUGUAUCUGUGUUUGUAUAUCAUUACUUAUCAGAUACUUAUUAUGAGCAUACGAUAUCGUAUAUUU -----.(((....)))..((((.(((((((((((((.(((((((.((..(((......)))..))))))))).....)))).)))).)))))))))..... ( -24.20) >DroSim_CAF1 60398 96 + 1 -----CGCACCCGUGCACUGCGUGUAUCUAUGUCGUUGUAUCUGUGUUUGUAUAUCAUUACUUAUCAGAUACUUAUUAUGAGCAUACGAUAUCGUAUAUUU -----.(((....)))..((((.(((((((((((((.(((((((.((..(((......)))..))))))))).....)))).)))).)))))))))..... ( -25.70) >DroEre_CAF1 62045 83 + 1 A------------------GCGUGUAUCUAUGUCGUUGUAUCUGUGUUUGUAUAUCAUUACUUAUCAGAUACUUAUUAUGAGCACACGAUAUCGUAUAUUU .------------------.(((((.((.(((.....(((((((.((..(((......)))..)))))))))....)))))..)))))............. ( -17.90) >DroYak_CAF1 62092 99 + 1 A--GUCAUACACACACACUGCGUGUAUCUAUGUCGUUGUAUCUGUGUUUGUAUAUCAUUACUUAUCAGAUACUUAUUAUGAGCACACGAUAUCGUAUAUUU .--...((((((.(.....).)))))).((((.((..(((((((((((..((((((...........)))).....))..)))))).)))))))))))... ( -19.30) >consensus _____CGCACCCGUGCACUGCGUGUAUCUAUGUCGUUGUAUCUGUGUUUGUAUAUCAUUACUUAUCAGAUACUUAUUAUGAGCACACGAUAUCGUAUAUUU ..................((((.(((((((((((((.(((((((.((..(((......)))..))))))))).....)))).)))).)))))))))..... (-15.79 = -17.18 + 1.39)



| Location | 3,860,553 – 3,860,643 |
|---|---|
| Length | 90 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 79.33 |
| Mean single sequence MFE | -22.88 |
| Consensus MFE | -11.59 |
| Energy contribution | -11.98 |
| Covariance contribution | 0.39 |
| Combinations/Pair | 1.09 |
| Mean z-score | -2.01 |
| Structure conservation index | 0.51 |
| SVM decision value | 0.05 |
| SVM RNA-class probability | 0.559097 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 3860553 90 + 23771897 GUAUCUAUGUC-GUUGUAUCUGUGUUUGUAUAUCAUUACUUAUCAGAUACUUAUUAUGAGCAUACGAUAUCGUAUAUU-UUGGCAUGCUC----------------------------CU .....((((((-(..(((((((.((..(((......)))..)))))))))...........(((((....)))))...-.)))))))...----------------------------.. ( -19.80) >DroPse_CAF1 68818 119 + 1 GUAUCUAUAGCUUUUGUAUCUGUGUUUGUAUAUCAUUACUUAUCAGAUACUUAUUAUGAGCAUACGAUAUCGUAUAUU-UUGGCAUGCCCACAGAUACAGAUACAGAUGCAGAAUGGGCU ........(((((((((((((((((((((((..............................(((((....)))))...-..((....)).....)))))))))))))))))))...)))) ( -32.40) >DroGri_CAF1 66310 88 + 1 AUAUGUGAGUA---UCUGUGUGUGUUUGUAUAUCAUUACUUAUCAGAUACUUAUUAUGAGCACACGAUAUCGUAUAUU-UUGGCAUGCGC----------------------------GU .((((((((((---((((...(((..((.....)).)))....)))))))))).)))).((.((.(((((...)))))-.))))......----------------------------.. ( -17.90) >DroEre_CAF1 62050 90 + 1 GUAUCUAUGUC-GUUGUAUCUGUGUUUGUAUAUCAUUACUUAUCAGAUACUUAUUAUGAGCACACGAUAUCGUAUAUU-UUGGCAUGCUC----------------------------CU .....((((((-(..(((((((.((..(((......)))..))))))))).............(((....))).....-.)))))))...----------------------------.. ( -17.40) >DroYak_CAF1 62113 90 + 1 GUAUCUAUGUC-GUUGUAUCUGUGUUUGUAUAUCAUUACUUAUCAGAUACUUAUUAUGAGCACACGAUAUCGUAUAUU-UUGGCAUGCUC----------------------------CU .....((((((-(..(((((((.((..(((......)))..))))))))).............(((....))).....-.)))))))...----------------------------.. ( -17.40) >DroPer_CAF1 69340 120 + 1 GUAUCUAUAGCUUUUGUAUCUGUGUUUGUAUAUCAUUACUUAUCAGAUACUUAUUAUGAGCAUACGAUAUCGUAUAUUUUUGGCAUGCCCACAGAUACAGAUACAGAUGCAGAAUGGGCU ........(((((((((((((((((((((((..............................(((((....)))))......((....)).....)))))))))))))))))))...)))) ( -32.40) >consensus GUAUCUAUGUC_GUUGUAUCUGUGUUUGUAUAUCAUUACUUAUCAGAUACUUAUUAUGAGCACACGAUAUCGUAUAUU_UUGGCAUGCUC____________________________CU (((((((........(((((((.((..(((......)))..))))))))).............(((....))).......))).))))................................ (-11.59 = -11.98 + 0.39)



| Location | 3,860,553 – 3,860,643 |
|---|---|
| Length | 90 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.33 |
| Mean single sequence MFE | -21.43 |
| Consensus MFE | -15.01 |
| Energy contribution | -15.26 |
| Covariance contribution | 0.25 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.83 |
| Structure conservation index | 0.70 |
| SVM decision value | 1.67 |
| SVM RNA-class probability | 0.971114 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 3860553 90 - 23771897 AG----------------------------GAGCAUGCCAA-AAUAUACGAUAUCGUAUGCUCAUAAUAAGUAUCUGAUAAGUAAUGAUAUACAAACACAGAUACAAC-GACAUAGAUAC ..----------------------------((((((((...-.............)))))))).......(((((((....(((......))).....)))))))...-........... ( -18.79) >DroPse_CAF1 68818 119 - 1 AGCCCAUUCUGCAUCUGUAUCUGUAUCUGUGGGCAUGCCAA-AAUAUACGAUAUCGUAUGCUCAUAAUAAGUAUCUGAUAAGUAAUGAUAUACAAACACAGAUACAAAAGCUAUAGAUAC .((.......))....(((((((((.((((((((((((...-.............)))))))))).....(((((((....(((......))).....)))))))...)).))))))))) ( -29.99) >DroGri_CAF1 66310 88 - 1 AC----------------------------GCGCAUGCCAA-AAUAUACGAUAUCGUGUGCUCAUAAUAAGUAUCUGAUAAGUAAUGAUAUACAAACACACACAGA---UACUCACAUAU ..----------------------------(((((((....-............)))))))........((((((((........((.....))........))))---))))....... ( -14.28) >DroEre_CAF1 62050 90 - 1 AG----------------------------GAGCAUGCCAA-AAUAUACGAUAUCGUGUGCUCAUAAUAAGUAUCUGAUAAGUAAUGAUAUACAAACACAGAUACAAC-GACAUAGAUAC ..----------------------------((((((((...-.............)))))))).......(((((((....(((......))).....)))))))...-........... ( -17.79) >DroYak_CAF1 62113 90 - 1 AG----------------------------GAGCAUGCCAA-AAUAUACGAUAUCGUGUGCUCAUAAUAAGUAUCUGAUAAGUAAUGAUAUACAAACACAGAUACAAC-GACAUAGAUAC ..----------------------------((((((((...-.............)))))))).......(((((((....(((......))).....)))))))...-........... ( -17.79) >DroPer_CAF1 69340 120 - 1 AGCCCAUUCUGCAUCUGUAUCUGUAUCUGUGGGCAUGCCAAAAAUAUACGAUAUCGUAUGCUCAUAAUAAGUAUCUGAUAAGUAAUGAUAUACAAACACAGAUACAAAAGCUAUAGAUAC .((.......))....(((((((((.((((((((((((.................)))))))))).....(((((((....(((......))).....)))))))...)).))))))))) ( -29.93) >consensus AG____________________________GAGCAUGCCAA_AAUAUACGAUAUCGUAUGCUCAUAAUAAGUAUCUGAUAAGUAAUGAUAUACAAACACAGAUACAAC_GACAUAGAUAC ..............................((((((((.................)))))))).......(((((((....(((......))).....)))))))............... (-15.01 = -15.26 + 0.25)
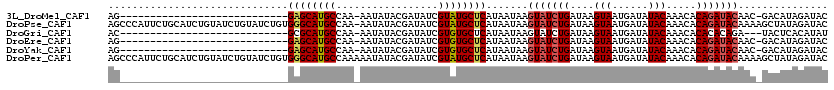

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:04:57 2006