
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 3,822,712 – 3,822,845 |
| Length | 133 |
| Max. P | 0.935480 |

| Location | 3,822,712 – 3,822,808 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | reverse |
| Mean pairwise identity | 75.03 |
| Mean single sequence MFE | -48.98 |
| Consensus MFE | -26.91 |
| Energy contribution | -27.08 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.32 |
| Mean z-score | -2.03 |
| Structure conservation index | 0.55 |
| SVM decision value | 1.24 |
| SVM RNA-class probability | 0.935480 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
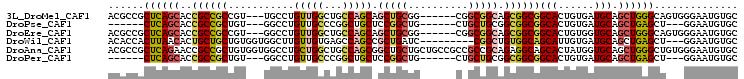
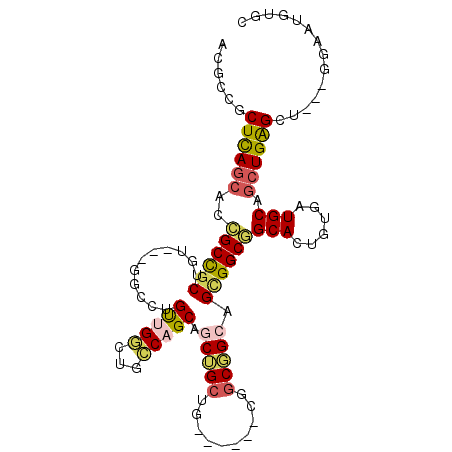
>3L_DroMel_CAF1 3822712 96 - 23771897 ACGCCGCUCAGCACCGCCGCCGU---UGCCUGUUGGCUGCCAGCAGCUGCGG------CGGCGGCAGCGGCGGCACUGUGAUGCAGCUGGGCAGUGGGAAUGUGC .(((.(((((((...((((((((---((((.....((((((.((....))))------))))))))))))))))..((.....))))))))).)))......... ( -56.20) >DroPse_CAF1 24782 87 - 1 ------CUCAGCACCGCCGCUGU---GGCCUGUUGCCCGGCUGCUCCGGCUG------CUGCUGCGGCGGCGGCACUGUGAUGCAGCUGAGCU---GGAAUGUGC ------.(((((.(((((((((.---.((..((.(((.((.....))))).)------).))..)))))))))(((((.....))).)).)))---))....... ( -43.40) >DroEre_CAF1 22830 96 - 1 ACGCCGCUCAGCACCGCCGCCGU---GGCCUGUUGGCUGCCAGCAGCUGCGG------CGGCGGCAGCGGCGGCACUGUGGUGCAGCUGGGCAGUGGGAAUGUGC .(((.(((((((.(((((((((.---.((.((((((...))))))))..)))------))))))........((((....)))).))))))).)))......... ( -58.60) >DroWil_CAF1 32731 93 - 1 ACACCACUUAACACUGCUGCUGUGGUGGCUUGUUGUGAGCCAGCCGCUGAUC---------CGGCUGUGGCAGCAUUGUGAUGCAGCUGAGCU---GGAAUGUGC ...........(((.......((((((((((.....)))))).))))...((---------(((((.((((.((((....)))).))))))))---)))..))). ( -34.30) >DroAna_CAF1 21680 105 - 1 ACGCCGCUCAGAACCGCCGCUGUGGUGGCCUGCUGGCUGCCAGCGGCUGCUGCUGCCGCCGCCGCAGAGGCAGCACUAUGGUGCAGCUGGGCUGUGGGAAUGUGC .(((.((((((....(((.((((((((((..(((((...)))))(((.......))))))))))))).))).((((....))))..)))))).)))......... ( -58.00) >DroPer_CAF1 25116 87 - 1 ------CUCAGCACCGCCGCUGU---GGCCUGUUGCCCGGCUGCUCCGGCUG------CUGCUGCGGCGGCGGCACUGUGAUGCAGCUGAGCU---GGAAUGUGC ------.(((((.(((((((((.---.((..((.(((.((.....))))).)------).))..)))))))))(((((.....))).)).)))---))....... ( -43.40) >consensus ACGCCGCUCAGCACCGCCGCUGU___GGCCUGUUGGCUGCCAGCAGCUGCUG______CGGCGGCAGCGGCGGCACUGUGAUGCAGCUGAGCU___GGAAUGUGC ......((((((..((((((...........(((((...))))).(((((..........))))).))))))(((......))).)))))).............. (-26.91 = -27.08 + 0.17)
| Location | 3,822,752 – 3,822,845 |
|---|---|
| Length | 93 |
| Sequences | 6 |
| Columns | 102 |
| Reading direction | forward |
| Mean pairwise identity | 91.11 |
| Mean single sequence MFE | -51.92 |
| Consensus MFE | -42.78 |
| Energy contribution | -43.08 |
| Covariance contribution | 0.31 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.35 |
| Structure conservation index | 0.82 |
| SVM decision value | 0.70 |
| SVM RNA-class probability | 0.825909 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
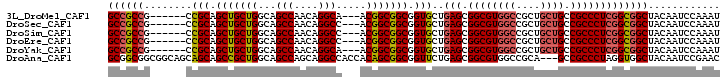
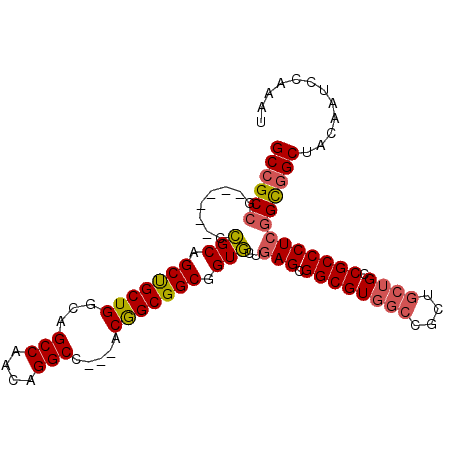
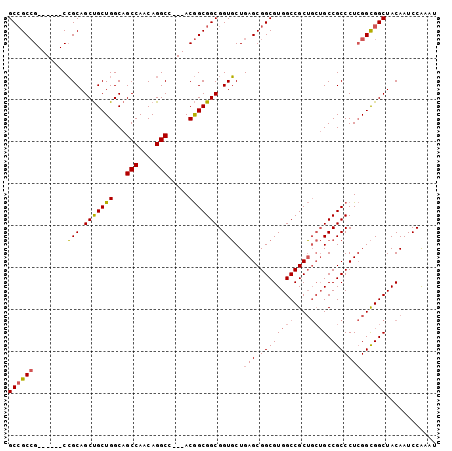
>3L_DroMel_CAF1 3822752 93 + 23771897 GCCGCCG------CCGCAGCUGCUGGCAGCCAACAGGCA---ACGGCGGCGGUGCUGAGCGGCGUGGCCGCUGCUGCCGCCCUCGGCGGCUACAAUCCAAAU (((((((------((((....)).))).(((....))).---..(((((((((....((((((...)))))))))))))))...))))))............ ( -52.60) >DroSec_CAF1 22448 93 + 1 GCCGCCG------CCGCAGCUGCUGGCAGCCAACAGGCC---ACGGCGGCGGUGCUGAGCGGCGUGGCCGCUGCUGCCGCCCUCGGCGGCUACAAUCCAAAU (((((((------((((....)).))).(((....))).---..(((((((((....((((((...)))))))))))))))...))))))............ ( -51.70) >DroSim_CAF1 22133 93 + 1 GCCGCCG------CCGCAGCUGCUGGCAGCCAACAGGCC---ACGGCGGCGGUGCUGAGCGGCGUGGCCGCUGCUGCCGCCCUCGGCGGCUACAAUCCAAAU (((((((------((((....)).))).(((....))).---..(((((((((....((((((...)))))))))))))))...))))))............ ( -51.70) >DroEre_CAF1 22870 93 + 1 GCCGCCG------CCGCAGCUGCUGGCAGCCAACAGGCC---ACGGCGGCGGUGCUGAGCGGCGUGGCCGCUGCUGCCGCCCUCGGCGGCUACAAUCCAAAU (((((((------((((....)).))).(((....))).---..(((((((((....((((((...)))))))))))))))...))))))............ ( -51.70) >DroYak_CAF1 22910 93 + 1 GCCGCCG------CCGCAGCUGCUGGCAGCCAACAGGCA---ACGGCGGCGGUGCUGAGCGGCGUGGCCGCUGCUGCCGCCCUCGGCGGCUACAAUCCAAAU (((((((------((((....)).))).(((....))).---..(((((((((....((((((...)))))))))))))))...))))))............ ( -52.60) >DroAna_CAF1 21720 99 + 1 GCGGCGGCGGCAGCAGCAGCCGCUGGCAGCCAGCAGGCCACCACAGCGGCGGUUCUGAGCGGCGUGGCCGCA---GCCGCCCUAGGUGGCUACAAUCCGAAC (((((.(((.(..(((..(((((((...(((....))).....)))))))....)))...).))).)))))(---((((((...)))))))........... ( -51.20) >consensus GCCGCCG______CCGCAGCUGCUGGCAGCCAACAGGCC___ACGGCGGCGGUGCUGAGCGGCGUGGCCGCUGCUGCCGCCCUCGGCGGCUACAAUCCAAAU ((((((........(((.(((((((...(((....))).....))))))).)))..(((.((((((((....)))).)))))))))))))............ (-42.78 = -43.08 + 0.31)
| Location | 3,822,752 – 3,822,845 |
|---|---|
| Length | 93 |
| Sequences | 6 |
| Columns | 102 |
| Reading direction | reverse |
| Mean pairwise identity | 91.11 |
| Mean single sequence MFE | -56.23 |
| Consensus MFE | -43.18 |
| Energy contribution | -43.93 |
| Covariance contribution | 0.75 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.87 |
| Structure conservation index | 0.77 |
| SVM decision value | 0.15 |
| SVM RNA-class probability | 0.607639 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
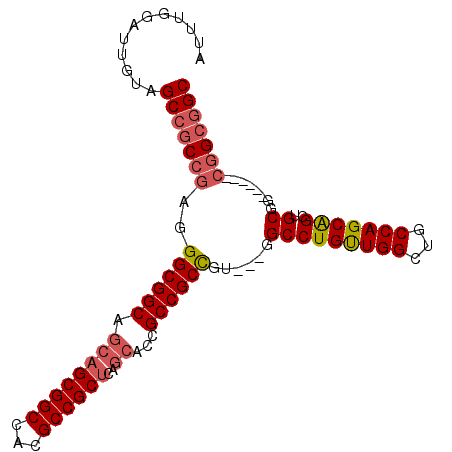
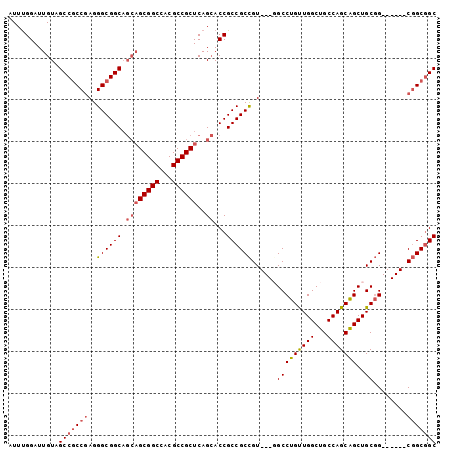
>3L_DroMel_CAF1 3822752 93 - 23771897 AUUUGGAUUGUAGCCGCCGAGGGCGGCAGCAGCGGCCACGCCGCUCAGCACCGCCGCCGU---UGCCUGUUGGCUGCCAGCAGCUGCGG------CGGCGGC ............((((((...)))))).((((((((...))))))..)).((((((((((---.((.((((((...)))))))).))))------)))))). ( -55.70) >DroSec_CAF1 22448 93 - 1 AUUUGGAUUGUAGCCGCCGAGGGCGGCAGCAGCGGCCACGCCGCUCAGCACCGCCGCCGU---GGCCUGUUGGCUGCCAGCAGCUGCGG------CGGCGGC ............((((((...)))))).((((((((...))))))..)).(((((((((.---.((.((((((...))))))))..)))------)))))). ( -58.70) >DroSim_CAF1 22133 93 - 1 AUUUGGAUUGUAGCCGCCGAGGGCGGCAGCAGCGGCCACGCCGCUCAGCACCGCCGCCGU---GGCCUGUUGGCUGCCAGCAGCUGCGG------CGGCGGC ............((((((...)))))).((((((((...))))))..)).(((((((((.---.((.((((((...))))))))..)))------)))))). ( -58.70) >DroEre_CAF1 22870 93 - 1 AUUUGGAUUGUAGCCGCCGAGGGCGGCAGCAGCGGCCACGCCGCUCAGCACCGCCGCCGU---GGCCUGUUGGCUGCCAGCAGCUGCGG------CGGCGGC ............((((((...)))))).((((((((...))))))..)).(((((((((.---.((.((((((...))))))))..)))------)))))). ( -58.70) >DroYak_CAF1 22910 93 - 1 AUUUGGAUUGUAGCCGCCGAGGGCGGCAGCAGCGGCCACGCCGCUCAGCACCGCCGCCGU---UGCCUGUUGGCUGCCAGCAGCUGCGG------CGGCGGC ............((((((...)))))).((((((((...))))))..)).((((((((((---.((.((((((...)))))))).))))------)))))). ( -55.70) >DroAna_CAF1 21720 99 - 1 GUUCGGAUUGUAGCCACCUAGGGCGGC---UGCGGCCACGCCGCUCAGAACCGCCGCUGUGGUGGCCUGCUGGCUGCCAGCGGCUGCUGCUGCCGCCGCCGC ...(((....(((....))).((((((---.(((((...(((((.(((........)))(((..(((....)))..)))))))).))))).)))))).))). ( -49.90) >consensus AUUUGGAUUGUAGCCGCCGAGGGCGGCAGCAGCGGCCACGCCGCUCAGCACCGCCGCCGU___GGCCUGUUGGCUGCCAGCAGCUGCGG______CGGCGGC ............(((((((..((((((.((((((((...))))))..))...))))))......(((((((((...)))))))..))........))))))) (-43.18 = -43.93 + 0.75)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:04:40 2006