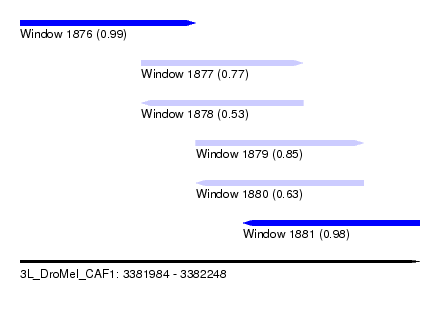
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 3,381,984 – 3,382,248 |
| Length | 264 |
| Max. P | 0.985335 |

| Location | 3,381,984 – 3,382,100 |
|---|---|
| Length | 116 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.16 |
| Mean single sequence MFE | -41.78 |
| Consensus MFE | -35.76 |
| Energy contribution | -37.36 |
| Covariance contribution | 1.60 |
| Combinations/Pair | 1.10 |
| Mean z-score | -1.99 |
| Structure conservation index | 0.86 |
| SVM decision value | 2.00 |
| SVM RNA-class probability | 0.985335 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 3381984 116 + 23771897 CAGCAUGUCUGCAGUGCGAGUGCGAAUCUUGCACCGUCUAUGGAACUAUGCUCAUCUGCCAUCGCGUGUGUAUGCGGGCAUGGGUCCAUAUAG----UAUGUACUGGCAUGCUGAUACUG ((((((((...(((((((.((((((...)))))))...((((((.((((((((...((((((...))).)))...)))))))).))))))...----...))))))))))))))...... ( -45.70) >DroSec_CAF1 49392 110 + 1 CAGCAUGUCUGCAGUG------CGAAUCUUGCACCGUCUAUGGAACUAUGCUCAUCUGCCAUCGCGUGUGUCUGCGGGCAUGGGUCCAUAUAG----UAUGUACUGGCAUGCUGAUACUG ((((((((((((((((------(((...))))))....((((((.((((((((....((....))((......)))))))))).))))))...----..))))..)))))))))...... ( -41.70) >DroSim_CAF1 47544 116 + 1 CAGCAUGUCUGCAGUGCGAGUGCGAAUCUUGCACCGUCUAUGGAACUAUGCUCAUCUGCCAUCGCGUGUGUCUGCGGGCAUGGGUCCAUAUAG----UAUGUACUGGCAUGCUGAUACUG ((((((((...(((((((.((((((...)))))))...((((((.((((((((....((....))((......)))))))))).))))))...----...))))))))))))))...... ( -45.70) >DroEre_CAF1 48384 116 + 1 CAGCAUGUCUGCAGUGCGAGUGCGAAUCUAGCACCGUCUAUGGAACCAUGCUCAUCUGCCAUCGCGUGUGUCUGCGGGCAUGGGUCUGUAUAG----UAUGUACUGGCGUGCUGAUACUG ((((((((...(((((((.((((.......)))))...((((((.((((((((....((....))((......)))))))))).))))))...----...))))))))))))))...... ( -44.10) >DroYak_CAF1 48290 112 + 1 CAGCAUGUCUGCAGUGCGAGUGCGAAUCUUGCACCGUCUAUGGAACUAUGCUCAUCUGCCAUCGCGUGUGUCU--------GGGUCUGUAUAGAAUGUAUGUACUGGCAUACUGAUACUG ..((((((((..((.(((.((((((...)))))))))))..)))..)))))(((..(((((..(((((..(((--------(........))).)..)))))..)))))...)))..... ( -31.70) >consensus CAGCAUGUCUGCAGUGCGAGUGCGAAUCUUGCACCGUCUAUGGAACUAUGCUCAUCUGCCAUCGCGUGUGUCUGCGGGCAUGGGUCCAUAUAG____UAUGUACUGGCAUGCUGAUACUG ((((((((((((((((((((.......)))))))....((((((.((((((((....(((((...))).))....)))))))).)))))).........))))..)))))))))...... (-35.76 = -37.36 + 1.60)

| Location | 3,382,064 – 3,382,171 |
|---|---|
| Length | 107 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.27 |
| Mean single sequence MFE | -33.74 |
| Consensus MFE | -26.48 |
| Energy contribution | -26.48 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.71 |
| Structure conservation index | 0.78 |
| SVM decision value | 0.52 |
| SVM RNA-class probability | 0.769023 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 3382064 107 + 23771897 UGGGUCCAUAUAG----UAUGUACUGGCAUGCUGAUACUGAUACGGAUAC------UGAUAGCUUAUCA---CCGCCGCUUCCUUGCUCGUUAGAGCAGCUUCACUGGCAAUCGUAUCAG ..........(((----(((((....))))))))...(((((((((....------(((((...)))))---..((((.....((((((....))))))......))))..))))))))) ( -33.10) >DroSec_CAF1 49466 107 + 1 UGGGUCCAUAUAG----UAUGUACUGGCAUGCUGAUACUGAUACGGAUAC------UGAUAGCUUAUCA---CCGCCGCUUCCUUGCUCGUUAGAGCAGCUCCACUGGCAAUCGUAUCAG ..........(((----(((((....))))))))...(((((((((....------(((((...)))))---..((((.....((((((....))))))......))))..))))))))) ( -33.10) >DroSim_CAF1 47624 107 + 1 UGGGUCCAUAUAG----UAUGUACUGGCAUGCUGAUACUGAUACGGAUAC------UGAUAGCUUAUCA---CCGCCGCUUCCUUGCUCGUUAGAGCAGCUCCACUGGCAAUCGUAUCAG ..........(((----(((((....))))))))...(((((((((....------(((((...)))))---..((((.....((((((....))))))......))))..))))))))) ( -33.10) >DroEre_CAF1 48464 113 + 1 UGGGUCUGUAUAG----UAUGUACUGGCGUGCUGAUACUGAUACGGAUACUGAUACUGAUAGCUUAUCA---CCGCCGCUUCCUUGCUCAUUAGAGCAGCUCCACUGGCAGGCGUAUCAG ...((((((((((----(((((((....))))..))))).)))))))).((((((((((((...)))))---..(((.......(((((....)))))(((.....))).)))))))))) ( -38.50) >DroYak_CAF1 48363 113 + 1 -GGGUCUGUAUAGAAUGUAUGUACUGGCAUACUGAUACUGAUACGGAUAC------UGAUAGCUUAUCACCACCGCCGCUUCCUUGCUCAUUAGAGCAGCUCCAAUGGCAAUCGUAUCAG -..(((.(((((.......))))).))).........(((((((((....------(((((...))))).....((((.....((((((....))))))......))))..))))))))) ( -30.90) >consensus UGGGUCCAUAUAG____UAUGUACUGGCAUGCUGAUACUGAUACGGAUAC______UGAUAGCUUAUCA___CCGCCGCUUCCUUGCUCGUUAGAGCAGCUCCACUGGCAAUCGUAUCAG ...(((..((((.......))))..))).........((((((((...........(((((...))))).....((((.....((((((....))))))......))))...)))))))) (-26.48 = -26.48 + 0.00)

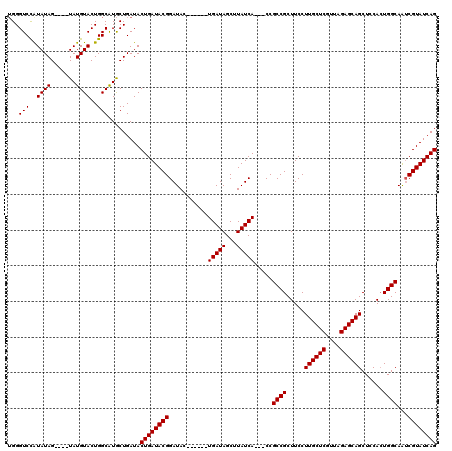
| Location | 3,382,064 – 3,382,171 |
|---|---|
| Length | 107 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.27 |
| Mean single sequence MFE | -32.68 |
| Consensus MFE | -24.72 |
| Energy contribution | -24.76 |
| Covariance contribution | 0.04 |
| Combinations/Pair | 1.04 |
| Mean z-score | -1.56 |
| Structure conservation index | 0.76 |
| SVM decision value | -0.01 |
| SVM RNA-class probability | 0.526673 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 3382064 107 - 23771897 CUGAUACGAUUGCCAGUGAAGCUGCUCUAACGAGCAAGGAAGCGGCGG---UGAUAAGCUAUCA------GUAUCCGUAUCAGUAUCAGCAUGCCAGUACAUA----CUAUAUGGACCCA (((((((.((((((.((.....(((((....))))).....)))))))---).....).)))))------)..(((((((.(((((..((......))..)))----))))))))).... ( -31.10) >DroSec_CAF1 49466 107 - 1 CUGAUACGAUUGCCAGUGGAGCUGCUCUAACGAGCAAGGAAGCGGCGG---UGAUAAGCUAUCA------GUAUCCGUAUCAGUAUCAGCAUGCCAGUACAUA----CUAUAUGGACCCA ((((((..((..((.((.(..((((((....))))..))...).))))---..))....)))))------)..(((((((.(((((..((......))..)))----))))))))).... ( -31.70) >DroSim_CAF1 47624 107 - 1 CUGAUACGAUUGCCAGUGGAGCUGCUCUAACGAGCAAGGAAGCGGCGG---UGAUAAGCUAUCA------GUAUCCGUAUCAGUAUCAGCAUGCCAGUACAUA----CUAUAUGGACCCA ((((((..((..((.((.(..((((((....))))..))...).))))---..))....)))))------)..(((((((.(((((..((......))..)))----))))))))).... ( -31.70) >DroEre_CAF1 48464 113 - 1 CUGAUACGCCUGCCAGUGGAGCUGCUCUAAUGAGCAAGGAAGCGGCGG---UGAUAAGCUAUCAGUAUCAGUAUCCGUAUCAGUAUCAGCACGCCAGUACAUA----CUAUACAGACCCA (((((((..(((((.((.....(((((....))))).....)))))))---(((((...))))))))))))..((.((((.(((((..((......))..)))----)))))).)).... ( -32.40) >DroYak_CAF1 48363 113 - 1 CUGAUACGAUUGCCAUUGGAGCUGCUCUAAUGAGCAAGGAAGCGGCGGUGGUGAUAAGCUAUCA------GUAUCCGUAUCAGUAUCAGUAUGCCAGUACAUACAUUCUAUACAGACCC- (((((((..((((((((((((...)))))))).))))(((.((...((((((.....)))))).------)).)))))))))).....(((((......)))))...............- ( -36.50) >consensus CUGAUACGAUUGCCAGUGGAGCUGCUCUAACGAGCAAGGAAGCGGCGG___UGAUAAGCUAUCA______GUAUCCGUAUCAGUAUCAGCAUGCCAGUACAUA____CUAUAUGGACCCA ((((((((.(((((.((.....(((((....))))).....)))))))...(((((...)))))...........))))))))..................................... (-24.72 = -24.76 + 0.04)

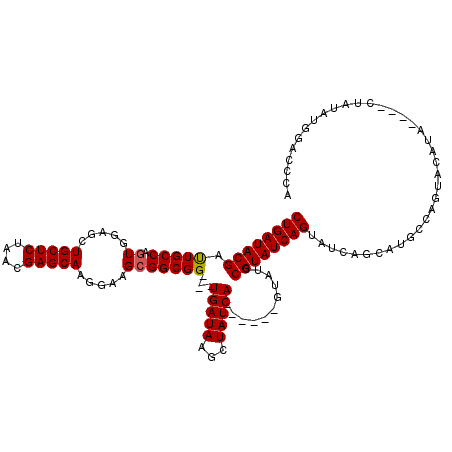
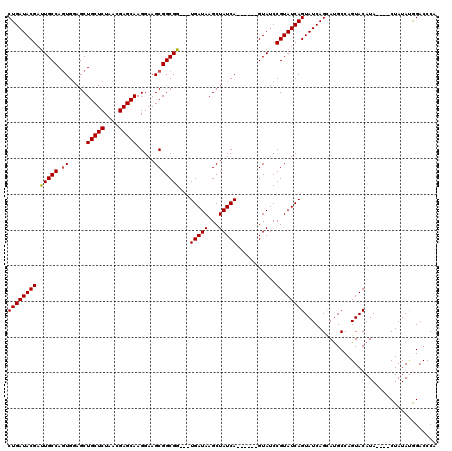
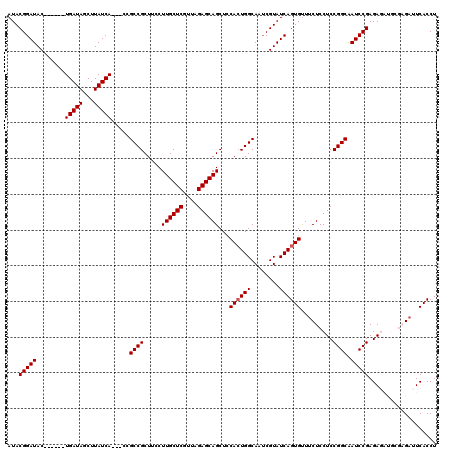
| Location | 3,382,100 – 3,382,211 |
|---|---|
| Length | 111 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 90.17 |
| Mean single sequence MFE | -31.60 |
| Consensus MFE | -25.52 |
| Energy contribution | -25.72 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.72 |
| Structure conservation index | 0.81 |
| SVM decision value | 0.77 |
| SVM RNA-class probability | 0.845641 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 3382100 111 + 23771897 AUACGGAUAC------UGAUAGCUUAUCA---CCGCCGCUUCCUUGCUCGUUAGAGCAGCUUCACUGGCAAUCGUAUCAGUGUUCCUCCUCCGGCGAUCCGAGAGAUGCGAGAUUCACCU ...(((....------(((((...)))))---))).(((.((((((.((((..(((.((...((((((........))))))...)).)))..))))..)))).)).))).......... ( -30.20) >DroSec_CAF1 49502 111 + 1 AUACGGAUAC------UGAUAGCUUAUCA---CCGCCGCUUCCUUGCUCGUUAGAGCAGCUCCACUGGCAAUCGUAUCAGUGUUUCUCCUCCGGCAAUCCGAGAGAUGCGAGAUUCACCU .....(((((------(((((...)))))---..((((.....((((((....))))))......))))....))))).(((..((((((((((....))).)))....))))..))).. ( -32.40) >DroSim_CAF1 47660 111 + 1 AUACGGAUAC------UGAUAGCUUAUCA---CCGCCGCUUCCUUGCUCGUUAGAGCAGCUCCACUGGCAAUCGUAUCAGUGUUUCUCCUCCGGCAAUCCGAGAGAUGCGAGAUUCACCU .....(((((------(((((...)))))---..((((.....((((((....))))))......))))....))))).(((..((((((((((....))).)))....))))..))).. ( -32.40) >DroEre_CAF1 48500 110 + 1 AUACGGAUACUGAUACUGAUAGCUUAUCA---CCGCCGCUUCCUUGCUCAUUAGAGCAGCUCCACUGGCAGGCGUAUCAGUGCUUCUCCUCCGGCAAUCCGAGA-------GAUUCACCU ....((.((((((((((((((...)))))---..(((.......(((((....)))))(((.....))).))))))))))))......((((((....))).))-------).....)). ( -34.40) >DroYak_CAF1 48402 107 + 1 AUACGGAUAC------UGAUAGCUUAUCACCACCGCCGCUUCCUUGCUCAUUAGAGCAGCUCCAAUGGCAAUCGUAUCAGUGUUUCUCCUCCGGCAAUCCGAGA-------GAUUCACCU .....(((((------(((((...))))).....((((.....((((((....))))))......))))....))))).(((..((((...(((....))).))-------))..))).. ( -28.60) >consensus AUACGGAUAC______UGAUAGCUUAUCA___CCGCCGCUUCCUUGCUCGUUAGAGCAGCUCCACUGGCAAUCGUAUCAGUGUUUCUCCUCCGGCAAUCCGAGAGAUGCGAGAUUCACCU ...(((((........(((((...))))).....((((.....((((((....))))))...((((((........)))))).........)))).)))))................... (-25.52 = -25.72 + 0.20)

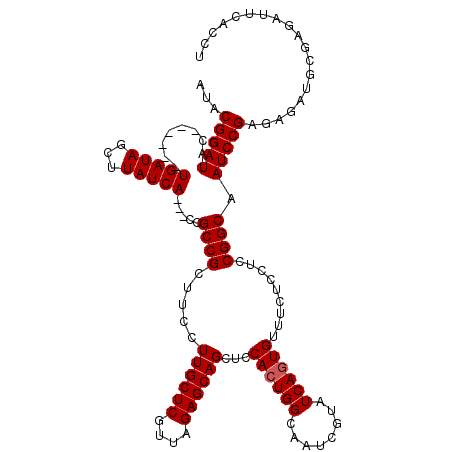
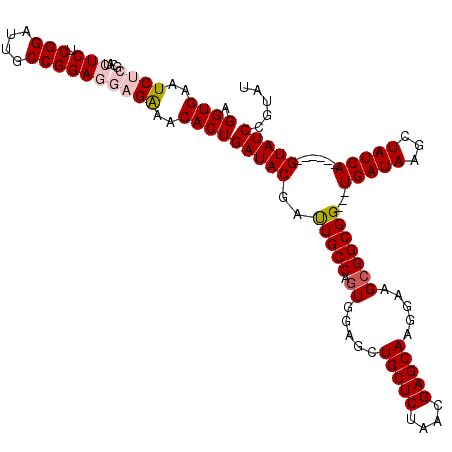
| Location | 3,382,100 – 3,382,211 |
|---|---|
| Length | 111 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.17 |
| Mean single sequence MFE | -36.44 |
| Consensus MFE | -28.32 |
| Energy contribution | -29.40 |
| Covariance contribution | 1.08 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.56 |
| Structure conservation index | 0.78 |
| SVM decision value | 0.20 |
| SVM RNA-class probability | 0.629058 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 3382100 111 - 23771897 AGGUGAAUCUCGCAUCUCUCGGAUCGCCGGAGGAGGAACACUGAUACGAUUGCCAGUGAAGCUGCUCUAACGAGCAAGGAAGCGGCGG---UGAUAAGCUAUCA------GUAUCCGUAU ..(((.....))).(((..(((....)))..)))(((..((((((((.((((((.((.....(((((....))))).....)))))))---).....).)))))------)).))).... ( -35.70) >DroSec_CAF1 49502 111 - 1 AGGUGAAUCUCGCAUCUCUCGGAUUGCCGGAGGAGAAACACUGAUACGAUUGCCAGUGGAGCUGCUCUAACGAGCAAGGAAGCGGCGG---UGAUAAGCUAUCA------GUAUCCGUAU .((((..((((....(((.(((....))))))))))..))))(((((.((((((.((.....(((((....))))).....)))))))---)((((...)))).------)))))..... ( -35.60) >DroSim_CAF1 47660 111 - 1 AGGUGAAUCUCGCAUCUCUCGGAUUGCCGGAGGAGAAACACUGAUACGAUUGCCAGUGGAGCUGCUCUAACGAGCAAGGAAGCGGCGG---UGAUAAGCUAUCA------GUAUCCGUAU .((((..((((....(((.(((....))))))))))..))))(((((.((((((.((.....(((((....))))).....)))))))---)((((...)))).------)))))..... ( -35.60) >DroEre_CAF1 48500 110 - 1 AGGUGAAUC-------UCUCGGAUUGCCGGAGGAGAAGCACUGAUACGCCUGCCAGUGGAGCUGCUCUAAUGAGCAAGGAAGCGGCGG---UGAUAAGCUAUCAGUAUCAGUAUCCGUAU ..((...((-------(..(((....)))..)))...))((((((((..(((((.((.....(((((....))))).....)))))))---(((((...)))))))))))))........ ( -38.20) >DroYak_CAF1 48402 107 - 1 AGGUGAAUC-------UCUCGGAUUGCCGGAGGAGAAACACUGAUACGAUUGCCAUUGGAGCUGCUCUAAUGAGCAAGGAAGCGGCGGUGGUGAUAAGCUAUCA------GUAUCCGUAU ..((...((-------(..(((....)))..)))...))(((((((..((..((((((..(((((((....)))).....)))..))))))..))....)))))------))........ ( -37.10) >consensus AGGUGAAUCUCGCAUCUCUCGGAUUGCCGGAGGAGAAACACUGAUACGAUUGCCAGUGGAGCUGCUCUAACGAGCAAGGAAGCGGCGG___UGAUAAGCUAUCA______GUAUCCGUAU .((((..((((....(((.(((....))))))))))..))))(((((..(((((.((.....(((((....))))).....)))))))...(((((...)))))......)))))..... (-28.32 = -29.40 + 1.08)

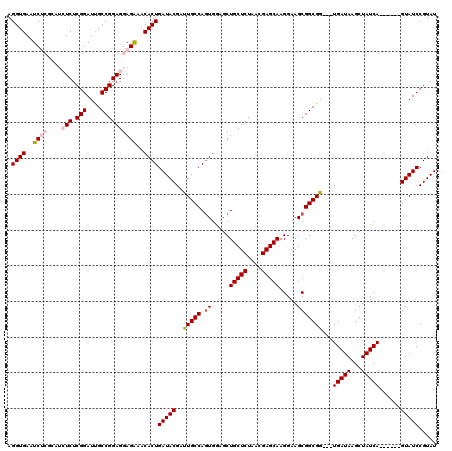
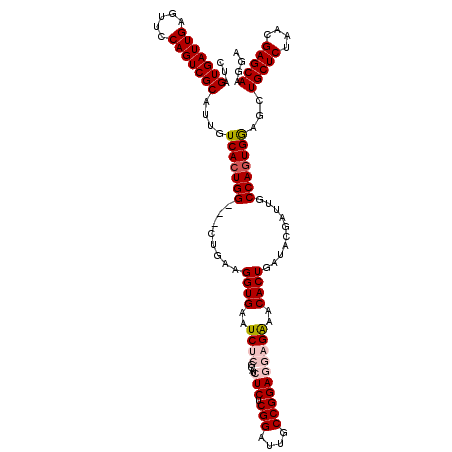
| Location | 3,382,131 – 3,382,248 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.17 |
| Mean single sequence MFE | -41.33 |
| Consensus MFE | -34.21 |
| Energy contribution | -35.29 |
| Covariance contribution | 1.08 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.06 |
| Structure conservation index | 0.83 |
| SVM decision value | 1.77 |
| SVM RNA-class probability | 0.976608 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 3382131 117 - 23771897 CUAGUGAUUGAGUUCCAGUCGCAUUGUCACUGG---CUGAAGGUGAAUCUCGCAUCUCUCGGAUCGCCGGAGGAGGAACACUGAUACGAUUGCCAGUGAAGCUGCUCUAACGAGCAAGGA ...(((((((.....)))))))....(((((((---(....((((..((((....(((.(((....))))))))))..)))).........))))))))...(((((....))))).... ( -42.42) >DroSec_CAF1 49533 117 - 1 CUAGUGAUUGAGUUCCAGUCGCAUUGUCACUGG---CUGAAGGUGAAUCUCGCAUCUCUCGGAUUGCCGGAGGAGAAACACUGAUACGAUUGCCAGUGGAGCUGCUCUAACGAGCAAGGA ...(((((((.....)))))))....(((((((---(....((((..((((....(((.(((....))))))))))..)))).........))))))))...(((((....))))).... ( -42.42) >DroSim_CAF1 47691 117 - 1 CUAGUGAUUGAGUUCCAGUCGCAUUGUCACUGG---CUGAAGGUGAAUCUCGCAUCUCUCGGAUUGCCGGAGGAGAAACACUGAUACGAUUGCCAGUGGAGCUGCUCUAACGAGCAAGGA ...(((((((.....)))))))....(((((((---(....((((..((((....(((.(((....))))))))))..)))).........))))))))...(((((....))))).... ( -42.42) >DroEre_CAF1 48537 110 - 1 CUAGUGAUUGAGUUCCAGUCGCAUUGUCACUGG---CUGAAGGUGAAUC-------UCUCGGAUUGCCGGAGGAGAAGCACUGAUACGCCUGCCAGUGGAGCUGCUCUAAUGAGCAAGGA ...(((((((.....)))))))....(((((((---(.(..((((..((-------((((((....)))..)))))..))))......)..))))))))...(((((....))))).... ( -40.90) >DroYak_CAF1 48436 113 - 1 GUAGUGAUUGAGUUCCAGUCGCAUUGUCACUGGCGGCUGAAGGUGAAUC-------UCUCGGAUUGCCGGAGGAGAAACACUGAUACGAUUGCCAUUGGAGCUGCUCUAAUGAGCAAGGA ((((((((((.....))))))).((.((.(((((((((((..(.....)-------..)))).)))))))..)).)).......)))..((((((((((((...)))))))).))))... ( -38.50) >consensus CUAGUGAUUGAGUUCCAGUCGCAUUGUCACUGG___CUGAAGGUGAAUCUCGCAUCUCUCGGAUUGCCGGAGGAGAAACACUGAUACGAUUGCCAGUGGAGCUGCUCUAACGAGCAAGGA ...(((((((.....)))))))....(((((((........((((..((((....(((.(((....))))))))))..))))..........)))))))...(((((....))))).... (-34.21 = -35.29 + 1.08)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 10:01:53 2006