| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
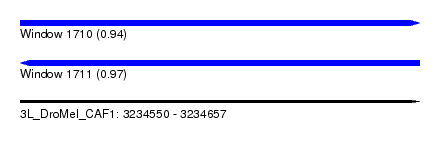
| Location | 3,234,550 – 3,234,657 |
| Length | 107 |
| Max. P | 0.971386 |

| Location | 3,234,550 – 3,234,657 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 79.42 |
| Mean single sequence MFE | -39.95 |
| Consensus MFE | -35.38 |
| Energy contribution | -35.54 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.00 |
| Mean z-score | -5.36 |
| Structure conservation index | 0.89 |
| SVM decision value | 1.34 |
| SVM RNA-class probability | 0.944256 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
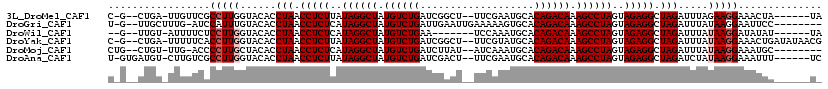
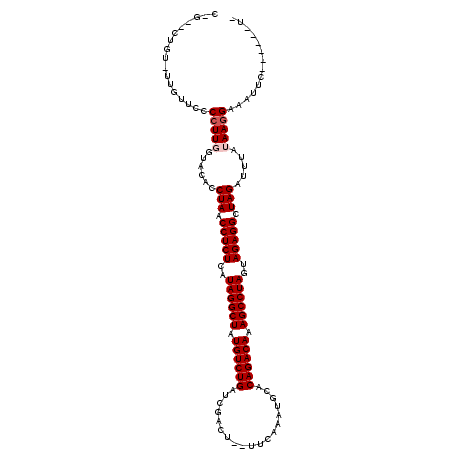
>3L_DroMel_CAF1 3234550 107 + 23771897 UA------UAGUUUCCUUCUAAAUCUAGCCUCUACUAGGCUUUGUCUGUGCAUUCGAA--AGCCGAUCAGACAUAGCCUAUAAGAGGUUAGGUGUACCAAGGCGAACAA-UCAG--C-G ..------..((((((((....(((((((((((..((((((.((((((.....(((..--...))).)))))).))))))..))))))))))).....)))).))))..-....--.-. ( -42.30) >DroGri_CAF1 14818 107 + 1 --------GGAAUUCCUUAUAAAUCUAGCCUCUACUAGGCUUUGUCUGUGCACUUUUUCAAUUCAAUCAGACAUAGCCUAUGAGAGGUUAGGUGUACAAAUGGAU-CAAAGCAA--C-A --------.....(((......(((((((((((..((((((.((((((...................)))))).))))))..)))))))))))........))).-........--.-. ( -36.05) >DroWil_CAF1 38118 101 + 1 UA------AUAUAUCCUUAUAAAUCUAGCCUCUACUAGGCUUUGUCUGUGCAUUUGGA-------UUCAGACAUAGCCUAUGAGAGGUUAGGUGUACCAAGGAGAAAAU-ACAA--C-- ..------.....(((((....(((((((((((..((((((.((((((..(.....).-------..)))))).))))))..))))))))))).....)))))......-....--.-- ( -39.90) >DroYak_CAF1 14160 113 + 1 CGUUAUAUCAGUUUCCUUAUAAAUCUAGCCUCUACUAGGCUUUGUCUGUGCAUACGAA--AGCCGAUCAGACAUAGCCUAUAAGAGGUUAGGUGUACCAAGGUGAAAAA-UCAG--C-G ((((.......((.((((....(((((((((((..((((((.((((((......(...--.).....)))))).))))))..))))))))))).....)))).))....-..))--)-) ( -40.22) >DroMoj_CAF1 13516 105 + 1 --------GCAUUUCCUUAUAAAUCUAGCCUCUACUAGGCUUUGUCUGUGCAUUUGAU--AUAAGAUCAGACAUAGCCUAUGAGAGGUUAGGUGUAGCAAGGGGU-CAA-ACAG--CAG --------((....((((.((.(((((((((((..((((((.((((((...((((...--...)))))))))).))))))..))))))))))).))..))))(..-...-.).)--).. ( -40.10) >DroAna_CAF1 14532 109 + 1 GA------AAAUUUCCUUAUAGAUCUAGCCUCUACUAGGCUUUGUCUGUGCAUUCGAA--AGUCGAUCAGACAUAGCCUAUAAGAGGUUAGGUGUACCAAGGCGACAAG-ACAUCAC-A ..------......((((.((.(((((((((((..((((((.((((((.....(((..--...))).)))))).))))))..))))))))))).))..)))).......-.......-. ( -41.10) >consensus _A______AAAUUUCCUUAUAAAUCUAGCCUCUACUAGGCUUUGUCUGUGCAUUCGAA__AGCCGAUCAGACAUAGCCUAUAAGAGGUUAGGUGUACCAAGGCGAAAAA_ACAG__C_G ..............((((.((.(((((((((((..((((((.((((((...................)))))).))))))..))))))))))).))..))))................. (-35.38 = -35.54 + 0.17)

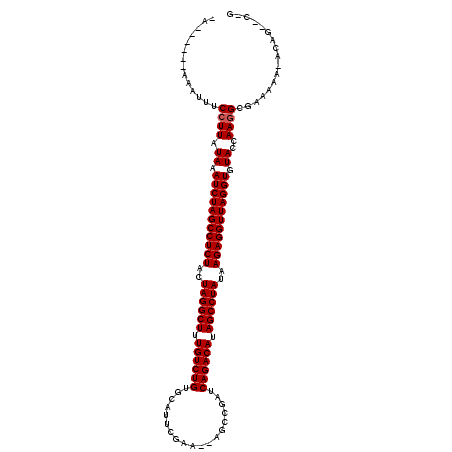
| Location | 3,234,550 – 3,234,657 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 79.42 |
| Mean single sequence MFE | -33.24 |
| Consensus MFE | -29.14 |
| Energy contribution | -29.64 |
| Covariance contribution | 0.50 |
| Combinations/Pair | 1.00 |
| Mean z-score | -3.56 |
| Structure conservation index | 0.88 |
| SVM decision value | 1.67 |
| SVM RNA-class probability | 0.971386 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 3234550 107 - 23771897 C-G--CUGA-UUGUUCGCCUUGGUACACCUAACCUCUUAUAGGCUAUGUCUGAUCGGCU--UUCGAAUGCACAGACAAAGCCUAGUAGAGGCUAGAUUUAGAAGGAAACUA------UA .-.--((((-.(((..((....))))).(((.(((((..((((((.((((((....((.--.......)).)))))).))))))..))))).)))..)))).((....)).------.. ( -34.40) >DroGri_CAF1 14818 107 - 1 U-G--UUGCUUUG-AUCCAUUUGUACACCUAACCUCUCAUAGGCUAUGUCUGAUUGAAUUGAAAAAGUGCACAGACAAAGCCUAGUAGAGGCUAGAUUUAUAAGGAAUUCC-------- .-.--........-.(((..(((((...(((.(((((..((((((.((((((..((..((....))...)))))))).))))))..))))).)))...)))))))).....-------- ( -32.50) >DroWil_CAF1 38118 101 - 1 --G--UUGU-AUUUUCUCCUUGGUACACCUAACCUCUCAUAGGCUAUGUCUGAA-------UCCAAAUGCACAGACAAAGCCUAGUAGAGGCUAGAUUUAUAAGGAUAUAU------UA --.--....-......((((((......(((.(((((..((((((.((((((..-------..........)))))).))))))..))))).))).....)))))).....------.. ( -33.00) >DroYak_CAF1 14160 113 - 1 C-G--CUGA-UUUUUCACCUUGGUACACCUAACCUCUUAUAGGCUAUGUCUGAUCGGCU--UUCGUAUGCACAGACAAAGCCUAGUAGAGGCUAGAUUUAUAAGGAAACUGAUAUAACG .-.--.(((-....)))(((((......(((.(((((..((((((.((((((....((.--.......)).)))))).))))))..))))).))).....))))).............. ( -35.00) >DroMoj_CAF1 13516 105 - 1 CUG--CUGU-UUG-ACCCCUUGCUACACCUAACCUCUCAUAGGCUAUGUCUGAUCUUAU--AUCAAAUGCACAGACAAAGCCUAGUAGAGGCUAGAUUUAUAAGGAAAUGC-------- ...--....-...-...(((((.((...(((.(((((..((((((.((((((.......--..........)))))).))))))..))))).)))...)))))))......-------- ( -30.33) >DroAna_CAF1 14532 109 - 1 U-GUGAUGU-CUUGUCGCCUUGGUACACCUAACCUCUUAUAGGCUAUGUCUGAUCGACU--UUCGAAUGCACAGACAAAGCCUAGUAGAGGCUAGAUCUAUAAGGAAAUUU------UC .-(((((..-...)))))((((......(((.(((((..((((((.((((((.(((...--..))).....)))))).))))))..))))).))).....)))).......------.. ( -34.20) >consensus C_G__CUGU_UUGUUCCCCUUGGUACACCUAACCUCUCAUAGGCUAUGUCUGAUCGACU__UUCAAAUGCACAGACAAAGCCUAGUAGAGGCUAGAUUUAUAAGGAAAUUC______U_ .................(((((......(((.(((((..((((((.((((((...................)))))).))))))..))))).))).....))))).............. (-29.14 = -29.64 + 0.50)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:59:10 2006