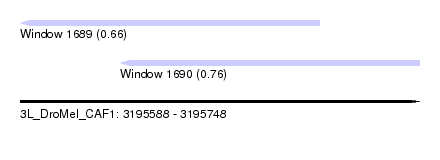
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 3,195,588 – 3,195,748 |
| Length | 160 |
| Max. P | 0.757416 |

| Location | 3,195,588 – 3,195,708 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.89 |
| Mean single sequence MFE | -40.59 |
| Consensus MFE | -23.66 |
| Energy contribution | -23.58 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.53 |
| Mean z-score | -2.01 |
| Structure conservation index | 0.58 |
| SVM decision value | 0.27 |
| SVM RNA-class probability | 0.662751 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
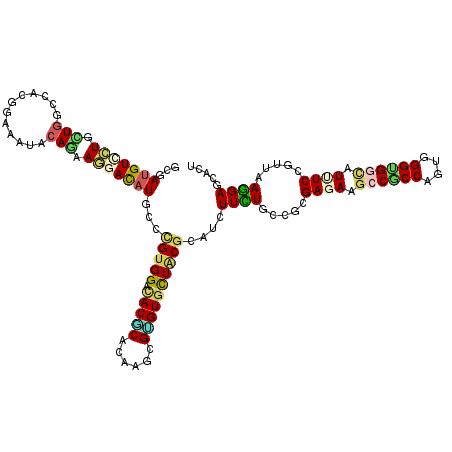
>3L_DroMel_CAF1 3195588 120 - 23771897 CGUGGACACCCACAAGCGGGUGCUACGUAUCUUCUGUCGGGAAAAAGCUCCAGUGGGUGGCAUAUCCGUUAAGGAGCAUUUCGAGAUUAAUGUGGCUCCUAUUACAAUAGCCAUAACGAA (((((.(((((......)))))))))).........(((.((((..(((((..((((((...))))))....))))).))))........(((((((...........))))))).))). ( -37.30) >DroVir_CAF1 16096 120 - 1 CGUGGACACGCACAAGCGUGUGCUGCGCAUCUUUUGCCGCGAGAAGCCGCCCGUAGGCGGCAUUUCAGUUAAAGAUCACUUCGAGAUUAACGUGGCACCUAUAACCAUAGCAAUUACCAA ..(((((((((....))))))((((.........((((((((((.((((((....)))))).)))).(((((...((.....))..)))))))))))..........)))).....))). ( -44.21) >DroGri_CAF1 17773 120 - 1 CGUGGACACGCAUAAGCGGGUGUUGCGCAUCUUCUGUCGCGAGAAGCCGCCAGUGGGUGGCAUUUCGGUUAAGGAUCACUUUGAGAUCAAUGUAGCGCCCAUAACCAUUGCGAUCACCAA .((((...((((.....((((((((((......((..(.(((((.((((((....)))))).))))))...))((((.......))))..))))))))))........)))).))))... ( -41.62) >DroWil_CAF1 14704 120 - 1 UGUGGAUACACACAAACGUGUUCUACGCAUCUUUUGUAGGGAGAAGCCACCAGUUGGUGGUAUCUCAGUUAAGGAACACUUUGAAAUCAAUGUGGCACCGAUUACCAUAGCCAUUACAAA ((((....))))((((.(((((((..(((.....)))...((((.((((((....)))))).))))......)))))))))))........(((((.............)))))...... ( -36.62) >DroMoj_CAF1 14238 120 - 1 CGUGGAUACGCAUAAGCGUGUGCUACGCAUCUUUUGCCGCGAGAAGCCGCCGGUUGGCGGCAUUUCCGUAAAGGACCACUUUGAGAUCAAUGUGGCGCCCAUUACCAUAGCGAUCACCAA (((((((((((....)))))).)))))..((((((((.(.((((.((((((....)))))).))))))))))))).........((((..(((((.........)))))..))))..... ( -45.50) >DroAna_CAF1 13980 120 - 1 GGUGGACACUCACAAGCGAGUGCUCCGCAUCUUCUGCAGGGAGAAGGCACCAGUCGGUGGUAUAUCCGUGAAGGAGCACUUCGAGAUCAAUGUGGCACCCAUUACUAUAGCCAUCACGAA .((((((((((......))))).)))))..(((((.((.(((...(.((((....)))).)...))).)).)))))....(((.(((...(((((.........)))))...))).))). ( -38.30) >consensus CGUGGACACGCACAAGCGUGUGCUACGCAUCUUCUGCCGCGAGAAGCCGCCAGUGGGUGGCAUUUCCGUUAAGGAGCACUUCGAGAUCAAUGUGGCACCCAUUACCAUAGCCAUCACCAA .((((.(((((......)))))))))((............((((.((((((....)))))).)))).((((.((..(((............)))....)).))))....))......... (-23.66 = -23.58 + -0.08)

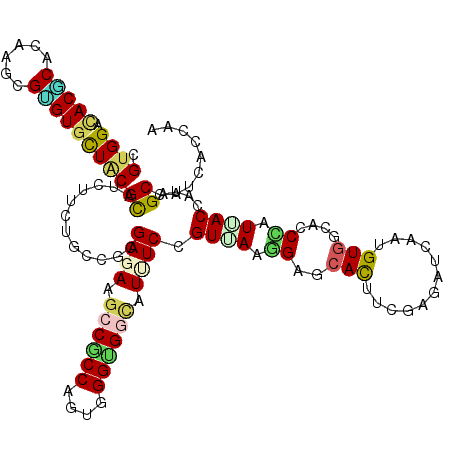
| Location | 3,195,628 – 3,195,748 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.94 |
| Mean single sequence MFE | -45.41 |
| Consensus MFE | -26.84 |
| Energy contribution | -26.73 |
| Covariance contribution | -0.10 |
| Combinations/Pair | 1.53 |
| Mean z-score | -2.18 |
| Structure conservation index | 0.59 |
| SVM decision value | 0.49 |
| SVM RNA-class probability | 0.757416 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
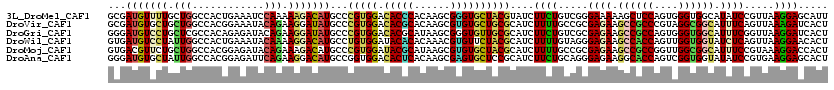
>3L_DroMel_CAF1 3195628 120 - 23771897 GCGAUGUUUUGCUGGCCACUGAAAUCCAAAAAGACAUGCCCGUGGACACCCACAAGCGGGUGCUACGUAUCUUCUGUCGGGAAAAAGCUCCAGUGGGUGGCAUAUCCGUUAAGGAGCAUU ((.......(((((.((((((...........((((....(((((.(((((......)))))))))).......))))(((......))))))))).)))))..(((.....)))))... ( -41.20) >DroVir_CAF1 16136 120 - 1 GCGAUGUGCUGCUGGCCACGGAAAUACAGAAGGAUAUGCCCGUGGACACGCACAAGCGUGUGCUGCGCAUCUUUUGCCGCGAGAAGCCGCCCGUAGGCGGCAUUUCAGUUAAAGAUCACU ..(((((((.((...((((((..(((........)))..))))))((((((....)))))))).)))))))(((((.(..((((.((((((....)))))).)))).).)))))...... ( -50.00) >DroGri_CAF1 17813 120 - 1 GGGAUGUCCUGCUCGCCACAGAGAUACAGAAGGAUAUGCCCGUGGACACGCAUAAGCGGGUGUUGCGCAUCUUCUGUCGCGAGAAGCCGCCAGUGGGUGGCAUUUCGGUUAAGGAUCACU ((((((((((.(((......))).......))))))).)))((((((((((....)))((((.....))))...)))).(((((.((((((....)))))).))))).........))). ( -45.30) >DroWil_CAF1 14744 120 - 1 GUGAUGUCCUAUUGGCCACUGAAAUACAAAAGGACAUGCCUGUGGAUACACACAAACGUGUUCUACGCAUCUUUUGUAGGGAGAAGCCACCAGUUGGUGGUAUCUCAGUUAAGGAACACU (((...((((..((((........((((((((...((((..(((((.((((......)))))))))))))))))))))..((((.((((((....)))))).)))).)))))))).))). ( -42.60) >DroMoj_CAF1 14278 120 - 1 GUGACGUUCUGCUGGCCACGGAGAUACAGAAAGACAUGCCCGUGGAUACGCAUAAGCGUGUGCUACGCAUCUUUUGCCGCGAGAAGCCGCCGGUUGGCGGCAUUUCCGUAAAGGACCACU (((......((((((((((((..................))))).((((((....)))))))))).)))((((((((.(.((((.((((((....)))))).))))))))))))).))). ( -45.17) >DroAna_CAF1 14020 120 - 1 GGGAUGUGCUAUUGGCCACGGAGAUUCAGAAGGACAUGCCGGUGGACACUCACAAGCGAGUGCUCCGCAUCUUCUGCAGGGAGAAGGCACCAGUCGGUGGUAUAUCCGUGAAGGAGCACU .(((((((((((((((...((...(((....)))..((((.((((((((((......))))).))))).(((((.....))))).)))))).)))))))))))))))(((......))). ( -48.20) >consensus GCGAUGUCCUGCUGGCCACGGAAAUACAGAAGGACAUGCCCGUGGACACGCACAAGCGUGUGCUACGCAUCUUCUGCCGCGAGAAGCCGCCAGUGGGUGGCAUUUCCGUUAAGGAGCACU ...(((((((.(((............))).)))))))...(((((.(((((......))))))))))....((((.....((((.((((((....)))))).)))).....))))..... (-26.84 = -26.73 + -0.10)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:58:49 2006