
| Sequence ID | 3L_DroMel_CAF1 |
|---|---|
| Location | 3,000,239 – 3,000,357 |
| Length | 118 |
| Max. P | 0.879667 |

| Location | 3,000,239 – 3,000,357 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 82.20 |
| Mean single sequence MFE | -37.90 |
| Consensus MFE | -25.29 |
| Energy contribution | -28.60 |
| Covariance contribution | 3.31 |
| Combinations/Pair | 1.09 |
| Mean z-score | -1.48 |
| Structure conservation index | 0.67 |
| SVM decision value | -0.05 |
| SVM RNA-class probability | 0.508796 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 3000239 118 + 23771897 UGUCAGAUCCCACGACCACAGCACCCACAUUGGGAUCCAAGUAGAUACUGUGGAUGUGAUCUGGCAUAGAGAAGCCCUGCAGAGAGGAAUGAUCCAAGGGCAGGGACUCGAUUCCUAC (((((((((..(((.((((((..(((.....)))(((......))).)))))).))))))))))))..(((...((((((.....(((....)))....)))))).)))......... ( -45.30) >DroSec_CAF1 34580 118 + 1 UGUCAGAUCCCACGACCACAGCACCCACAUUGGGAUCCAAGAAGAUACUGUGGAUGUGAUCUGGCAUAGAGAAGCCCUGCAGAGAGGAAUGAUCCAGAGGCAGGGACUCGAUUCCCAC (((((((((..(((.((((((..(((.....)))(((......))).)))))).))))))))))))..(((...((((((.....(((....)))....)))))).)))......... ( -45.30) >DroSim_CAF1 35120 118 + 1 UGUCAGAUCCCACGACCACAGCACCCACAUUGGGAUCCAAGAAGAUACUGUGGAUGUGAUCUGGCAUAGAGAAGCCCUGCAGAGAGGAAUGAUCCAAGGGCAAGGACUCGAUUCCCAC (((((((((..(((.((((((..(((.....)))(((......))).)))))).))))))))))))..(((...((.(((.....(((....)))....))).)).)))......... ( -38.80) >DroEre_CAF1 37195 118 + 1 UGUCAGUUCCGACCACCACAGCACCCACAUUGGGAUCCAAGUAGAUACUGUGGAUGUGAUCUGGCAUAGAAAAGCCCUGCAGCGAGGAAUGAUCCAGGGGCAGGGAAUCGAUGCCCAC .(((......)))..((((((..(((.....)))(((......))).)))))).........(((((.((....((((((..(..(((....))).)..))))))..)).)))))... ( -38.30) >DroYak_CAF1 34936 118 + 1 UGUCAGCACCCACCACCACAGCACCCACAUUGGGAUCCAAGAAGAUACUGUGUAUGUGAUCUGGCAUGGAAAAUCCCUGCAAGGAGGAAUGAUCCAGUGGCAUGGACUCGAUACCCAC ((((((....(((...(((((..(((.....)))(((......))).)))))...)))..))))))(((....(((((....)).))).(((((((......)))).)))....))). ( -29.40) >DroPer_CAF1 37361 112 + 1 UGU------CGACGGCCACGGCGCCCACAUUCGCAUCUAAGUACAUUUUUUGCACCUGGUCCACCAUGGAGUUCUCCUGCGUGGAGGCCAAGUCCAGCGGCAGACUCUCGAUCCCGAC .((------(((.((((...(((........)))..(((.((.((.....)).)).)))(((((((.(((....))))).))))))))).((((........)))).)))))...... ( -30.30) >consensus UGUCAGAUCCCACGACCACAGCACCCACAUUGGGAUCCAAGAAGAUACUGUGGAUGUGAUCUGGCAUAGAGAAGCCCUGCAGAGAGGAAUGAUCCAGGGGCAGGGACUCGAUUCCCAC ((((((....((((.((((((..(((.....)))(((......))).)))))).))))..))))))..(((...((((((.....(((....)))....)))))).)))......... (-25.29 = -28.60 + 3.31)

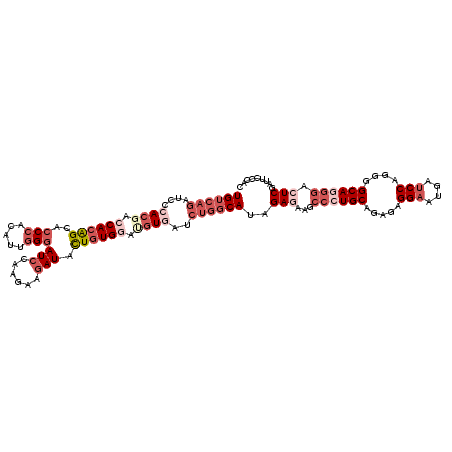
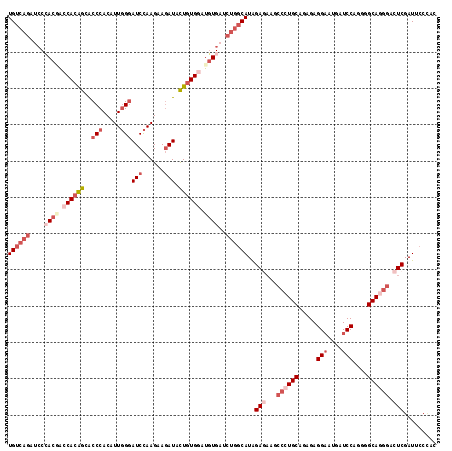
| Location | 3,000,239 – 3,000,357 |
|---|---|
| Length | 118 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 82.20 |
| Mean single sequence MFE | -46.47 |
| Consensus MFE | -30.41 |
| Energy contribution | -33.38 |
| Covariance contribution | 2.98 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.93 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.91 |
| SVM RNA-class probability | 0.879667 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3L_DroMel_CAF1 3000239 118 - 23771897 GUAGGAAUCGAGUCCCUGCCCUUGGAUCAUUCCUCUCUGCAGGGCUUCUCUAUGCCAGAUCACAUCCACAGUAUCUACUUGGAUCCCAAUGUGGGUGCUGUGGUCGUGGGAUCUGACA (((((....(((.((((((....(((....))).....))))))))).)))))..((((((.((((((((((((((((((((...)))).)))))))))))))..))).))))))... ( -51.70) >DroSec_CAF1 34580 118 - 1 GUGGGAAUCGAGUCCCUGCCUCUGGAUCAUUCCUCUCUGCAGGGCUUCUCUAUGCCAGAUCACAUCCACAGUAUCUUCUUGGAUCCCAAUGUGGGUGCUGUGGUCGUGGGAUCUGACA ((.(((.(((((.((((((....(((....))).....))))))...))).........((((..(((((((((((.(((((...)))).).)))))))))))..))))))))).)). ( -47.30) >DroSim_CAF1 35120 118 - 1 GUGGGAAUCGAGUCCUUGCCCUUGGAUCAUUCCUCUCUGCAGGGCUUCUCUAUGCCAGAUCACAUCCACAGUAUCUUCUUGGAUCCCAAUGUGGGUGCUGUGGUCGUGGGAUCUGACA ((.(((.(((((.((((((....(((....))).....))))))...))).........((((..(((((((((((.(((((...)))).).)))))))))))..))))))))).)). ( -44.60) >DroEre_CAF1 37195 118 - 1 GUGGGCAUCGAUUCCCUGCCCCUGGAUCAUUCCUCGCUGCAGGGCUUUUCUAUGCCAGAUCACAUCCACAGUAUCUACUUGGAUCCCAAUGUGGGUGCUGUGGUGGUCGGAACUGACA ...(((((.((..((((((....(((....))).....))))))....)).))))).(((((...(((((((((((((((((...)))).)))))))))))))))))).......... ( -51.10) >DroYak_CAF1 34936 118 - 1 GUGGGUAUCGAGUCCAUGCCACUGGAUCAUUCCUCCUUGCAGGGAUUUUCCAUGCCAGAUCACAUACACAGUAUCUUCUUGGAUCCCAAUGUGGGUGCUGUGGUGGUGGGUGCUGACA ((.((((((((((((.(((....(((....))).....))).))))))............(((...((((((((((.(((((...)))).).))))))))))...))))))))).)). ( -42.90) >DroPer_CAF1 37361 112 - 1 GUCGGGAUCGAGAGUCUGCCGCUGGACUUGGCCUCCACGCAGGAGAACUCCAUGGUGGACCAGGUGCAAAAAAUGUACUUAGAUGCGAAUGUGGGCGCCGUGGCCGUCG------ACA (((((((.(....))))(((((((.((((((..(((((.(((((....))).))))))))))))).))......(..((((.((....)).))))..).)))))...))------)). ( -41.20) >consensus GUGGGAAUCGAGUCCCUGCCCCUGGAUCAUUCCUCUCUGCAGGGCUUCUCUAUGCCAGAUCACAUCCACAGUAUCUACUUGGAUCCCAAUGUGGGUGCUGUGGUCGUGGGAUCUGACA ((.(((.(((((.((((((....(((....))).....))))))...)))..........(((..(((((((((((.(((((...)))).).)))))))))))..))))).))).)). (-30.41 = -33.38 + 2.98)

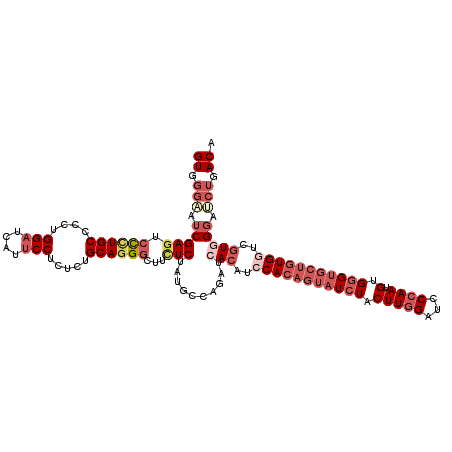
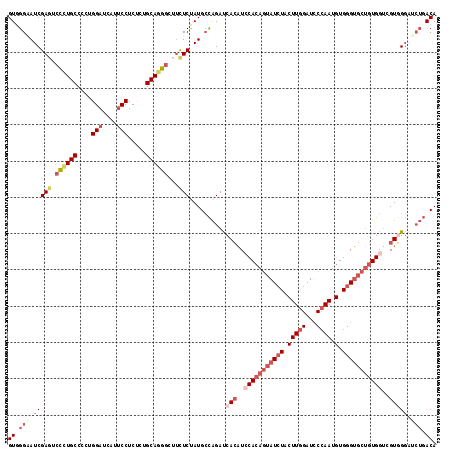
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:56:47 2006