
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 869,419 – 869,530 |
| Length | 111 |
| Max. P | 0.996101 |

| Location | 869,419 – 869,530 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 76.75 |
| Mean single sequence MFE | -49.38 |
| Consensus MFE | -25.60 |
| Energy contribution | -25.77 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.12 |
| Mean z-score | -3.31 |
| Structure conservation index | 0.52 |
| SVM decision value | 2.65 |
| SVM RNA-class probability | 0.996101 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
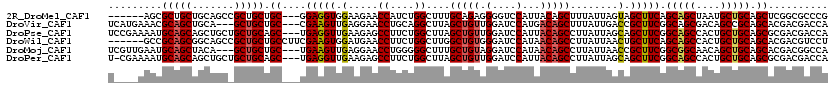
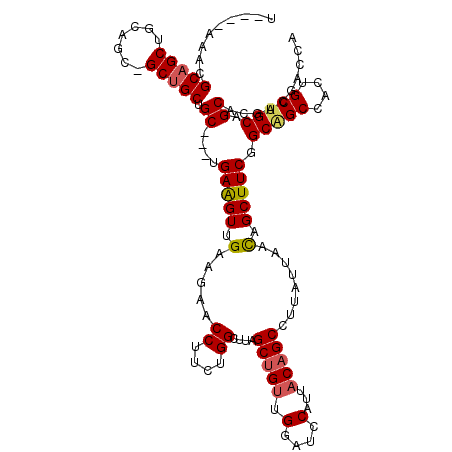
>2R_DroMel_CAF1 869419 111 + 20766785 ------AGCGCUGCUGCAGCCGCUGCUGC---GGAGGUGGAAGAACCAUCUGGCUUUGCAGAGGGGUCCAUUACAGCUUUAUUAGUAGCUUCAGCAGCUAAUGCUGCAGCUCGGCGCCCG ------.(((((((((((((.(((((((.---.(..(((((....((.((((......)))).)).)))))..)((((........)))).)))))))....))))))))..)))))... ( -51.70) >DroVir_CAF1 6801 114 + 1 UCAUGAAACGCAGCUGCA---GCUGCUGC---CGAAGUUGAGGAACCUGCAGGCUUAGCUGUUGGAUCCAUGACAGCUUUAUUGACCGCUUCGGCAGCGACAGCCGCAGCACGACGACCA ............(((((.---((((((((---((((((.(.(((.((.((((......)))).)).)))....(((.....))).).)))))))))))...))).))))).......... ( -45.00) >DroPse_CAF1 1586 117 + 1 UCCGAAAAUGCAGCAGCUGCUGCUGCAGC---UGAGGUUGAAGAGCCUUCUGGCUUAGCUGUUGGAUCCAUUACAGCCUUAUUAGCAGCUUCGGCAGCCACUGCUGCAGCGCGACGACCA ..((....((((((((..(((((((.(((---((....(((.(((((....))))).(((((((....))..))))).)))....))))).)))))))..))))))))...))....... ( -52.10) >DroWil_CAF1 16127 114 + 1 ------GCCGCAGCGGCAGCCGCUGCUGCCUUCGAAGUGGAUGAACCUUCUGGCUUGGCUGUGGGAUCCAUAACAGCCUUAUUAACUGCUUCAGCAGCCACUGCUGCAGCACGACGUCCU ------(((((((((((....))))))))....((((.(......))))).)))..((((((.(....)...))))))........((((.((((((...)))))).))))......... ( -46.40) >DroMoj_CAF1 6153 114 + 1 UCGUUGAAUGCAGCUACA---GCUGCUGC---UGAAGUUGAGGAACCUGGGGGCUUUGCUGUAGGAUCCAUAACAGCCUUAUUAACCGCUUCGGCGGCAACAGCUGCAGCACGACGGCCA ((((((..(((((((.((---((....))---))..((((.(((.((((.((......)).)))).)))................((((....)))))))))))))))...))))))... ( -48.00) >DroPer_CAF1 27357 116 + 1 U-CGAAAAUGCAGCAGCUGCUGCUGCAGC---UGAGGUUGAAGAGCCUUCUGGCUUAGCUGUUGGAUCCAUUACAGCCUUAUUAGCAGCUUCGGCAGCCACUGCUGCAGCGCGACGACCA (-((....((((((((..(((((((.(((---((....(((.(((((....))))).(((((((....))..))))).)))....))))).)))))))..))))))))...)))...... ( -53.10) >consensus U____AAACGCAGCUGCAGC_GCUGCUGC___UGAAGUUGAAGAACCUUCUGGCUUAGCUGUUGGAUCCAUUACAGCCUUAUUAACAGCUUCGGCAGCCACUGCUGCAGCACGACGACCA .........(((((.......))))).((....(((((.(.....((....))....(((((.(....)...)))))........).))))).(((((....))))).)).......... (-25.60 = -25.77 + 0.17)
| Location | 869,419 – 869,530 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 76.75 |
| Mean single sequence MFE | -47.30 |
| Consensus MFE | -28.76 |
| Energy contribution | -29.90 |
| Covariance contribution | 1.14 |
| Combinations/Pair | 1.29 |
| Mean z-score | -2.86 |
| Structure conservation index | 0.61 |
| SVM decision value | 2.31 |
| SVM RNA-class probability | 0.992093 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
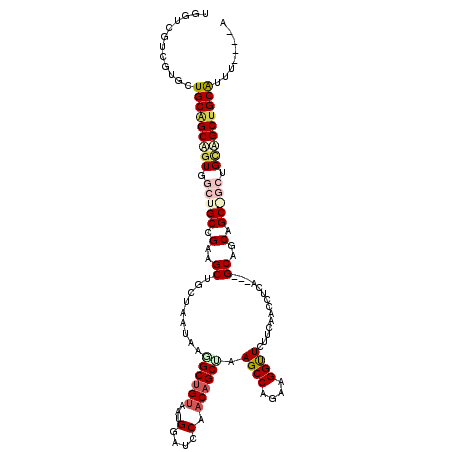
>2R_DroMel_CAF1 869419 111 - 20766785 CGGGCGCCGAGCUGCAGCAUUAGCUGCUGAAGCUACUAAUAAAGCUGUAAUGGACCCCUCUGCAAAGCCAGAUGGUUCUUCCACCUCC---GCAGCAGCGGCUGCAGCAGCGCU------ ..(((((...((((((((....(((((((.((((........)))).....(((((..((((......)))).)))))..........---.))))))).)))))))).)))))------ ( -49.30) >DroVir_CAF1 6801 114 - 1 UGGUCGUCGUGCUGCGGCUGUCGCUGCCGAAGCGGUCAAUAAAGCUGUCAUGGAUCCAACAGCUAAGCCUGCAGGUUCCUCAACUUCG---GCAGCAGC---UGCAGCUGCGUUUCAUGA (((.(((...(((((((((...((((((((((..(.......((((((..((....)))))))).((((....))))...)..)))))---))))))))---)))))).)))..)))... ( -49.50) >DroPse_CAF1 1586 117 - 1 UGGUCGUCGCGCUGCAGCAGUGGCUGCCGAAGCUGCUAAUAAGGCUGUAAUGGAUCCAACAGCUAAGCCAGAAGGCUCUUCAACCUCA---GCUGCAGCAGCAGCUGCUGCAUUUUCGGA ......(((...(((((((((.(((((.(.(((((.......((((((..((....)))))))).((((....)))).........))---))).).))))).)))))))))....))). ( -47.40) >DroWil_CAF1 16127 114 - 1 AGGACGUCGUGCUGCAGCAGUGGCUGCUGAAGCAGUUAAUAAGGCUGUUAUGGAUCCCACAGCCAAGCCAGAAGGUUCAUCCACUUCGAAGGCAGCAGCGGCUGCCGCUGCGGC------ ..........((((((((.(..(((((((.............((((((...((...))))))))..(((.((((.........))))...)))..)))))))..).))))))))------ ( -50.40) >DroMoj_CAF1 6153 114 - 1 UGGCCGUCGUGCUGCAGCUGUUGCCGCCGAAGCGGUUAAUAAGGCUGUUAUGGAUCCUACAGCAAAGCCCCCAGGUUCCUCAACUUCA---GCAGCAGC---UGUAGCUGCAUUCAACGA ........(((((((((((((((((((....))))).......((((....(((.(((...((...))....))).))).......))---))))))))---)))))).))......... ( -39.70) >DroPer_CAF1 27357 116 - 1 UGGUCGUCGCGCUGCAGCAGUGGCUGCCGAAGCUGCUAAUAAGGCUGUAAUGGAUCCAACAGCUAAGCCAGAAGGCUCUUCAACCUCA---GCUGCAGCAGCAGCUGCUGCAUUUUCG-A ......(((...(((((((((.(((((.(.(((((.......((((((..((....)))))))).((((....)))).........))---))).).))))).)))))))))....))-) ( -47.50) >consensus UGGUCGUCGUGCUGCAGCAGUGGCUGCCGAAGCUGCUAAUAAGGCUGUAAUGGAUCCAACAGCUAAGCCAGAAGGUUCUUCAACCUCA___GCAGCAGC_GCUGCAGCUGCAUUU____A ............(((((((((.(((((.(..((.........((((((...(....).)))))).((((....))))..............))..).))))).)))))))))........ (-28.76 = -29.90 + 1.14)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:23:18 2006