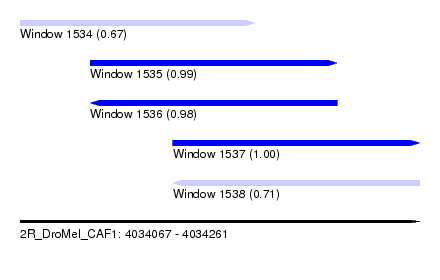
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 4,034,067 – 4,034,261 |
| Length | 194 |
| Max. P | 0.998568 |

| Location | 4,034,067 – 4,034,181 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 90.12 |
| Mean single sequence MFE | -40.47 |
| Consensus MFE | -31.68 |
| Energy contribution | -32.80 |
| Covariance contribution | 1.12 |
| Combinations/Pair | 1.09 |
| Mean z-score | -1.58 |
| Structure conservation index | 0.78 |
| SVM decision value | 0.29 |
| SVM RNA-class probability | 0.668443 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 4034067 114 + 20766785 UAGC------AGUACAAGCGUUGAAUUUAAAUUGUCACUUAAUGCGGCGGCCGACCUUCCGUCAUCAGUCGCUCCGGCUAAUUGCUGGUCGGUCGCGCUGCCUGCCAGCAAUUGUUGCUG ..((------(((....(((((((..............))))))).((((((((((....((.((.(((((...))))).)).)).)))))))))))))))....(((((.....))))) ( -42.14) >DroSec_CAF1 16691 114 + 1 UACC------AGUACAAGCGUUGAAUUUAAAUUGUCGCUUAAUGCGGCGGCCGACAUUCCGUCAUCAGUCGCCCCGGCUAAUUGCUGGUCGGUCGCGCUGCCUGCCAGCAAUUGUUGCUG ....------.......(((((((.......(((((((.....)))))))..(((.....))).)))).)))..(((((((((((((((.(((......))).)))))))))))..)))) ( -40.10) >DroSim_CAF1 18841 114 + 1 UAAC------AGUACAAGCGUUGAAUUUAAAUUGUCGCUUAAUGCGGCGGGCGACCUUCCGUCAUCAGUCGCCCCGGUUAAUUGCUGGUCGGUCGCGCUGCCUGCCAGCAAUUGUUGCUG ...(------((((....((.............(((((.....)))))(((((((............)))))))))..(((((((((((.(((......))).))))))))))).))))) ( -43.30) >DroEre_CAF1 16030 114 + 1 UACC------AGUACAAGCGUUGAAUUUAAAUUGCCGCUUAAUGCGGCGCCCGACCUUCCGUCAUCAGUCGGCCCGGCUAAUUGCUGGUCGGUCGGCCUGGCUGCCAGCAAUUGCUGCUG ...(------(((((((..((((..........(((((.....)))))((((((((..(((.(....).))).(((((.....)))))..)))))....)))...))))..))).))))) ( -43.00) >DroYak_CAF1 19835 120 + 1 UACCAGUGCCAGUACAAGCGUUGCAUUUAAAUUGUCGCUUAAUGCGACGGCCGACCUUCCGUCAUCAUUCGCCCCUGCUAAUUGCUGUUCGGUGGGCCCGCCUGCCAGCAAUUGUUGAUG ..(((((((..((....))...)))))......(((((.....)))))))..(((.....)))((((...((....))(((((((((...((((....))))...))))))))).)))). ( -33.80) >consensus UACC______AGUACAAGCGUUGAAUUUAAAUUGUCGCUUAAUGCGGCGGCCGACCUUCCGUCAUCAGUCGCCCCGGCUAAUUGCUGGUCGGUCGCGCUGCCUGCCAGCAAUUGUUGCUG .................(((((((........((((((.....))))))...(((.....))).)))).)))..(((((((((((((((.(((......))).)))))))))))..)))) (-31.68 = -32.80 + 1.12)

| Location | 4,034,101 – 4,034,221 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 90.75 |
| Mean single sequence MFE | -54.92 |
| Consensus MFE | -43.20 |
| Energy contribution | -44.68 |
| Covariance contribution | 1.48 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.96 |
| Structure conservation index | 0.79 |
| SVM decision value | 2.41 |
| SVM RNA-class probability | 0.993533 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 4034101 120 + 20766785 AAUGCGGCGGCCGACCUUCCGUCAUCAGUCGCUCCGGCUAAUUGCUGGUCGGUCGCGCUGCCUGCCAGCAAUUGUUGCUGCUGCAGCAGCAACAUGAACGCAAGCAGCAGCGGUGGCAUC .((((.((((((((((....((.((.(((((...))))).)).)).))))))))))(((((.(((..((...(((((((((....))))))))).....))..))))))))....)))). ( -59.20) >DroSec_CAF1 16725 120 + 1 AAUGCGGCGGCCGACAUUCCGUCAUCAGUCGCCCCGGCUAAUUGCUGGUCGGUCGCGCUGCCUGCCAGCAAUUGUUGCUGCUGCAGCAGCAAUAUGAACGCGAGCAGCAGCGGCGGCAUC .((((.(((((((((....((.(....).))...((((.....))))))))))).((((((.(((..((...(((((((((....))))))))).....))..))))))))))).)))). ( -57.50) >DroSim_CAF1 18875 120 + 1 AAUGCGGCGGGCGACCUUCCGUCAUCAGUCGCCCCGGUUAAUUGCUGGUCGGUCGCGCUGCCUGCCAGCAAUUGUUGCUGCUGCAGCAGCAAUAUGAACGCGAGCAGCAGCGGCGGCAUC ....(((((((((((............)))))))((((.....))))))))((((((((((.(((..((...(((((((((....))))))))).....))..)))))))).)))))... ( -57.50) >DroEre_CAF1 16064 120 + 1 AAUGCGGCGCCCGACCUUCCGUCAUCAGUCGGCCCGGCUAAUUGCUGGUCGGUCGGCCUGGCUGCCAGCAAUUGCUGCUGCUGCAGCAGCAAAAUGAAGGCGAGCAGCAACAGUUGCAUC ...(((((..((((((..(((.(....).))).(((((.....)))))..))))))....)))))..(((((((.(((((((((..((......))...)).))))))).)))))))... ( -54.80) >DroYak_CAF1 19875 120 + 1 AAUGCGACGGCCGACCUUCCGUCAUCAUUCGCCCCUGCUAAUUGCUGUUCGGUGGGCCCGCCUGCCAGCAAUUGUUGAUGCUGCAGCAGCAAUAUGAACGCGAGCAGAAGCGGUUGCAUC .(((((((((........))))).....((((..(((((((((((((...((((....))))...))))))))(((.(((.(((....))).))).)))...)))))..))))..)))). ( -45.60) >consensus AAUGCGGCGGCCGACCUUCCGUCAUCAGUCGCCCCGGCUAAUUGCUGGUCGGUCGCGCUGCCUGCCAGCAAUUGUUGCUGCUGCAGCAGCAAUAUGAACGCGAGCAGCAGCGGUGGCAUC .(((((((((........)))))......((((......((((((((((.(((......))).))))))))))(((((((((((..((......))...)).))))))))))))))))). (-43.20 = -44.68 + 1.48)

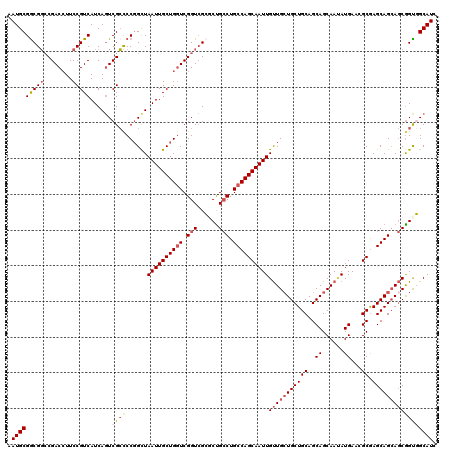
| Location | 4,034,101 – 4,034,221 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.75 |
| Mean single sequence MFE | -53.52 |
| Consensus MFE | -41.70 |
| Energy contribution | -43.30 |
| Covariance contribution | 1.60 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.52 |
| Structure conservation index | 0.78 |
| SVM decision value | 1.76 |
| SVM RNA-class probability | 0.975780 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 4034101 120 - 20766785 GAUGCCACCGCUGCUGCUUGCGUUCAUGUUGCUGCUGCAGCAGCAACAAUUGCUGGCAGGCAGCGCGACCGACCAGCAAUUAGCCGGAGCGACUGAUGACGGAAGGUCGGCCGCCGCAUU (((((....(((((((((.(((....(((((((((....)))))))))..))).)))).)))))(((.((((((..(.(((((.((...)).)))))....)..)))))).))).))))) ( -56.90) >DroSec_CAF1 16725 120 - 1 GAUGCCGCCGCUGCUGCUCGCGUUCAUAUUGCUGCUGCAGCAGCAACAAUUGCUGGCAGGCAGCGCGACCGACCAGCAAUUAGCCGGGGCGACUGAUGACGGAAUGUCGGCCGCCGCAUU (((((..((((((((((..((((.......)).)).))))))))...(((((((((..((........))..)))))))))....))((((.(((((........))))).))))))))) ( -52.80) >DroSim_CAF1 18875 120 - 1 GAUGCCGCCGCUGCUGCUCGCGUUCAUAUUGCUGCUGCAGCAGCAACAAUUGCUGGCAGGCAGCGCGACCGACCAGCAAUUAACCGGGGCGACUGAUGACGGAAGGUCGCCCGCCGCAUU (((((.((.((((((((..((((.......)).)).))))))))...(((((((((..((........))..))))))))).....((((((((..........)))))))))).))))) ( -55.50) >DroEre_CAF1 16064 120 - 1 GAUGCAACUGUUGCUGCUCGCCUUCAUUUUGCUGCUGCAGCAGCAGCAAUUGCUGGCAGCCAGGCCGACCGACCAGCAAUUAGCCGGGCCGACUGAUGACGGAAGGUCGGGCGCCGCAUU (((((....(((((((.(((((..((..(((((((((...))))))))).))..))).....((....)))).)))))))..(((.((((..(((....)))..)))).)))...))))) ( -52.80) >DroYak_CAF1 19875 120 - 1 GAUGCAACCGCUUCUGCUCGCGUUCAUAUUGCUGCUGCAGCAUCAACAAUUGCUGGCAGGCGGGCCCACCGAACAGCAAUUAGCAGGGGCGAAUGAUGACGGAAGGUCGGCCGUCGCAUU (((((...((((((((((.........(((((((((((((((........)))).)))).(((.....)))..))))))).))))))))))......(((((........)))))))))) ( -49.60) >consensus GAUGCCACCGCUGCUGCUCGCGUUCAUAUUGCUGCUGCAGCAGCAACAAUUGCUGGCAGGCAGCGCGACCGACCAGCAAUUAGCCGGGGCGACUGAUGACGGAAGGUCGGCCGCCGCAUU (((((..((((((((((..(((.......)))....))))))))...(((((((((..((........))..)))))))))....))((((.(((((..(....)))))).))))))))) (-41.70 = -43.30 + 1.60)

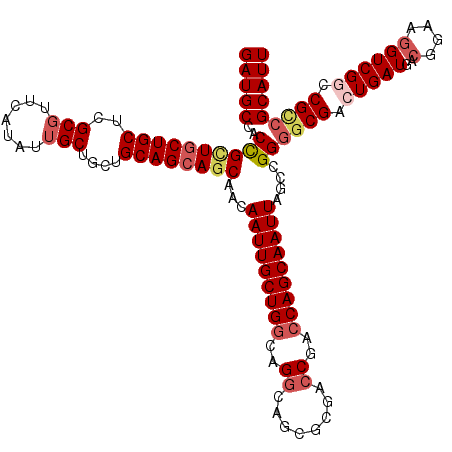
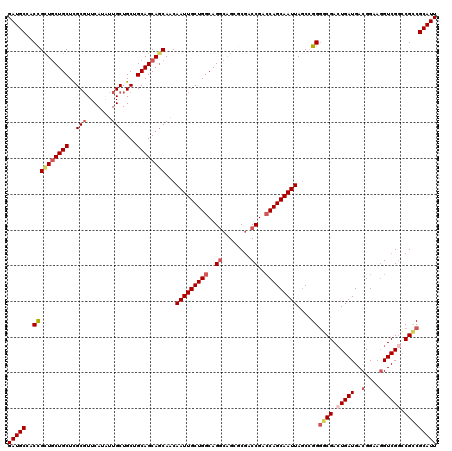
| Location | 4,034,141 – 4,034,261 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 92.08 |
| Mean single sequence MFE | -54.48 |
| Consensus MFE | -44.74 |
| Energy contribution | -46.74 |
| Covariance contribution | 2.00 |
| Combinations/Pair | 1.12 |
| Mean z-score | -3.08 |
| Structure conservation index | 0.82 |
| SVM decision value | 3.14 |
| SVM RNA-class probability | 0.998568 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 4034141 120 + 20766785 AUUGCUGGUCGGUCGCGCUGCCUGCCAGCAAUUGUUGCUGCUGCAGCAGCAACAUGAACGCAAGCAGCAGCGGUGGCAUCGUUAUUCCUGGUGCACUUCAGCUGCAGUGGGCGAUCAGUG ...((((((((.(((((((((.(((...((..(((((((((....)))))))))))...))).))))).(((((.((((((.......))))))......))))).)))).)))))))). ( -60.00) >DroSec_CAF1 16765 120 + 1 AUUGCUGGUCGGUCGCGCUGCCUGCCAGCAAUUGUUGCUGCUGCAGCAGCAAUAUGAACGCGAGCAGCAGCGGCGGCAUCGUUAUUCCUGGUGCACUUCAGCUGUAGUGGGCGAUCAGUG ...((((((((.(((((((((.(((...((..(((((((((....)))))))))))...))).))))).(((((.((((((.......))))))......))))).)))).)))))))). ( -56.70) >DroSim_CAF1 18915 120 + 1 AUUGCUGGUCGGUCGCGCUGCCUGCCAGCAAUUGUUGCUGCUGCAGCAGCAAUAUGAACGCGAGCAGCAGCGGCGGCAUCGUUAUUCCUGGUGCACUUCAGCUGCAGUGGGCGAUCAGUG ...((((((((.(((((((((.(((...((..(((((((((....)))))))))))...))).))))).(((((.((((((.......))))))......))))).)))).)))))))). ( -59.00) >DroEre_CAF1 16104 119 + 1 AUUGCUGGUCGGUCGGCCUGGCUGCCAGCAAUUGCUGCUGCUGCAGCAGCAAAAUGAAGGCGAGCAGCAACAGUUGCAUCGUUACUCCUGGUGCACUUCAGCUGCAGA-GGCGAUCAGUG ...(((((((.....((((..((((.(((..(((((((((...)))))))))..((((((((((((((....))))).))))(((.....)))..)))))))))))))-)))))))))). ( -50.70) >DroYak_CAF1 19915 120 + 1 AUUGCUGUUCGGUGGGCCCGCCUGCCAGCAAUUGUUGAUGCUGCAGCAGCAAUAUGAACGCGAGCAGAAGCGGUUGCAUCGUUAUUCCUGGUGCACUUCAGCUGCGGUGGGCGAUCAGUG ...((((.((.....((((((((((.((((........)))))))(((((........(((........)))..(((((((.......))))))).....)))))))))))))).)))). ( -46.00) >consensus AUUGCUGGUCGGUCGCGCUGCCUGCCAGCAAUUGUUGCUGCUGCAGCAGCAAUAUGAACGCGAGCAGCAGCGGUGGCAUCGUUAUUCCUGGUGCACUUCAGCUGCAGUGGGCGAUCAGUG ...((((((((.(((((((((.(((...((..(((((((((....)))))))))))...))).))))).(((((.((((((.......))))))......))))).)))).)))))))). (-44.74 = -46.74 + 2.00)

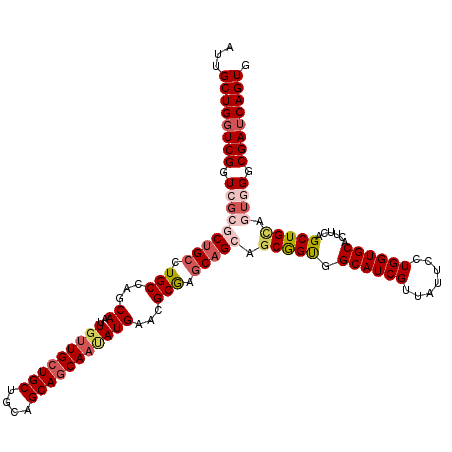
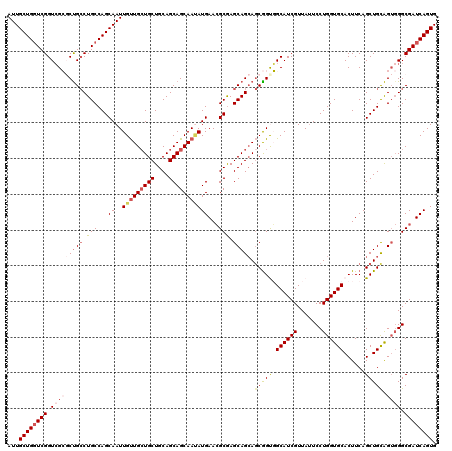
| Location | 4,034,141 – 4,034,261 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.08 |
| Mean single sequence MFE | -41.68 |
| Consensus MFE | -34.76 |
| Energy contribution | -35.40 |
| Covariance contribution | 0.64 |
| Combinations/Pair | 1.09 |
| Mean z-score | -1.42 |
| Structure conservation index | 0.83 |
| SVM decision value | 0.38 |
| SVM RNA-class probability | 0.712727 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 4034141 120 - 20766785 CACUGAUCGCCCACUGCAGCUGAAGUGCACCAGGAAUAACGAUGCCACCGCUGCUGCUUGCGUUCAUGUUGCUGCUGCAGCAGCAACAAUUGCUGGCAGGCAGCGCGACCGACCAGCAAU ...(((.(((.....(((((.(..(((((...(......)..)).)))..).)))))..))).)))(((((((((....)))))))))((((((((..((........))..)))))))) ( -46.50) >DroSec_CAF1 16765 120 - 1 CACUGAUCGCCCACUACAGCUGAAGUGCACCAGGAAUAACGAUGCCGCCGCUGCUGCUCGCGUUCAUAUUGCUGCUGCAGCAGCAACAAUUGCUGGCAGGCAGCGCGACCGACCAGCAAU ..................((((....(((...(......)..)))(((.(((((((((.(((......(((((((....)))))))....))).)))).))))))))......))))... ( -40.00) >DroSim_CAF1 18915 120 - 1 CACUGAUCGCCCACUGCAGCUGAAGUGCACCAGGAAUAACGAUGCCGCCGCUGCUGCUCGCGUUCAUAUUGCUGCUGCAGCAGCAACAAUUGCUGGCAGGCAGCGCGACCGACCAGCAAU ...(((.(((.....(((((....(.(((...(......)..))))...))))).....))).)))..(((((((....)))))))..((((((((..((........))..)))))))) ( -41.60) >DroEre_CAF1 16104 119 - 1 CACUGAUCGCC-UCUGCAGCUGAAGUGCACCAGGAGUAACGAUGCAACUGUUGCUGCUCGCCUUCAUUUUGCUGCUGCAGCAGCAGCAAUUGCUGGCAGCCAGGCCGACCGACCAGCAAU ..(((.(((((-(((((((((((((.((...((.(((((((.......))))))).)).)))))))..(((((((((...)))))))))..))).))))..)))).....)).))).... ( -43.70) >DroYak_CAF1 19915 120 - 1 CACUGAUCGCCCACCGCAGCUGAAGUGCACCAGGAAUAACGAUGCAACCGCUUCUGCUCGCGUUCAUAUUGCUGCUGCAGCAUCAACAAUUGCUGGCAGGCGGGCCCACCGAACAGCAAU ...(((.(((.....((((..(...((((...(......)..))))....)..))))..))).))).(((((((((((((((........)))).)))).(((.....)))..))))))) ( -36.60) >consensus CACUGAUCGCCCACUGCAGCUGAAGUGCACCAGGAAUAACGAUGCCACCGCUGCUGCUCGCGUUCAUAUUGCUGCUGCAGCAGCAACAAUUGCUGGCAGGCAGCGCGACCGACCAGCAAU ...(((.(((.....(((((.(..(((((...(......)..))).))..).)))))..))).)))..(((((((....)))))))..((((((((..((........))..)))))))) (-34.76 = -35.40 + 0.64)

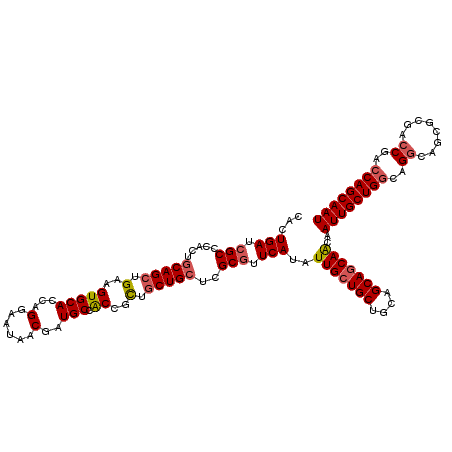
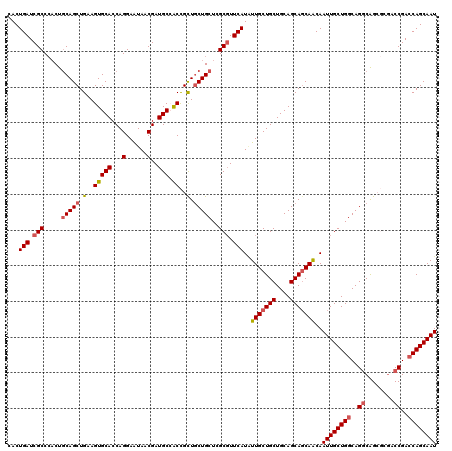
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:49:57 2006