
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 4,033,008 – 4,033,167 |
| Length | 159 |
| Max. P | 0.999893 |

| Location | 4,033,008 – 4,033,128 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.83 |
| Mean single sequence MFE | -57.16 |
| Consensus MFE | -55.20 |
| Energy contribution | -55.52 |
| Covariance contribution | 0.32 |
| Combinations/Pair | 1.04 |
| Mean z-score | -2.86 |
| Structure conservation index | 0.97 |
| SVM decision value | 4.42 |
| SVM RNA-class probability | 0.999893 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
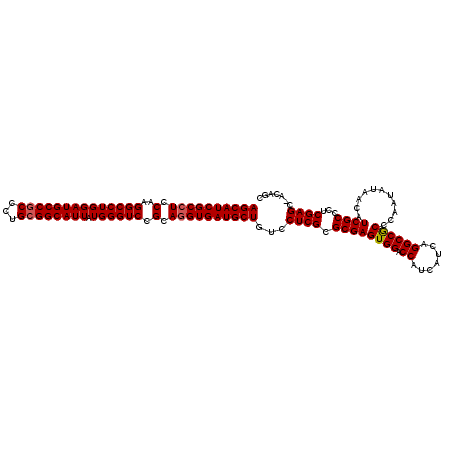
>2R_DroMel_CAF1 4033008 120 + 20766785 CGGCAGCAGCUGUUGCUUGUUCACCUGGUCCAAGGUGUAUAGCAUCGCCUCCAAGGCCUGGAUGCCGCCCUGCGGCAUUAUGGGUCCGCAAGUGAUGCUGUCCUCGCGCGAGUGGACCAU .((((((....))))))........(((((((..((((((((((((((......((((((((((((((...)))))))).)))))).....))))))))))....))))...))))))). ( -53.70) >DroSec_CAF1 15659 120 + 1 CGGCAGGAGCUGUUGCUUGUUCACCUGGUCCAAGGUGUACAGCAUCGCCUCCAAGGCCUGGAUGCCGCCCUGCGGCAUUAUGGGUCCGCAGGUGAUGCUGUCCUCGCGCGAGUGGACCAU .((...((((........)))).))(((((((..((((((((((((((((.(..((((((((((((((...)))))))).)))))).).))))))))))))....))))...))))))). ( -59.00) >DroSim_CAF1 17802 120 + 1 CGGCAGGAGCUGUUGCUUGUUCACCUGGUCCAAGGUGUACAGCAUCGCCUCCAAGGCCUGGAUGCCGCCCUGCGGCAUUAUGGGUCCGCAGGUGAUGCUGUCCUCGCGCGAGUGGACCAU .((...((((........)))).))(((((((..((((((((((((((((.(..((((((((((((((...)))))))).)))))).).))))))))))))....))))...))))))). ( -59.00) >DroEre_CAF1 15010 120 + 1 CGGCAGGAGCUGUUGCUUGUUUACCUGGUCCAAGGUGUACAGCAUCGCCUCCAAGGCCUGGAUGCCGCCCUGGGGCAUUAUGGGUCCGCAGGUGAUGCUGUCCUCGCGCGAGUGGACCAU .((.(.((((....)))).)...))(((((((..((((((((((((((((.(..((((((((((((.(....))))))).)))))).).))))))))))))....))))...))))))). ( -55.60) >DroYak_CAF1 18743 120 + 1 CGGCAGAAGCUGUUGCUUGUUAACCUGGUCCAAGGUGUACAGCAUCGCCUCCAAGGCCUGGAUGCCGCCCUGCGGCAUUAUGGGUCCGCAGGUGAUGCUGUCCUCGCGCGAGUGGACCAU .((.(.((((....)))).)...))(((((((..((((((((((((((((.(..((((((((((((((...)))))))).)))))).).))))))))))))....))))...))))))). ( -58.50) >consensus CGGCAGGAGCUGUUGCUUGUUCACCUGGUCCAAGGUGUACAGCAUCGCCUCCAAGGCCUGGAUGCCGCCCUGCGGCAUUAUGGGUCCGCAGGUGAUGCUGUCCUCGCGCGAGUGGACCAU .((.(.((((....)))).)...))(((((((..((((((((((((((((.(..((((((((((((((...)))))))).)))))).).))))))))))))....))))...))))))). (-55.20 = -55.52 + 0.32)


| Location | 4,033,008 – 4,033,128 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.83 |
| Mean single sequence MFE | -51.14 |
| Consensus MFE | -49.92 |
| Energy contribution | -50.24 |
| Covariance contribution | 0.32 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.44 |
| Structure conservation index | 0.98 |
| SVM decision value | 3.96 |
| SVM RNA-class probability | 0.999729 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 4033008 120 - 20766785 AUGGUCCACUCGCGCGAGGACAGCAUCACUUGCGGACCCAUAAUGCCGCAGGGCGGCAUCCAGGCCUUGGAGGCGAUGCUAUACACCUUGGACCAGGUGAACAAGCAACAGCUGCUGCCG .((((((((((....)))...((((((.(((.(((..((...(((((((...)))))))...))..))).))).))))))........)))))))((((....(((....)))..)))). ( -46.30) >DroSec_CAF1 15659 120 - 1 AUGGUCCACUCGCGCGAGGACAGCAUCACCUGCGGACCCAUAAUGCCGCAGGGCGGCAUCCAGGCCUUGGAGGCGAUGCUGUACACCUUGGACCAGGUGAACAAGCAACAGCUCCUGCCG .((((((((((....))).((((((((.(((.(((..((...(((((((...)))))))...))..))).))).))))))))......)))))))((((....(((....)))..)))). ( -52.30) >DroSim_CAF1 17802 120 - 1 AUGGUCCACUCGCGCGAGGACAGCAUCACCUGCGGACCCAUAAUGCCGCAGGGCGGCAUCCAGGCCUUGGAGGCGAUGCUGUACACCUUGGACCAGGUGAACAAGCAACAGCUCCUGCCG .((((((((((....))).((((((((.(((.(((..((...(((((((...)))))))...))..))).))).))))))))......)))))))((((....(((....)))..)))). ( -52.30) >DroEre_CAF1 15010 120 - 1 AUGGUCCACUCGCGCGAGGACAGCAUCACCUGCGGACCCAUAAUGCCCCAGGGCGGCAUCCAGGCCUUGGAGGCGAUGCUGUACACCUUGGACCAGGUAAACAAGCAACAGCUCCUGCCG .((((((((((....))).((((((((......(....).....((((((((((.........))))))).)))))))))))......)))))))((((....(((....)))..)))). ( -50.60) >DroYak_CAF1 18743 120 - 1 AUGGUCCACUCGCGCGAGGACAGCAUCACCUGCGGACCCAUAAUGCCGCAGGGCGGCAUCCAGGCCUUGGAGGCGAUGCUGUACACCUUGGACCAGGUUAACAAGCAACAGCUUCUGCCG .((((((((((....))).((((((((.(((.(((..((...(((((((...)))))))...))..))).))).))))))))......)))))))(((....((((....))))..))). ( -54.20) >consensus AUGGUCCACUCGCGCGAGGACAGCAUCACCUGCGGACCCAUAAUGCCGCAGGGCGGCAUCCAGGCCUUGGAGGCGAUGCUGUACACCUUGGACCAGGUGAACAAGCAACAGCUCCUGCCG .((((((((((....))).((((((((.(((.(((..((...(((((((...)))))))...))..))).))).))))))))......)))))))((((....(((....)))..)))). (-49.92 = -50.24 + 0.32)



| Location | 4,033,048 – 4,033,167 |
|---|---|
| Length | 119 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.06 |
| Mean single sequence MFE | -48.38 |
| Consensus MFE | -45.24 |
| Energy contribution | -45.48 |
| Covariance contribution | 0.24 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.76 |
| Structure conservation index | 0.94 |
| SVM decision value | 2.46 |
| SVM RNA-class probability | 0.994230 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 4033048 119 + 20766785 AGCAUCGCCUCCAAGGCCUGGAUGCCGCCCUGCGGCAUUAUGGGUCCGCAAGUGAUGCUGUCCUCGCGCGAGUGGACCAUCAUCAGGCCGCCCAAUAUAACAUCGCCCUCGAGC-ACAGC ((((((((......((((((((((((((...)))))))).)))))).....))))))))((.((((.((((((((.((.......))))))...........))))...)))).-))... ( -44.60) >DroSec_CAF1 15699 119 + 1 AGCAUCGCCUCCAAGGCCUGGAUGCCGCCCUGCGGCAUUAUGGGUCCGCAGGUGAUGCUGUCCUCGCGCGAGUGGACCAUCAUCAGGCCGCCCAAUAUAACAUCGCCCUCGAGC-ACAGC ((((((((((.(..((((((((((((((...)))))))).)))))).).))))))))))((.((((.((((((((.((.......))))))...........))))...)))).-))... ( -49.70) >DroSim_CAF1 17842 120 + 1 AGCAUCGCCUCCAAGGCCUGGAUGCCGCCCUGCGGCAUUAUGGGUCCGCAGGUGAUGCUGUCCUCGCGCGAGUGGACCAUCAUCAGGCCGCCUAAUAUAACAUCGCCCUCGAGUUAUUUU ((((((((((.(..((((((((((((((...)))))))).)))))).).))))))))))...((((.((((((((.((.......))))))...........))))...))))....... ( -47.80) >DroEre_CAF1 15050 119 + 1 AGCAUCGCCUCCAAGGCCUGGAUGCCGCCCUGGGGCAUUAUGGGUCCGCAGGUGAUGCUGUCCUCGCGCGAGUGGACCAUCAUCAGGCCGCCCAGGAUGACAUCGCCCUCGAGC-ACAGC ((((((((((.(..((((((((((((.(....))))))).)))))).).))))))))))......((.((((.((....(((((.((....))..))))).....)))))).))-..... ( -48.50) >DroYak_CAF1 18783 119 + 1 AGCAUCGCCUCCAAGGCCUGGAUGCCGCCCUGCGGCAUUAUGGGUCCGCAGGUGAUGCUGUCCUCGCGCGAGUGGACCAUCAUCAGGCCACCCAGGAUUACAUCGCCCUCGAGC-ACAGC ((((((((((.(..((((((((((((((...)))))))).)))))).).))))))))))((.((((.((((((((.((.......))))))...(.....).))))...)))).-))... ( -51.30) >consensus AGCAUCGCCUCCAAGGCCUGGAUGCCGCCCUGCGGCAUUAUGGGUCCGCAGGUGAUGCUGUCCUCGCGCGAGUGGACCAUCAUCAGGCCGCCCAAUAUAACAUCGCCCUCGAGC_ACAGC ((((((((((.(..((((((((((((((...)))))))).)))))).).))))))))))...((((.((((((((.((.......))))))...........))))...))))....... (-45.24 = -45.48 + 0.24)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:49:48 2006