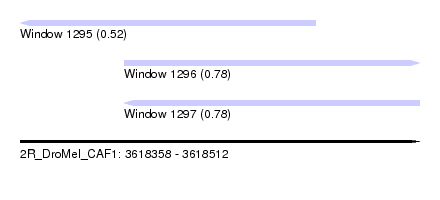
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 3,618,358 – 3,618,512 |
| Length | 154 |
| Max. P | 0.780039 |

| Location | 3,618,358 – 3,618,472 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.70 |
| Mean single sequence MFE | -36.40 |
| Consensus MFE | -30.52 |
| Energy contribution | -30.25 |
| Covariance contribution | -0.27 |
| Combinations/Pair | 1.37 |
| Mean z-score | -1.30 |
| Structure conservation index | 0.84 |
| SVM decision value | -0.02 |
| SVM RNA-class probability | 0.524824 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
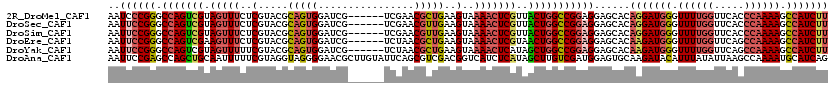
>2R_DroMel_CAF1 3618358 114 - 20766785 ------UCGAACGCUGAAGUAAAACUCGUUACUGGCCGGAGGAGCACAGGAUGGGUUUUGGUUCACCCAAAAGCCAUCUUCGGGACUGGUGGCGAUCUCAAGCGUGCGUUUCAAAACAUU ------.((.(((((..((......(((((((..(((.(((.....).((((((.((((((.....)))))).)))))))).)).)..))))))).))..))))).))............ ( -40.10) >DroSec_CAF1 5939 114 - 1 ------UCGAACGUUGAAGUAAAACUCGUUACUGGCCGGAGGAGCACAGGAUGGGUUUUGGUUCACCCAAAAGCCAUCUUCGGGACUGGUGGCGAUCUCAAGCGUGCGUUUCAAAACAUU ------.((.(((((..((......(((((((..(((.(((.....).((((((.((((((.....)))))).)))))))).)).)..))))))).))..))))).))............ ( -37.70) >DroSim_CAF1 3593 114 - 1 ------UCGAACGUUGAAGUAAAACUCGUUACUGGCCGGAGGAGCACAGGAUGGGUUUUGGUUCACCCAAAAGCCAUCUUCGGGACUGGUGGCGAUCUCAAGCGUGCGUUUCAAAACAUU ------.((.(((((..((......(((((((..(((.(((.....).((((((.((((((.....)))))).)))))))).)).)..))))))).))..))))).))............ ( -37.70) >DroEre_CAF1 7384 114 - 1 ------UCUAACGCUGAAGUAAAACUCGUAACUGGCCGGAGGAGCACAAGAUGGGUUUUGGUUCAGCCAAAAGCCAUCUUCGGGACUGGUGGCGAUUUCAAGCGUGCGUUUCAGAACAUU ------....(((((((((......((((.((..(((.(((.....).((((((.((((((.....)))))).)))))))).)).)..)).)))))))).)))))............... ( -37.30) >DroYak_CAF1 5352 114 - 1 ------UCUAACGCUGAAGUAAAACUCAUAGCUGGCCGGAGGAGCACAAGAUGGGUUUUGGUUCAGCCAAAAGCCAUCUUCGGGACUGGUGGCGAUUUCAAGCGUGCGUUUCAAGACGUU ------....((((((((((..........((((.((((.(.....)(((((((.((((((.....)))))).))))))).....)))))))).))))).)))))(((((....))))). ( -37.20) >DroAna_CAF1 7333 120 - 1 CUUGUAUUCAGCGUCGACGGUCAUCUCAUAGCUUGUCGAUGGAGUGCAAGAUACAUUUAUAUUAAGCCAAAAUGCAUCAGUGGGAUCGGUUGCGAUUGUCAGAGUUCGCUGCAUAACAUU (((((((((..(((((((((.(........).)))))))))))))))))).....................(((((...(((((((((....)))))........)))))))))...... ( -28.40) >consensus ______UCGAACGCUGAAGUAAAACUCGUAACUGGCCGGAGGAGCACAAGAUGGGUUUUGGUUCACCCAAAAGCCAUCUUCGGGACUGGUGGCGAUCUCAAGCGUGCGUUUCAAAACAUU ........((((((...........(((((((..(((.((........((((((.((((((.....)))))).)))))))).)).)..)))))))..........))))))......... (-30.52 = -30.25 + -0.27)


| Location | 3,618,398 – 3,618,512 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 83.91 |
| Mean single sequence MFE | -35.67 |
| Consensus MFE | -29.39 |
| Energy contribution | -28.89 |
| Covariance contribution | -0.49 |
| Combinations/Pair | 1.35 |
| Mean z-score | -1.93 |
| Structure conservation index | 0.82 |
| SVM decision value | 0.55 |
| SVM RNA-class probability | 0.777599 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 3618398 114 + 20766785 AAGAUGGCUUUUGGGUGAACCAAAACCCAUCCUGUGCUCCUCCGGCCAGUAACGAGUUUUACUUCAGCGUUCGA------CGAUCCACUGCGUACGAGAAACUACGACUGGCCCGGGAUU ..(((((.((((((.....)))))).)))))......(((.(.(((((((....((((((((......))((((------((........))).)))))))))...))))))).)))).. ( -38.70) >DroSec_CAF1 5979 114 + 1 AAGAUGGCUUUUGGGUGAACCAAAACCCAUCCUGUGCUCCUCCGGCCAGUAACGAGUUUUACUUCAACGUUCGA------CGAUCCACUGCGUACGAGAAACUACGACUGGCCCGGAAUU ..(((((.((((((.....)))))).))))).........((((((((((....((((((((......))((((------((........))).)))))))))...))))).)))))... ( -38.40) >DroSim_CAF1 3633 114 + 1 AAGAUGGCUUUUGGGUGAACCAAAACCCAUCCUGUGCUCCUCCGGCCAGUAACGAGUUUUACUUCAACGUUCGA------CGAUCCACUGCGUACGAGAAACUACGACUGGCCCGGAAUU ..(((((.((((((.....)))))).))))).........((((((((((....((((((((......))((((------((........))).)))))))))...))))).)))))... ( -38.40) >DroEre_CAF1 7424 114 + 1 AAGAUGGCUUUUGGCUGAACCAAAACCCAUCUUGUGCUCCUCCGGCCAGUUACGAGUUUUACUUCAGCGUUAGA------CGAUCCACUGCGUACGAGAAACUUCGACUGGCCCGGAAUU (((((((.((((((.....)))))).))))))).......(((((((((((..((((((..(((..((((.((.------.......))))))..))))))))).)))))).)))))... ( -39.60) >DroYak_CAF1 5392 114 + 1 AAGAUGGCUUUUGGCUGAACCAAAACCCAUCUUGUGCUCCUCCGGCCAGCUAUGAGUUUUACUUCAGCGUUAGA------CGAUCCACUGCGUACGAAAAACUACGACUGGCCCGGAAUU (((((((.((((((.....)))))).))))))).......(((((((((.....((((((.(....((((.((.------.......))))))..).))))))....)))).)))))... ( -35.40) >DroAna_CAF1 7373 120 + 1 CUGAUGCAUUUUGGCUUAAUAUAAAUGUAUCUUGCACUCCAUCGACAAGCUAUGAGAUGACCGUCGACGCUGAAUACAAGCGUUCCCCUACCUACGAAAAAUUGCAGCUGGCUCGGAAUU ..(((((((((...........)))))))))..........((((..((((..(((((....)))((((((.......)))))).................))..))))...)))).... ( -23.50) >consensus AAGAUGGCUUUUGGCUGAACCAAAACCCAUCCUGUGCUCCUCCGGCCAGUAACGAGUUUUACUUCAACGUUCGA______CGAUCCACUGCGUACGAGAAACUACGACUGGCCCGGAAUU ..(((((.((((((.....)))))).))))).........((((((((((..((((((((.(....((((...................))))..).)))))).))))))).)))))... (-29.39 = -28.89 + -0.49)

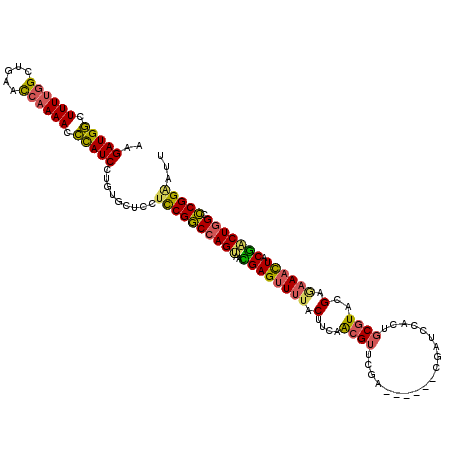
| Location | 3,618,398 – 3,618,512 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 83.91 |
| Mean single sequence MFE | -40.65 |
| Consensus MFE | -31.10 |
| Energy contribution | -32.07 |
| Covariance contribution | 0.98 |
| Combinations/Pair | 1.22 |
| Mean z-score | -2.33 |
| Structure conservation index | 0.76 |
| SVM decision value | 0.55 |
| SVM RNA-class probability | 0.780039 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 3618398 114 - 20766785 AAUCCCGGGCCAGUCGUAGUUUCUCGUACGCAGUGGAUCG------UCGAACGCUGAAGUAAAACUCGUUACUGGCCGGAGGAGCACAGGAUGGGUUUUGGUUCACCCAAAAGCCAUCUU ..((((.(((((((((.(((((..(.....(((((.....------.....)))))..)..)))))))..))))))).).)))....(((((((.((((((.....)))))).))))))) ( -45.10) >DroSec_CAF1 5979 114 - 1 AAUUCCGGGCCAGUCGUAGUUUCUCGUACGCAGUGGAUCG------UCGAACGUUGAAGUAAAACUCGUUACUGGCCGGAGGAGCACAGGAUGGGUUUUGGUUCACCCAAAAGCCAUCUU ..((((((.(((((((.(((((.(((.(((........))------))))((......)).)))))))..)))))))))))......(((((((.((((((.....)))))).))))))) ( -39.70) >DroSim_CAF1 3633 114 - 1 AAUUCCGGGCCAGUCGUAGUUUCUCGUACGCAGUGGAUCG------UCGAACGUUGAAGUAAAACUCGUUACUGGCCGGAGGAGCACAGGAUGGGUUUUGGUUCACCCAAAAGCCAUCUU ..((((((.(((((((.(((((.(((.(((........))------))))((......)).)))))))..)))))))))))......(((((((.((((((.....)))))).))))))) ( -39.70) >DroEre_CAF1 7424 114 - 1 AAUUCCGGGCCAGUCGAAGUUUCUCGUACGCAGUGGAUCG------UCUAACGCUGAAGUAAAACUCGUAACUGGCCGGAGGAGCACAAGAUGGGUUUUGGUUCAGCCAAAAGCCAUCUU ..((((((.((((((((.((((..(.....(((((.....------.....)))))..)..)))))))..)))))))))))......(((((((.((((((.....)))))).))))))) ( -44.90) >DroYak_CAF1 5392 114 - 1 AAUUCCGGGCCAGUCGUAGUUUUUCGUACGCAGUGGAUCG------UCUAACGCUGAAGUAAAACUCAUAGCUGGCCGGAGGAGCACAAGAUGGGUUUUGGUUCAGCCAAAAGCCAUCUU ..((((((.(((((..(((((((.(.....(((((.....------.....)))))..).)))))).)..)))))))))))......(((((((.((((((.....)))))).))))))) ( -45.60) >DroAna_CAF1 7373 120 - 1 AAUUCCGAGCCAGCUGCAAUUUUUCGUAGGUAGGGGAACGCUUGUAUUCAGCGUCGACGGUCAUCUCAUAGCUUGUCGAUGGAGUGCAAGAUACAUUUAUAUUAAGCCAAAAUGCAUCAG ..((((...(((.((((........)))).).))))))..(((((((((..(((((((((.(........).))))))))))))))))))...............((......))..... ( -28.90) >consensus AAUUCCGGGCCAGUCGUAGUUUCUCGUACGCAGUGGAUCG______UCGAACGCUGAAGUAAAACUCGUAACUGGCCGGAGGAGCACAAGAUGGGUUUUGGUUCACCCAAAAGCCAUCUU ..((((((.(((((((.(((((..(.....(((((................)))))..)..)))))))..)))))))))))......(((((((.((((((.....)))))).))))))) (-31.10 = -32.07 + 0.98)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:46:01 2006