
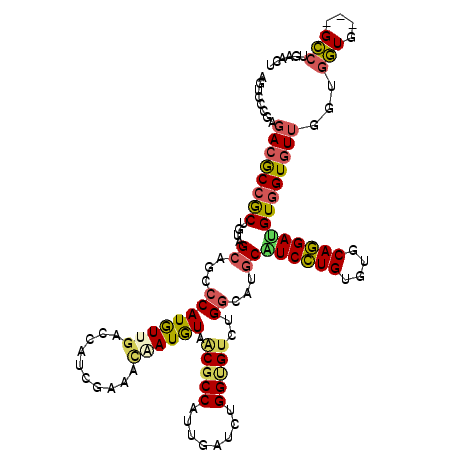
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 20,692,941 – 20,693,098 |
| Length | 157 |
| Max. P | 0.914436 |

| Location | 20,692,941 – 20,693,058 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 79.77 |
| Mean single sequence MFE | -42.88 |
| Consensus MFE | -25.97 |
| Energy contribution | -25.95 |
| Covariance contribution | -0.02 |
| Combinations/Pair | 1.38 |
| Mean z-score | -1.52 |
| Structure conservation index | 0.61 |
| SVM decision value | -0.01 |
| SVM RNA-class probability | 0.527410 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
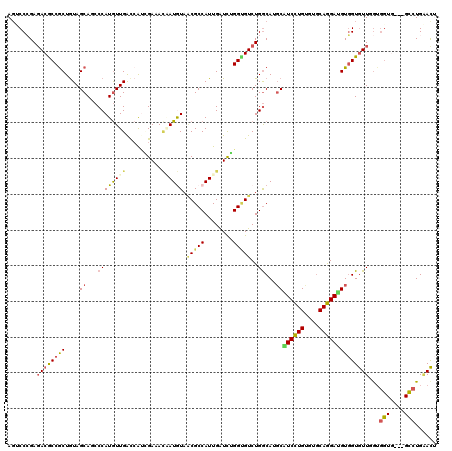
>2R_DroMel_CAF1 20692941 117 + 20766785 AUACCCGAGACACCGCUGUAGCAUCCCAUGUUGACCAUUGACAUUAUAUAACGCCAUUGAUCAGGGGUCUGGCAAGCAUCCUGUAUACAGGACGUGGUGUUUGAUGUA---GCUUGGACU ....((((((((((((....((.....((((..(...)..))))........((((..(((.....)))))))..)).(((((....))))).)))))))........---.)))))... ( -33.30) >DroVir_CAF1 224552 120 + 1 AGGCCAGAGACGCCGCUGUAGCAGCCCAUGUUGACGGACGAAACGAUGUAGCGCCAUUGAUCGGGCGUCUGGCAGGCAUCCUGUGUGCAGGAUGUGGUGUUGAUGCUGCCCGGCUGGACG .((((..(((((((.((((((((.....)))).)))).(((..(((((......))))).))))))))))(((((((((((((....))))))))..........))))).))))..... ( -48.80) >DroPse_CAF1 131608 117 + 1 AGUCCCGAGACGCCGCUGUAGCAGCCCAUGUUGGUCAUAGAAACAAUGUAACGCCAUUGAUCUGGUGUCUGGCAUGCGUCCUGUGUGCAGGAUGUGGUGUUGGUGGUG---GCCUGAACU .(((....)))(((((..(..((((((((((..(.((((((..(((((......))))).))).))).)..))))((((((((....)))))))))).)))))..)))---))....... ( -44.90) >DroWil_CAF1 216336 117 + 1 AGUCCAGAGACACCACUAUAGCAUCCCAUAUUGACCACCGAUAAUAUAUAACGCCAUUGAUCUGGUGUCUGGCAUUCAUCUUGAGUGCAAGAUGUUGUAUUUGUGGUA---GCCUGAAUC ....(((.(..((((((((((((((...(((((.....))))).......((((((......))))))...((((((.....))))))..)))))))))...))))).---.)))).... ( -34.60) >DroAna_CAF1 134420 117 + 1 AGGCCCGACACGCCGCUGUAGCAUCCCAUGUUGGCCAUCGACACAAUGUACCGCCAUUGGUCGGGCGUCUGACAGGCGUCCUGGUUGCAGGACGUGGUGUUCGUGGUG---GCCUGUACU ((((((.(((((((((.........((.(((..(.(.(((((.(((((......))))))))))..).)..))))).((((((....)))))))))))))..)).).)---))))..... ( -49.90) >DroPer_CAF1 142461 117 + 1 AGUCCCGAGACGCCGCUGUAGCAGCCCAUGUUGGUCAUAGAAACAAUGUAACGCCAUUGAUUUGGUGUCUGGCAUGCGUCCUGUGUGCAGGAUGUGGUGUUGGUGGUG---GCCUGAACU .(((....)))(((((..(..((((((((((..(.(((.(((.(((((......))))).))).))).)..))))((((((((....)))))))))).)))))..)))---))....... ( -45.80) >consensus AGUCCCGAGACGCCGCUGUAGCAGCCCAUGUUGACCAUCGAAACAAUGUAACGCCAUUGAUCUGGUGUCUGGCAUGCAUCCUGUGUGCAGGAUGUGGUGUUGGUGGUG___GCCUGAACU ........((((((((....((...((((((((..........)))))).(((((........)))))..))...))((((((....))))))))))))))...(((....)))...... (-25.97 = -25.95 + -0.02)

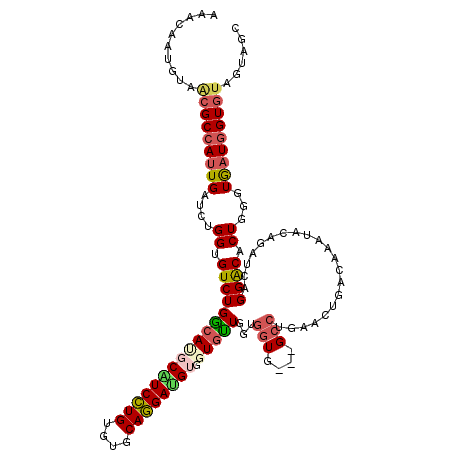
| Location | 20,692,981 – 20,693,098 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.59 |
| Mean single sequence MFE | -41.65 |
| Consensus MFE | -24.83 |
| Energy contribution | -25.45 |
| Covariance contribution | 0.62 |
| Combinations/Pair | 1.29 |
| Mean z-score | -2.18 |
| Structure conservation index | 0.60 |
| SVM decision value | 1.10 |
| SVM RNA-class probability | 0.914436 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
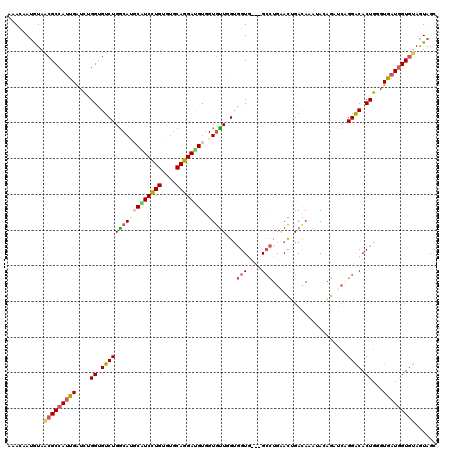
>2R_DroMel_CAF1 20692981 117 + 20766785 ACAUUAUAUAACGCCAUUGAUCAGGGGUCUGGCAAGCAUCCUGUAUACAGGACGUGGUGUUUGAUGUA---GCUUGGACUGACAAAUACAAGUCUGGACACUGGGUGAUAGUAUAGUAGC ..(((((((..((((......(((..(((..((..((.(((((....))))).)).(((((((...((---(......))).)))))))..).)..))).)))))))...)))))))... ( -31.70) >DroPse_CAF1 131648 117 + 1 AAACAAUGUAACGCCAUUGAUCUGGUGUCUGGCAUGCGUCCUGUGUGCAGGAUGUGGUGUUGGUGGUG---GCCUGAACUGACAGAAACAGAUCAGGACACUGUGUGAUGGUGUAGUAGC ..........((((((((..........(..((((((((((((....)))))))).))))..).((((---.(((((.(((.......))).))))).))))....))))))))...... ( -42.70) >DroGri_CAF1 229628 120 + 1 AGACGACGUAGCGCCACUGAUCGGGCGUCUGGCACUCAUCCUGGGUGCAGGAUGUGGUGCUGGUGGUGCCCGGCUGGACGGACAAAUAGAGCGGAGGACACUGGGUGAUGGUGUAGUAGC ..........(((((((((..((((((.(..((((((((((((....))))))).)))))..)...))))))..))).....((..(((.((....).).)))..)).))))))...... ( -45.60) >DroWil_CAF1 216376 117 + 1 AUAAUAUAUAACGCCAUUGAUCUGGUGUCUGGCAUUCAUCUUGAGUGCAAGAUGUUGUAUUUGUGGUA---GCCUGAAUCGACAAAAAUAGAUCCGGACACUGGGUAAUGGUGUAAUAGC ......(((.((((((((.(((..((((((((....(((((((....)))))))((((.(((((.(..---........).))))).))))..))))))))..))))))))))).))).. ( -42.00) >DroAna_CAF1 134460 117 + 1 ACACAAUGUACCGCCAUUGGUCGGGCGUCUGACAGGCGUCCUGGUUGCAGGACGUGGUGUUCGUGGUG---GCCUGUACUGAUACGUAAAGGUCGGGGCACUGUGUAAUGGUGUAGUAGC (((((.((..(((((((..(((((....)))))..((((((((....)))))))).......)))))(---((((.(((......))).)))))))..)).))))).............. ( -44.60) >DroPer_CAF1 142501 117 + 1 AAACAAUGUAACGCCAUUGAUUUGGUGUCUGGCAUGCGUCCUGUGUGCAGGAUGUGGUGUUGGUGGUG---GCCUGAACUGACAGAUACAGAUCAGGACACUGUGUGAUGGUGUAGUAGC ..........(((((((((...(((((((..((((((((((((....)))))))).))))..).....---.(((((.(((.......))).)))))))))))..)))))))))...... ( -43.30) >consensus AAACAAUGUAACGCCAUUGAUCUGGUGUCUGGCAUGCAUCCUGUGUGCAGGAUGUGGUGUUGGUGGUG___GCCUGAACUGACAAAUACAGAUCAGGACACUGGGUGAUGGUGUAGUAGC ..........(((((((((....((.(((((((((((((((((....)))))))).)))))...(((....))).....................)))).))...)))))))))...... (-24.83 = -25.45 + 0.62)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:14:52 2006