
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 20,574,166 – 20,574,291 |
| Length | 125 |
| Max. P | 0.947568 |

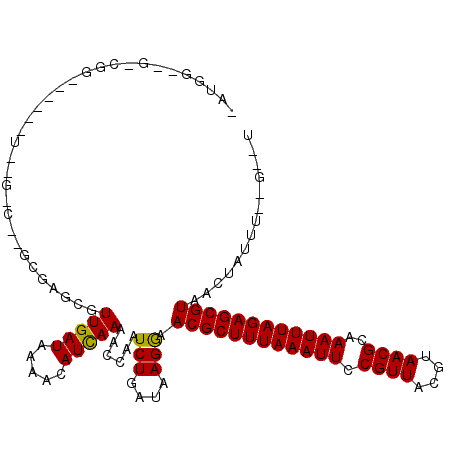
| Location | 20,574,166 – 20,574,273 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 76.79 |
| Mean single sequence MFE | -25.60 |
| Consensus MFE | -16.56 |
| Energy contribution | -16.28 |
| Covariance contribution | -0.28 |
| Combinations/Pair | 1.08 |
| Mean z-score | -1.65 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.88 |
| SVM RNA-class probability | 0.874069 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 20574166 107 + 20766785 CAUGAGAG-CGG------UAAG-C-ACCAAGCGUUGAUAAAACAUUAAAACCAAUCUGAUAAGAAACGCUUUAAAUUCCGUUACGUAACGCAAAUUUAGAGCGUGUCUGUGU--GGAU .((.(((.-.((------(..(-(-.....))(((.....)))......)))..))).)).(((.((((((((((((.((((....))))..)))))))))))).)))....--.... ( -26.70) >DroVir_CAF1 38836 99 + 1 UAUGGGCG-CGU------U-------GCGAGCGUUGAUAAAGCAUCAAGACCAAUCUGAUAAGGAACGCUUUAAAUUCCGUUACGUAACGCAAAUUUAGAGCGUUACGAUUC--G--- ..((((((-(..------.-------....))))((((.....))))...)))....((...(.(((((((((((((.((((....))))..))))))))))))).)...))--.--- ( -26.10) >DroWil_CAF1 39556 101 + 1 -ACGGAAGUUGG------UAGA-U-AUUCGGCGUUGAUAAAACAUCAAAACCAAUCUGAUAAGGAACGCUUUAAAUUCCGUUACGUAACGCAAAUUUAGAGCGUAACA--------AU -((....))...------((((-(-....((..(((((.....)))))..)).)))))....(..((((((((((((.((((....))))..))))))))))))..).--------.. ( -24.70) >DroMoj_CAF1 42903 90 + 1 UUUGG-----------------------GAGCGUUGAUAAAGCAUCAAAACCAAUCUGAUAAGGAACGCUUUAAAUUCCGUUACGUAACGCAAAUUUAGAGCGUUACAAUUU--G--- .((((-----------------------.....(((((.....)))))..))))........(.(((((((((((((.((((....))))..))))))))))))).).....--.--- ( -21.80) >DroAna_CAF1 26289 101 + 1 CAUGA--G-CGG---------G-C-UGCGAGCGUUGAUAAAACAUCAAAACCAAUCUGAUAAGGAACGCUUUAAAUUCCGUUACGUAACGCAAAUUUAGAGCGUAUCUCU-U--GGUU ((.((--.-.((---------(-(-.....)).(((((.....)))))..))..)))).((((((((((((((((((.((((....))))..)))))))))))).)).))-)--)... ( -24.30) >DroPer_CAF1 28162 110 + 1 -------G-CGGCCGAGGGAAGGCGGGCGAGCGUUGAUAAAACAUCAAAACCAAUCUGAUAAGGAACGCUUUAAAUUCCGUUACGUAACGCAAAUUUAGAGCGUAUCUCUUUCUGCUU -------(-(((..((((((...((((......(((((.....)))))......)))).......((((((((((((.((((....))))..)))))))))))).)))))).)))).. ( -30.00) >consensus _AUGG__G_CGG______U__G_C__GCGAGCGUUGAUAAAACAUCAAAACCAAUCUGAUAAGGAACGCUUUAAAUUCCGUUACGUAACGCAAAUUUAGAGCGUAACUAUUU__G__U .................................(((((.....)))))......(((....))).((((((((((((.((((....))))..)))))))))))).............. (-16.56 = -16.28 + -0.28)

| Location | 20,574,166 – 20,574,273 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 76.79 |
| Mean single sequence MFE | -22.48 |
| Consensus MFE | -16.66 |
| Energy contribution | -16.68 |
| Covariance contribution | 0.03 |
| Combinations/Pair | 1.04 |
| Mean z-score | -1.06 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.41 |
| SVM RNA-class probability | 0.726638 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 20574166 107 - 20766785 AUCC--ACACAGACACGCUCUAAAUUUGCGUUACGUAACGGAAUUUAAAGCGUUUCUUAUCAGAUUGGUUUUAAUGUUUUAUCAACGCUUGGU-G-CUUA------CCG-CUCUCAUG ....--....(((.(((((.(((((((.((((....))))))))))).))))).)))(((.(((.((((......(((.....)))((.....-)-)..)------)))-.))).))) ( -21.20) >DroVir_CAF1 38836 99 - 1 ---C--GAAUCGUAACGCUCUAAAUUUGCGUUACGUAACGGAAUUUAAAGCGUUCCUUAUCAGAUUGGUCUUGAUGCUUUAUCAACGCUCGC-------A------ACG-CGCCCAUA ---.--....((((((((.........))))))))....((....(((((((((....(((.....)))...)))))))))....(((....-------.------..)-)).))... ( -23.40) >DroWil_CAF1 39556 101 - 1 AU--------UGUUACGCUCUAAAUUUGCGUUACGUAACGGAAUUUAAAGCGUUCCUUAUCAGAUUGGUUUUGAUGUUUUAUCAACGCCGAAU-A-UCUA------CCAACUUCCGU- ..--------.(..(((((.(((((((.((((....))))))))))).)))))..).....((((((((.((((((...)))))).)))))..-.-))).------...........- ( -23.00) >DroMoj_CAF1 42903 90 - 1 ---C--AAAUUGUAACGCUCUAAAUUUGCGUUACGUAACGGAAUUUAAAGCGUUCCUUAUCAGAUUGGUUUUGAUGCUUUAUCAACGCUC-----------------------CCAAA ---.--.....(((((((.........))))))).....(((...(((((((((....(((.....)))...))))))))).......))-----------------------).... ( -19.70) >DroAna_CAF1 26289 101 - 1 AACC--A-AGAGAUACGCUCUAAAUUUGCGUUACGUAACGGAAUUUAAAGCGUUCCUUAUCAGAUUGGUUUUGAUGUUUUAUCAACGCUCGCA-G-C---------CCG-C--UCAUG ....--.-.(((..(((((.(((((((.((((....))))))))))).)))))..)))....((..((((.(((((((.....)))).))).)-)-)---------)..-.--))... ( -22.20) >DroPer_CAF1 28162 110 - 1 AAGCAGAAAGAGAUACGCUCUAAAUUUGCGUUACGUAACGGAAUUUAAAGCGUUCCUUAUCAGAUUGGUUUUGAUGUUUUAUCAACGCUCGCCCGCCUUCCCUCGGCCG-C------- ..((.(((((.((.(((((.(((((((.((((....))))))))))).)))))))))).)).....(((.((((((...)))))).))).))..(((.......)))..-.------- ( -25.40) >consensus A__C__AAAGAGAUACGCUCUAAAUUUGCGUUACGUAACGGAAUUUAAAGCGUUCCUUAUCAGAUUGGUUUUGAUGUUUUAUCAACGCUCGC__G_C__A______CCG_C__CCAU_ ...........(..(((((.(((((((.((((....))))))))))).)))))..)..........(((.((((((...)))))).)))............................. (-16.66 = -16.68 + 0.03)



| Location | 20,574,195 – 20,574,291 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 100 |
| Reading direction | forward |
| Mean pairwise identity | 78.97 |
| Mean single sequence MFE | -22.50 |
| Consensus MFE | -15.27 |
| Energy contribution | -15.52 |
| Covariance contribution | 0.25 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.26 |
| Structure conservation index | 0.68 |
| SVM decision value | 1.37 |
| SVM RNA-class probability | 0.947568 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 20574195 96 + 20766785 AAAACAUUAAAACCAAUCUGAUAAGAAACGCUUUAAAUUCCGUUACGUAACGCAAAUUUAGAGCGUGUCUGUGU--GGAUUCUGAGCGCCCUGUG--GUA ...........(((((((((...(((.((((((((((((.((((....))))..)))))))))))).)))...)--)))))....((.....)))--)). ( -23.80) >DroVir_CAF1 38860 93 + 1 AAAGCAUCAAGACCAAUCUGAUAAGGAACGCUUUAAAUUCCGUUACGUAACGCAAAUUUAGAGCGUUACGAUUC--G-----CGUUGGCCGUGUGCGACA ...((((...(.(((((.(((...(.(((((((((((((.((((....))))..))))))))))))).)...))--)-----.))))).)..)))).... ( -29.70) >DroPse_CAF1 28252 95 + 1 AAAACAUCAAAACCAAUCUGAUAAGGAACGCUUUAAAUUCCGUUACGUAACGCAAAUUUAGAGCGUAUCUCUUUCUGCUUUGUUUCGGCACUGUG----- ...(((.............((.(((((((((((((((((.((((....))))..)))))))))))).)).)))))((((.......)))).))).----- ( -19.50) >DroMoj_CAF1 42918 82 + 1 AAAGCAUCAAAACCAAUCUGAUAAGGAACGCUUUAAAUUCCGUUACGUAACGCAAAUUUAGAGCGUUACAAUUU--G-----CGU-----------GACA ...(((..........(((....)))(((((((((((((.((((....))))..)))))))))))))......)--)-----)..-----------.... ( -19.10) >DroAna_CAF1 26313 86 + 1 AAAACAUCAAAACCAAUCUGAUAAGGAACGCUUUAAAUUCCGUUACGUAACGCAAAUUUAGAGCGUAUCUCU-U--GGUUUCUGAG-----------GCA ......(((((((((((((.....)))((((((((((((.((((....))))..)))))))))))).....)-)--))))).))).-----------... ( -23.40) >DroPer_CAF1 28192 95 + 1 AAAACAUCAAAACCAAUCUGAUAAGGAACGCUUUAAAUUCCGUUACGUAACGCAAAUUUAGAGCGUAUCUCUUUCUGCUUUGUUUCGGCACUGUG----- ...(((.............((.(((((((((((((((((.((((....))))..)))))))))))).)).)))))((((.......)))).))).----- ( -19.50) >consensus AAAACAUCAAAACCAAUCUGAUAAGGAACGCUUUAAAUUCCGUUACGUAACGCAAAUUUAGAGCGUAUCUCUUU__G_UUU_UGUCGGC_CUGUG___CA ........................((.((((((((((((.((((....))))..)))))))))))).))............................... (-15.27 = -15.52 + 0.25)



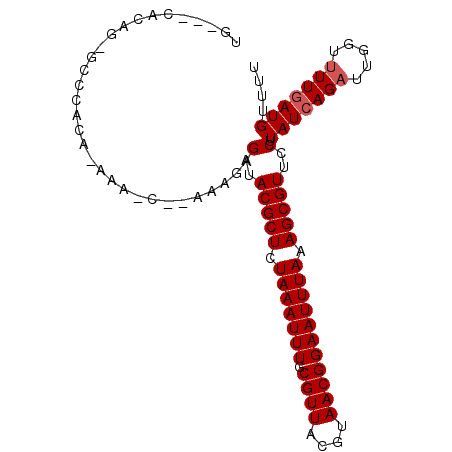
| Location | 20,574,195 – 20,574,291 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 100 |
| Reading direction | reverse |
| Mean pairwise identity | 78.97 |
| Mean single sequence MFE | -22.00 |
| Consensus MFE | -15.57 |
| Energy contribution | -15.90 |
| Covariance contribution | 0.33 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.55 |
| Structure conservation index | 0.71 |
| SVM decision value | 0.70 |
| SVM RNA-class probability | 0.826553 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 20574195 96 - 20766785 UAC--CACAGGGCGCUCAGAAUCC--ACACAGACACGCUCUAAAUUUGCGUUACGUAACGGAAUUUAAAGCGUUUCUUAUCAGAUUGGUUUUAAUGUUUU .((--((..(((..(...)..)))--....(((.(((((.(((((((.((((....))))))))))).))))).)))........))))........... ( -19.10) >DroVir_CAF1 38860 93 - 1 UGUCGCACACGGCCAACG-----C--GAAUCGUAACGCUCUAAAUUUGCGUUACGUAACGGAAUUUAAAGCGUUCCUUAUCAGAUUGGUCUUGAUGCUUU ....(((((.(((((((.-----.--((..((((((((.........))))))))....(((((.......)))))...)).).)))))).)).)))... ( -27.70) >DroPse_CAF1 28252 95 - 1 -----CACAGUGCCGAAACAAAGCAGAAAGAGAUACGCUCUAAAUUUGCGUUACGUAACGGAAUUUAAAGCGUUCCUUAUCAGAUUGGUUUUGAUGUUUU -----.........(((((..........(((..(((((.(((((((.((((....))))))))))).)))))..)))((((((.....))))))))))) ( -21.40) >DroMoj_CAF1 42918 82 - 1 UGUC-----------ACG-----C--AAAUUGUAACGCUCUAAAUUUGCGUUACGUAACGGAAUUUAAAGCGUUCCUUAUCAGAUUGGUUUUGAUGCUUU ....-----------..(-----(--.....(.((((((.(((((((.((((....))))))))))).)))))).)..((((((.....))))))))... ( -19.30) >DroAna_CAF1 26313 86 - 1 UGC-----------CUCAGAAACC--A-AGAGAUACGCUCUAAAUUUGCGUUACGUAACGGAAUUUAAAGCGUUCCUUAUCAGAUUGGUUUUGAUGUUUU .((-----------.((((((.((--(-((((..(((((.(((((((.((((....))))))))))).)))))..)))......)))))))))).))... ( -23.10) >DroPer_CAF1 28192 95 - 1 -----CACAGUGCCGAAACAAAGCAGAAAGAGAUACGCUCUAAAUUUGCGUUACGUAACGGAAUUUAAAGCGUUCCUUAUCAGAUUGGUUUUGAUGUUUU -----.........(((((..........(((..(((((.(((((((.((((....))))))))))).)))))..)))((((((.....))))))))))) ( -21.40) >consensus UG___CACAG_GCCCACA_AAA_C__AAAGAGAUACGCUCUAAAUUUGCGUUACGUAACGGAAUUUAAAGCGUUCCUUAUCAGAUUGGUUUUGAUGUUUU ...............................(..(((((.(((((((.((((....))))))))))).)))))..).(((((((.....))))))).... (-15.57 = -15.90 + 0.33)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:14:14 2006