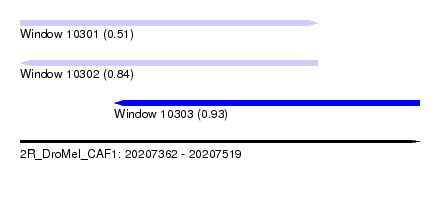
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 20,207,362 – 20,207,519 |
| Length | 157 |
| Max. P | 0.928562 |

| Location | 20,207,362 – 20,207,479 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 86.33 |
| Mean single sequence MFE | -51.82 |
| Consensus MFE | -42.60 |
| Energy contribution | -43.88 |
| Covariance contribution | 1.28 |
| Combinations/Pair | 1.10 |
| Mean z-score | -1.61 |
| Structure conservation index | 0.82 |
| SVM decision value | -0.05 |
| SVM RNA-class probability | 0.507339 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
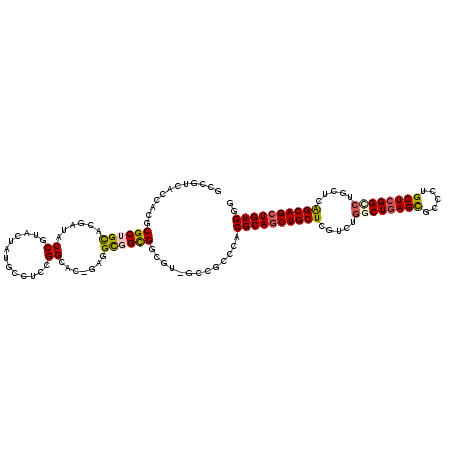
>2R_DroMel_CAF1 20207362 117 + 20766785 CACUGCUCCACUGACAGCUCCA---CCUCUGACGCGCCGCUCCACAGCUGCUGAGCAGGCCGAGCAGGGCGCUCAGCCAAACCAGCAGCUGCGUGGGCGGCUUGGCCACCGCUCCCGUCC ..((((......).))).....---.....((((.((((((((.(((((((((....(((.((((.....)))).)))....))))))))).).)))))))..(((....)))..)))). ( -50.50) >DroSec_CAF1 420 117 + 1 CACUGCUCCACUGACAGCUCCA---CCUCUGACGCGCCGCUCCACAGCUGCUGAGCAGGCCGAGCAGGGCGCUCAGCCAGACCAGCAACUGCGUGGGCGGCUUGGCCACCGCGCCCGUCC ..((((......).))).....---.....((((.((((((((.(((.(((((..(.(((.((((.....)))).))).)..))))).))).).)))))))..(((......))))))). ( -47.70) >DroSim_CAF1 1 101 + 1 ----------------GCUCCA---CAUCUGACGCGCCGCUCCACAGCUGCUGAGCAGGCCGAGCAGGGCGCUCAGCCAGACCAGCAGCUGCGUGGGCGGCUUGGCCACCGCGCCCGUCC ----------------......---.....((((.((((((((.(((((((((..(.(((.((((.....)))).))).)..))))))))).).)))))))..(((......))))))). ( -53.00) >DroEre_CAF1 441 117 + 1 CACCGCUCCACUGGCAGCUCCA---CCGCUGACGCGCCGCUCCACAGCUGCUGAGCAGGCCGAGCAGGGCGCUCAGCCAGACCAGCAGCUGCGUUGGCGGCUUGGCCACCGCACCCGUCC ....((.....(((((((....---..)))..((.((((((.(.(((((((((..(.(((.((((.....)))).))).)..))))))))).)..)))))).))))))..))........ ( -51.60) >DroYak_CAF1 459 117 + 1 CACCGCUCCACUGGCAGCUCCA---CCUCUGGCGCGCCGCUCCACAGCUGCUGAGCAGGCCGAGCAGGGCGCUCAGCCAGACCAGCAGCUGCGUCGGCGGCUUGGCCACCGCACCCGUCC ....((.....((((.((.(((---....))).))((((((.(.(((((((((..(.(((.((((.....)))).))).)..))))))))).)..))))))...))))..))........ ( -54.60) >DroAna_CAF1 153 120 + 1 CGCCCCACCACUGGCAGCUCCACCACCGCUGCCGGGCCGCCCCACAGCUGCCCAGUAGCCCGAGCAGAGCGCUCAGCCACACCAGCAGUUGCGUGGGCGGCAACGCCACCGCCUCCGUGC .....(((..((((((((.........))))))))((((((((.(((((((...((.((..((((.....)))).)).))....))))))).).)))))))...............))). ( -53.50) >consensus CACCGCUCCACUGGCAGCUCCA___CCUCUGACGCGCCGCUCCACAGCUGCUGAGCAGGCCGAGCAGGGCGCUCAGCCAGACCAGCAGCUGCGUGGGCGGCUUGGCCACCGCACCCGUCC ..............................((((.((((((((.(((((((((..(.(((.((((.....)))).))).)..))))))))).).)))))))...((....))...)))). (-42.60 = -43.88 + 1.28)



| Location | 20,207,362 – 20,207,479 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 86.33 |
| Mean single sequence MFE | -64.18 |
| Consensus MFE | -54.87 |
| Energy contribution | -56.05 |
| Covariance contribution | 1.18 |
| Combinations/Pair | 1.18 |
| Mean z-score | -2.01 |
| Structure conservation index | 0.85 |
| SVM decision value | 0.74 |
| SVM RNA-class probability | 0.837014 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 20207362 117 - 20766785 GGACGGGAGCGGUGGCCAAGCCGCCCACGCAGCUGCUGGUUUGGCUGAGCGCCCUGCUCGGCCUGCUCAGCAGCUGUGGAGCGGCGCGUCAGAGG---UGGAGCUGUCAGUGGAGCAGUG .((((.(.((((((((...))))))..(((((((((((((..((((((((.....)))))))).)).)))))))))))..))..).)))).....---....(((((.......))))). ( -62.70) >DroSec_CAF1 420 117 - 1 GGACGGGCGCGGUGGCCAAGCCGCCCACGCAGUUGCUGGUCUGGCUGAGCGCCCUGCUCGGCCUGCUCAGCAGCUGUGGAGCGGCGCGUCAGAGG---UGGAGCUGUCAGUGGAGCAGUG .(((((.(((((((((...))))))..(((((((((((..(.((((((((.....)))))))).)..)))))))))))..))).).)))).....---....(((((.......))))). ( -63.10) >DroSim_CAF1 1 101 - 1 GGACGGGCGCGGUGGCCAAGCCGCCCACGCAGCUGCUGGUCUGGCUGAGCGCCCUGCUCGGCCUGCUCAGCAGCUGUGGAGCGGCGCGUCAGAUG---UGGAGC---------------- .(((((.(((((((((...))))))..(((((((((((..(.((((((((.....)))))))).)..)))))))))))..))).).)))).....---......---------------- ( -61.50) >DroEre_CAF1 441 117 - 1 GGACGGGUGCGGUGGCCAAGCCGCCAACGCAGCUGCUGGUCUGGCUGAGCGCCCUGCUCGGCCUGCUCAGCAGCUGUGGAGCGGCGCGUCAGCGG---UGGAGCUGCCAGUGGAGCGGUG ..((.(.(.((.((((...(((((...(((((((((((..(.((((((((.....)))))))).)..)))))))))))..)))))((..((....---))..)).)))).)).).).)). ( -66.10) >DroYak_CAF1 459 117 - 1 GGACGGGUGCGGUGGCCAAGCCGCCGACGCAGCUGCUGGUCUGGCUGAGCGCCCUGCUCGGCCUGCUCAGCAGCUGUGGAGCGGCGCGCCAGAGG---UGGAGCUGCCAGUGGAGCGGUG ..((.(.(.((.((((...(((((...(((((((((((..(.((((((((.....)))))))).)..)))))))))))..)))))((.(((....---))).)).)))).)).).).)). ( -68.20) >DroAna_CAF1 153 120 - 1 GCACGGAGGCGGUGGCGUUGCCGCCCACGCAACUGCUGGUGUGGCUGAGCGCUCUGCUCGGGCUACUGGGCAGCUGUGGGGCGGCCCGGCAGCGGUGGUGGAGCUGCCAGUGGUGGGGCG .(((((.((((....))))(((((((.((((.(((((.(.((((((((((.....)))).))))))).))))).)))))))))))))((((((.........)))))).)))........ ( -63.50) >consensus GGACGGGCGCGGUGGCCAAGCCGCCCACGCAGCUGCUGGUCUGGCUGAGCGCCCUGCUCGGCCUGCUCAGCAGCUGUGGAGCGGCGCGUCAGAGG___UGGAGCUGCCAGUGGAGCGGUG .(((((.(((((((((...))))))..((((((((((((.(.((((((((.....)))))))).).))))))))))))..))).).))))............(((((.......))))). (-54.87 = -56.05 + 1.18)
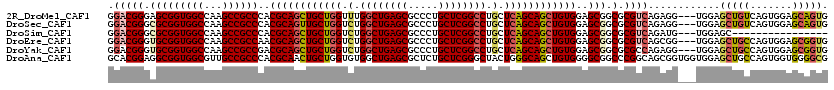

| Location | 20,207,399 – 20,207,519 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 76.83 |
| Mean single sequence MFE | -55.62 |
| Consensus MFE | -35.00 |
| Energy contribution | -35.06 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.23 |
| Mean z-score | -1.90 |
| Structure conservation index | 0.63 |
| SVM decision value | 1.19 |
| SVM RNA-class probability | 0.928562 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
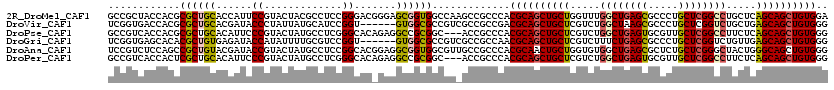
>2R_DroMel_CAF1 20207399 120 - 20766785 GCCGCUACCACGCGCUGCACCAUUCCGUACUACGCCUCCGGGACGGGAGCGGUGGCCAAGCCGCCCACGCAGCUGCUGGUUUGGCUGAGCGCCCUGCUCGGCCUGCUCAGCAGCUGUGGA ((.(((.((((.((((.(.((.(((((...........))))).)))))))))))...))).))...(((((((((((((..((((((((.....)))))))).)).))))))))))).. ( -62.80) >DroVir_CAF1 211 114 - 1 UCGGUGACCACGCGCUGCACGAUACCCUAUUAUGCAUCCGGU------GUGGCGCCGUCGCCGCCGACGCAGCUGCUCGUCUGGCUAAGCGCCCUGCUCGGUCUGCUGAGCAGCUGUGGG .(((((((...((((..((((((.(........).)))..))------)..)))).)))))))....((((((((((((.(.(((..(((.....)))..))).).)))))))))))).. ( -56.10) >DroPse_CAF1 301 117 - 1 GCCGUCACCACGCGCUGCACAUUCCCGUACUAUGCCUCGGGCACAGAGGCCGCGGC---ACCGCCCACGCAGCUGCUCGUCUGGCUGAGUGCGUUGCUCGGCCUUCUCAGCAGCUGUGGG .......(((((.(((((.....((((..........))))....(((((((.(((---(.(((..((.(((((........))))).))))).)))))))))))....)))))))))). ( -51.40) >DroGri_CAF1 244 114 - 1 UCGGUGAGCACACGCUGUGAGAUACCAUAUUUUGCGUCCGGU------GUGGCGCCGUCGCCGCCAACGCAGCUGCUCGUCUUUCUGAGCGCCCUGCUCGGUCUGUUGAGCAGCUGUGGG .((((((((.(((((((..(((((...)))))..)....)))------)))..))..))))))....((((((((((((.(...((((((.....))))))...).)))))))))))).. ( -52.20) >DroAna_CAF1 193 120 - 1 UCCGUCUCCAGCCGCUGUACGAUACCGUACUAUGCCUCCGGCACGGAGGCGGUGGCGUUGCCGCCCACGCAACUGCUGGUGUGGCUGAGCGCUCUGCUCGGGCUACUGGGCAGCUGUGGG .(((.(((..(((((((((((....)))))...(((((((...)))))))))))))...((((((((.((....))))).))))).))))((.((((((((....))))))))..)))). ( -60.30) >DroPer_CAF1 301 117 - 1 GCCGUCACCACUCGCUGCACAUUCCCGUACUAUGCCUCGGGCACAGAGGCCGCGGC---ACCGCCCACGCAGCUGCUCGUCUGGCUGAGUGCGUUGCUCGGCCUUCUCAGCAGCUGUGGG .............(((((((......)).....(((((.......))))).)))))---.....((.((((((((((.....((((((((.....)))))))).....)))))))))))) ( -50.90) >consensus GCCGUCACCACGCGCUGCACGAUACCGUACUAUGCCUCCGGCAC_GAGGCGGCGGCGU_GCCGCCCACGCAGCUGCUCGUCUGGCUGAGCGCCCUGCUCGGCCUGCUCAGCAGCUGUGGG ............((((((......((.............)).......)))))).............((((((((((.....((((((((.....)))))))).....)))))))))).. (-35.00 = -35.06 + 0.06)
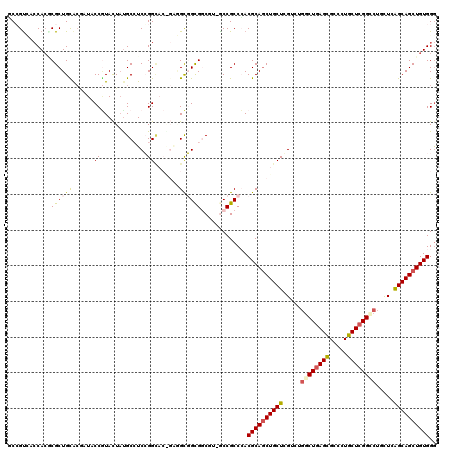
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:11:32 2006