
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 20,108,290 – 20,108,407 |
| Length | 117 |
| Max. P | 0.676620 |

| Location | 20,108,290 – 20,108,407 |
|---|---|
| Length | 117 |
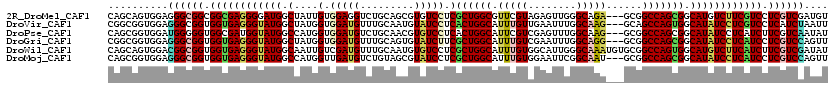
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.54 |
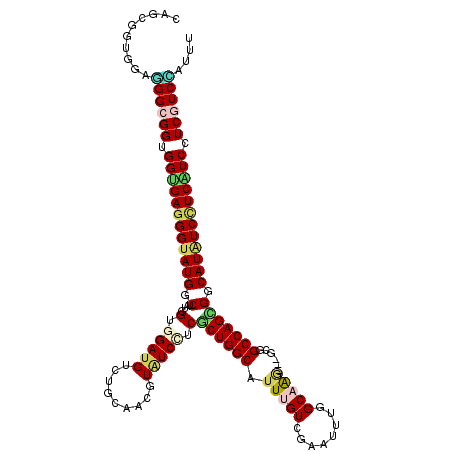
| Mean single sequence MFE | -47.28 |
| Consensus MFE | -35.21 |
| Energy contribution | -35.47 |
| Covariance contribution | 0.26 |
| Combinations/Pair | 1.36 |
| Mean z-score | -1.71 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.30 |
| SVM RNA-class probability | 0.676620 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 20108290 117 - 20766785 CAGCAGUGGAGGGCGGCGGCGAGGGGAUGGCUAUUGUGGAGGUCUGCAGCGUGUCCUCGCUGGCGUUCGUAGAGUUGGGCAGA---GCGGCCAGCGGCAUGUCUUCGUCCUCGUCGAUGU ........((((((((.((((((((.(((....(((..(....)..)))))).)))))((((((((((((........)).))---)).)))))).....))).))))))))........ ( -47.90) >DroVir_CAF1 15856 117 - 1 CGGCGGUGGAGGGCGGUGGUGAGGGUAUGGCUAUGGUGGAUGUUUGCAAUGUAUCCUCACUGGCAUUUGUUGAAUUUGGCAAG---GCAGCCAGUGGCAUAUCCUCGUCCUCAUCUAAUU ....((((.(((((......(((((((((........(((((.........)))))((((((((.((((((......))))))---...))))))))))))))))))))))))))..... ( -41.60) >DroPse_CAF1 8990 117 - 1 CAGCGGUGGAUGGGGGUGGCGAUGGUAUGGCCAUGGUGGAUGUCUGCAACGUGUCCUCACUGGCAUUCGUCGAGUUUGGCAAG---GCGGCCAGCGGCAUAUCCUCAUCUUCGUCAAUAU ..((((.(((((((((((((..((((...((((.(.(.((((..(((...(((....)))..)))..)))).).).))))...---...))))...)).)))))))))))))))...... ( -44.20) >DroGri_CAF1 11092 117 - 1 CGGCGGUGGAGGGCGGUGGUGAGGGUAUGGCUAUGGUGGAUGUUUGCAGUGUAUCUUCGCUGGCAUUUGUCGAAUUUGGCAGG---GCGGCCAGCGGCAUAUCCUCAUCCUCGUCCAGUU ..........((((((.((((((((((((.(...((((.((.......)).))))...((((((.(((((((....)))))))---...))))))).)))))))))))).)))))).... ( -51.40) >DroWil_CAF1 16921 120 - 1 CAGCAGUGGACGGCGGUGGUGAGGGUAUGGCAAUUGUCGAUGUUUGCAAUGUGUCCUCGCUGGCAUUUGUGGCAUUGGGCAAAUGUGCGGCCAGUGGCAUGUCUUCAUCUUCGUCGAUAU ..........((((((.(((((((..(((.((...((((((((((((...(((((...((........)))))))...)))))))).))))...)).)))..))))))).)))))).... ( -45.50) >DroMoj_CAF1 13906 117 - 1 CAGCGGUGGAGGGCGGUGGUGAGGGUAUGGCCAUGGUUGAUGUCUGUAGCGUAUCCUCGCUGGCAUUUGUGGAAUUCGGCAAU---GCGGCCAGCGGCAUAUCCUCAUCCUCGUCCAGUU ..........((((((.((((((((((((.((..((...((((.....))))..))..((((((..((((.(....).)))).---...)))))))))))))))))))).)))))).... ( -53.10) >consensus CAGCGGUGGAGGGCGGUGGUGAGGGUAUGGCUAUGGUGGAUGUCUGCAACGUAUCCUCGCUGGCAUUUGUCGAAUUUGGCAAG___GCGGCCAGCGGCAUAUCCUCAUCCUCGUCCAUUU ..........((((((.((((((((((((.(....(.(((((.........))))).)((((((.(((((........)))))......))))))).)))))))))))).)))))).... (-35.21 = -35.47 + 0.26)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:11:07 2006