
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 20,104,813 – 20,104,906 |
| Length | 93 |
| Max. P | 0.967005 |

| Location | 20,104,813 – 20,104,906 |
|---|---|
| Length | 93 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 78.71 |
| Mean single sequence MFE | -35.27 |
| Consensus MFE | -21.77 |
| Energy contribution | -22.15 |
| Covariance contribution | 0.38 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.58 |
| Structure conservation index | 0.62 |
| SVM decision value | 1.60 |
| SVM RNA-class probability | 0.967005 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
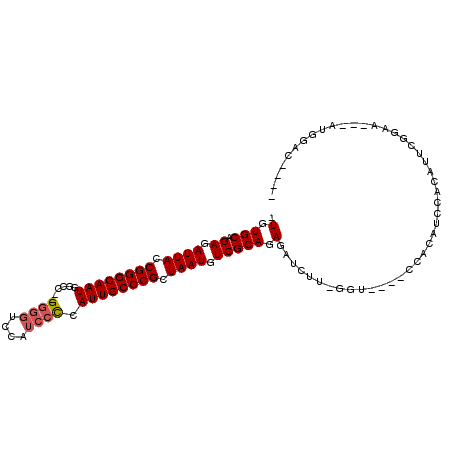
>2R_DroMel_CAF1 20104813 93 + 20766785 -UGUGCAGAGAUUACCGGGCAAUGGCC--GGGGUCCAUCCCCAUUGCCCGCUAAUGUCGCAGAGAUCUU-GGU----CCACAUCCACAUUCGGAA---AUGGAC---- -(.(((.((.((((.((((((((((..--(((.....))))))))))))).)))).))))).)......-.((----(((..(((......))).---.)))))---- ( -38.30) >DroGri_CAF1 7209 94 + 1 UUGUGCAGAGAUUACCGGGCAAUUUCC---CAUU--------AUUGCCCGCUAAUGUCGCAGAGUUGACCAGUGCUGCCACAUGCACA--A-GCAACCUUAGAGUUGC (((((((((.((((.((((((((....---....--------)))))))).)))).))((((..((....))..))))....))))))--)-(((((......))))) ( -36.90) >DroSec_CAF1 5231 93 + 1 -UGUGCAGAGAUUACCGGGCAAUGGUC--GGGGUCCAUCCCCAUUGCCCGCUAAUGUCGCAGAGAUCUU-GGU----CCACAUCCACAUUCGGAA---AUGGAC---- -(.(((.((.((((.((((((((((..--(((.....))))))))))))).)))).))))).)......-.((----(((..(((......))).---.)))))---- ( -38.30) >DroSim_CAF1 6573 92 + 1 -UGUGCAGAGAUUACCGGGCAAUGGUC--GGGGUCCAUCCCCAUUGCCCGCUAAUGUCGCAGAGAUCUU-GG-----CCACAUCCACAUUCGGAA---AUGGAC---- -(.(((.((.((((.((((((((((..--(((.....))))))))))))).)))).))))).)......-..-----(((..(((......))).---.)))..---- ( -35.00) >DroEre_CAF1 7634 89 + 1 -UGUGCAGAGAUUACCGGGCAAUGGCC--GGGGUCCAUCCUAAUUGCCCGCUAAUGUCGCAGAGAUCUU-GGUGC--CC------ACAUGAGGAA---ACGGAU---- -(.(((.((.((((.((((((((....--.(((.....))).)))))))).)))).))))).)..((((-.(((.--..------.))).)))).---......---- ( -29.50) >DroYak_CAF1 7552 97 + 1 -UGUGCAGAGAUUACCGGGCAAUGGCUAAGGGGUCCAUCCUAAUUGCCCGCUAAUGUCGCAGAGAUCUU-GGUGC--CCCCACCCCCAUUCGGAA---ACGGAU---- -(.(((.((.((((.((((((((.....((((.....)))).)))))))).)))).))))).)......-((((.--...))))...(((((...---.)))))---- ( -33.60) >consensus _UGUGCAGAGAUUACCGGGCAAUGGCC__GGGGUCCAUCCCCAUUGCCCGCUAAUGUCGCAGAGAUCUU_GGU____CCACAUCCACAUUCGGAA___AUGGAC____ .(.(((.((.((((.((((((((......((((....)))).)))))))).)))).))))).)............................................. (-21.77 = -22.15 + 0.38)

| Location | 20,104,813 – 20,104,906 |
|---|---|
| Length | 93 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | reverse |
| Mean pairwise identity | 78.71 |
| Mean single sequence MFE | -35.25 |
| Consensus MFE | -19.16 |
| Energy contribution | -19.20 |
| Covariance contribution | 0.04 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.40 |
| Structure conservation index | 0.54 |
| SVM decision value | 1.11 |
| SVM RNA-class probability | 0.916698 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
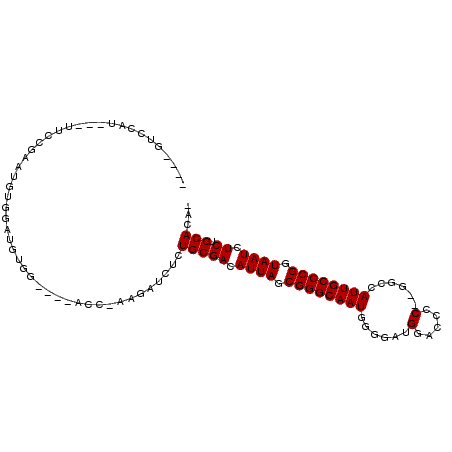
>2R_DroMel_CAF1 20104813 93 - 20766785 ----GUCCAU---UUCCGAAUGUGGAUGUGG----ACC-AAGAUCUCUGCGACAUUAGCGGGCAAUGGGGAUGGACCCC--GGCCAUUGCCCGGUAAUCUCUGCACA- ----((((((---.((((....)))).))))----)).-........(((((.((((.((((((((((...(((...))--).)))))))))).)))).)).)))..- ( -40.10) >DroGri_CAF1 7209 94 - 1 GCAACUCUAAGGUUGC-U--UGUGCAUGUGGCAGCACUGGUCAACUCUGCGACAUUAGCGGGCAAU--------AAUG---GGAAAUUGCCCGGUAAUCUCUGCACAA ((((((....))))))-.--(((((((((.((((....((....)))))).)))(((.((((((((--------....---....)))))))).)))....)))))). ( -37.10) >DroSec_CAF1 5231 93 - 1 ----GUCCAU---UUCCGAAUGUGGAUGUGG----ACC-AAGAUCUCUGCGACAUUAGCGGGCAAUGGGGAUGGACCCC--GACCAUUGCCCGGUAAUCUCUGCACA- ----((((((---.((((....)))).))))----)).-........(((((.((((.((((((((((...(((...))--).)))))))))).)))).)).)))..- ( -40.10) >DroSim_CAF1 6573 92 - 1 ----GUCCAU---UUCCGAAUGUGGAUGUGG-----CC-AAGAUCUCUGCGACAUUAGCGGGCAAUGGGGAUGGACCCC--GACCAUUGCCCGGUAAUCUCUGCACA- ----(.((((---.((((....)))).))))-----.)-........(((((.((((.((((((((((...(((...))--).)))))))))).)))).)).)))..- ( -36.00) >DroEre_CAF1 7634 89 - 1 ----AUCCGU---UUCCUCAUGU------GG--GCACC-AAGAUCUCUGCGACAUUAGCGGGCAAUUAGGAUGGACCCC--GGCCAUUGCCCGGUAAUCUCUGCACA- ----....((---.(((......------))--).)).-........(((((.((((.((((((((..((.(((...))--).)))))))))).)))).)).)))..- ( -26.60) >DroYak_CAF1 7552 97 - 1 ----AUCCGU---UUCCGAAUGGGGGUGGGG--GCACC-AAGAUCUCUGCGACAUUAGCGGGCAAUUAGGAUGGACCCCUUAGCCAUUGCCCGGUAAUCUCUGCACA- ----.(((((---(....))))))((((...--.))))-........(((((.((((.((((((((..((..((....))...)))))))))).)))).)).)))..- ( -31.60) >consensus ____GUCCAU___UUCCGAAUGUGGAUGUGG____ACC_AAGAUCUCUGCGACAUUAGCGGGCAAUGGGGAUGGACCCC__GGCCAUUGCCCGGUAAUCUCUGCACA_ ...............................................(((((.((((.((((((((......(.....)......)))))))).)))).)).)))... (-19.16 = -19.20 + 0.04)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:11:01 2006