
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 20,032,886 – 20,033,028 |
| Length | 142 |
| Max. P | 0.989914 |

| Location | 20,032,886 – 20,032,988 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 77.80 |
| Mean single sequence MFE | -52.55 |
| Consensus MFE | -36.76 |
| Energy contribution | -38.15 |
| Covariance contribution | 1.39 |
| Combinations/Pair | 1.22 |
| Mean z-score | -1.93 |
| Structure conservation index | 0.70 |
| SVM decision value | 1.34 |
| SVM RNA-class probability | 0.943786 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
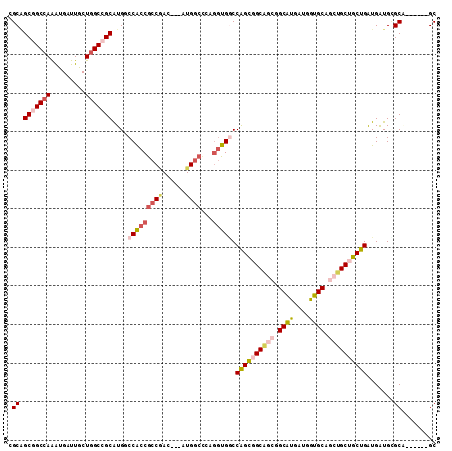
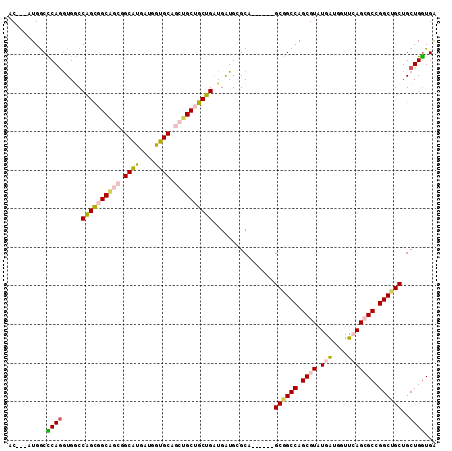
>2R_DroMel_CAF1 20032886 102 + 20766785 GGCGGCCGCCAGGUGGUUACUGGCUGCGUGCCCACCGCCGAC---GUGGCUCAGGUGACCAGCUGCAGCGGCGUGGUGGUGCAGCUGCUGCUGGUGGUGCGCC------GC (((((((((((.(..((....(((((((...(((((((((..---(..(((..(....).)))..)..))))..))))))))))))))..)))))))).))))------.. ( -58.40) >DroVir_CAF1 1072 111 + 1 UGCAGCGGCCAAAUGAUUGCUGGCCGCAUGGCCGCCGCCAACGCCAUGGCUCAAAUGGCCGGUGGCCGCGGCAUGAUGAUGCAGCUGCUGCUGAUGAUGGGCCGCGGCGGC ....(((((((.........)))))))...((((((((((..(((((.......)))))...)))).(((((.(.((.((.((((....)))))).)).)))))))))))) ( -58.90) >DroPse_CAF1 1117 99 + 1 CGCAGCGGCCAAAUGAUUGCUGGCCGCGUGUCCACCGCCGAC---AUGGCCCAGGUGGCCGGCGGCA---GCGUGAUGGUGCAACUGCUGCUGAUGAUGUGCA------GC .((((((((((.........)))))))....((((((((...---..)))...))))).((((((((---(..((......)).)))))))))......))).------.. ( -42.30) >DroMoj_CAF1 3383 111 + 1 AGCAGCAGCCAAAUGAUUGCUAGCCGCAUGGCCGCCGCCAACGCCAUGGCUCAGGUGCCCAGUUGCAGCAGCAUGAUGGUGCAGCUGCUGCUGAUGAUGGGCAGCAGCGGC (((((((......)).))))).(((((.((((((..((....))..)))).))(.((((((...((((((((.((......))))))))))......)))))).).))))) ( -51.10) >DroAna_CAF1 3081 102 + 1 GGCCGCGGCCAGAUGAUUGCUGGCCGCAUGACCACCACCUAC---GUGACCCAGGUGGCCAGCGGCGGCGGCAUGGUGAUGCAGCUGCUGCUGGUGGUGCGCG------GC .(((((((((((.......))))))((((.(((.((((((..---.......))))))..(((((((((.((((....)))).)))))))))))).)))))))------)) ( -62.30) >DroPer_CAF1 1117 99 + 1 CGCAGCGGCCAAAUGAUUGCUGGCCGCGUGUCCACCGCCGAC---AUGGCCCAGGUGGCCGGCGGCA---GCGUGAUGGUGCAACUGCUGCUGAUGAUGUGCA------GC .((((((((((.........)))))))....((((((((...---..)))...))))).((((((((---(..((......)).)))))))))......))).------.. ( -42.30) >consensus CGCAGCGGCCAAAUGAUUGCUGGCCGCAUGGCCACCGCCGAC___AUGGCCCAGGUGGCCAGCGGCAGCGGCAUGAUGGUGCAGCUGCUGCUGAUGAUGCGCA______GC .((.(((((((.........)))))))....(((((((((......))))...))))).((((((((((.((((....)))).)))))))))).......))......... (-36.76 = -38.15 + 1.39)

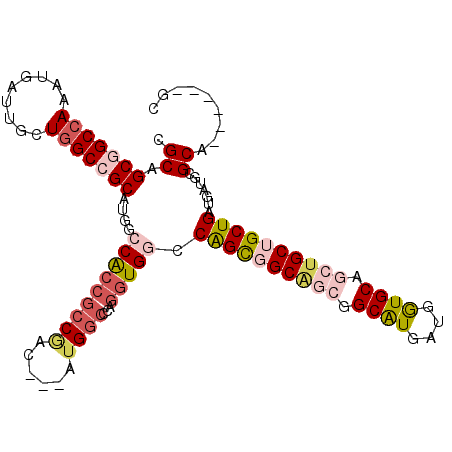
| Location | 20,032,886 – 20,032,988 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 77.80 |
| Mean single sequence MFE | -51.87 |
| Consensus MFE | -31.78 |
| Energy contribution | -32.15 |
| Covariance contribution | 0.37 |
| Combinations/Pair | 1.30 |
| Mean z-score | -3.03 |
| Structure conservation index | 0.61 |
| SVM decision value | 2.19 |
| SVM RNA-class probability | 0.989914 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 20032886 102 - 20766785 GC------GGCGCACCACCAGCAGCAGCUGCACCACCACGCCGCUGCAGCUGGUCACCUGAGCCAC---GUCGGCGGUGGGCACGCAGCCAGUAACCACCUGGCGGCCGCC ((------(((.((((((((((.(((((.((........)).))))).)))))).......(((..---...))))))).))..((.(((((.......))))).))))). ( -51.80) >DroVir_CAF1 1072 111 - 1 GCCGCCGCGGCCCAUCAUCAGCAGCAGCUGCAUCAUCAUGCCGCGGCCACCGGCCAUUUGAGCCAUGGCGUUGGCGGCGGCCAUGCGGCCAGCAAUCAUUUGGCCGCUGCA ((.((((((((..((.((((((....)))).)).))...))))))))...((((((..(((((....))((((((.(((....))).))))))..)))..))))))..)). ( -56.70) >DroPse_CAF1 1117 99 - 1 GC------UGCACAUCAUCAGCAGCAGUUGCACCAUCACGC---UGCCGCCGGCCACCUGGGCCAU---GUCGGCGGUGGACACGCGGCCAGCAAUCAUUUGGCCGCUGCG ((------(((.........)))))....(((...((.(((---((((((.((((.....))))..---).))))))))))...((((((((.......))))))))))). ( -46.70) >DroMoj_CAF1 3383 111 - 1 GCCGCUGCUGCCCAUCAUCAGCAGCAGCUGCACCAUCAUGCUGCUGCAACUGGGCACCUGAGCCAUGGCGUUGGCGGCGGCCAUGCGGCUAGCAAUCAUUUGGCUGCUGCU ((((((((((((((......((((((((((......)).))))))))...)))))......((....))...)))))))))...(((((.(((.........)))))))). ( -54.30) >DroAna_CAF1 3081 102 - 1 GC------CGCGCACCACCAGCAGCAGCUGCAUCACCAUGCCGCCGCCGCUGGCCACCUGGGUCAC---GUAGGUGGUGGUCAUGCGGCCAGCAAUCAUCUGGCCGCGGCC ((------((((((((((((((.((.((.((((....)))).)).)).)))))(((((((.(...)---.)))))))))))...))((((((.......))))))))))). ( -55.00) >DroPer_CAF1 1117 99 - 1 GC------UGCACAUCAUCAGCAGCAGUUGCACCAUCACGC---UGCCGCCGGCCACCUGGGCCAU---GUCGGCGGUGGACACGCGGCCAGCAAUCAUUUGGCCGCUGCG ((------(((.........)))))....(((...((.(((---((((((.((((.....))))..---).))))))))))...((((((((.......))))))))))). ( -46.70) >consensus GC______UGCACAUCAUCAGCAGCAGCUGCACCAUCACGCCGCUGCCGCCGGCCACCUGAGCCAU___GUCGGCGGUGGACACGCGGCCAGCAAUCAUUUGGCCGCUGCC .........((...(((((.((.(((((.((........)).)))))....(((.......))).........)))))))....((((((((.......)))))))).)). (-31.78 = -32.15 + 0.37)

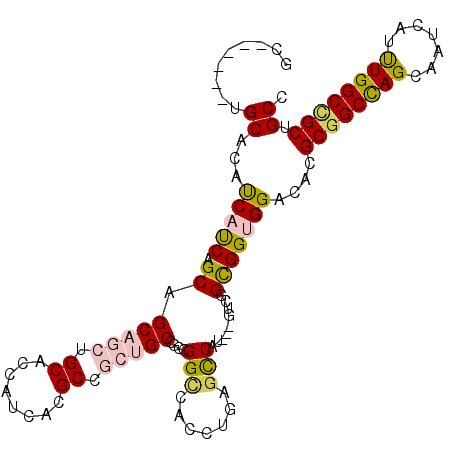

| Location | 20,032,926 – 20,033,028 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 80.78 |
| Mean single sequence MFE | -53.58 |
| Consensus MFE | -36.72 |
| Energy contribution | -37.12 |
| Covariance contribution | 0.39 |
| Combinations/Pair | 1.29 |
| Mean z-score | -2.83 |
| Structure conservation index | 0.69 |
| SVM decision value | 1.31 |
| SVM RNA-class probability | 0.940390 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
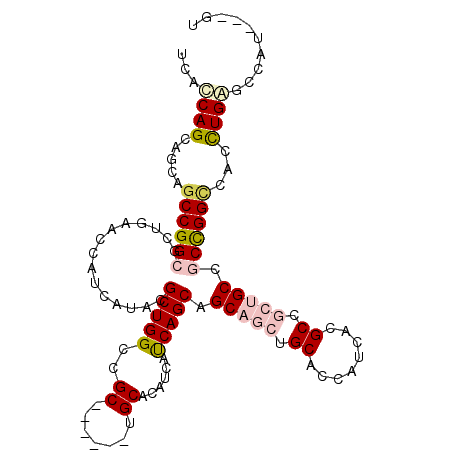
>2R_DroMel_CAF1 20032926 102 + 20766785 AC---GUGGCUCAGGUGACCAGCUGCAGCGGCGUGGUGGUGCAGCUGCUGCUGGUGGUGCGCC------GCUGCCAGCGUGUGGUGGUUCAGGGCCGGCUGCUGCUGGUGG ..---(((((.((..(.((((((.(((((.(((......))).))))).)))))).))).)))------)).(((((((.(..(((((.....))).))..)))))))).. ( -52.70) >DroVir_CAF1 1112 111 + 1 ACGCCAUGGCUCAAAUGGCCGGUGGCCGCGGCAUGAUGAUGCAGCUGCUGCUGAUGAUGGGCCGCGGCGGCGGCCAGCGUAUGAUGAUUAAGCGCCGGCUGCUGCUGAUGA ..(((((((((.....)))).)))))((((((.(.((.((.((((....)))))).)).)))))))(((((((((.((((.(((...))).)))).)))))))))...... ( -56.50) >DroPse_CAF1 1157 99 + 1 AC---AUGGCCCAGGUGGCCGGCGGCA---GCGUGAUGGUGCAACUGCUGCUGAUGAUGUGCA------GCGGCCAGCGUAUGAUGGUUCAGCGCCGGCUGCUGCUGGUGA ((---((((((.....))))(((((((---(..((......)).))))))))....))))(((------((((((.((((.(((....))))))).)))))))))...... ( -48.30) >DroMoj_CAF1 3423 111 + 1 ACGCCAUGGCUCAGGUGCCCAGUUGCAGCAGCAUGAUGGUGCAGCUGCUGCUGAUGAUGGGCAGCAGCGGCAGCCAGCGUAUGAUGGUUAAGCGCCGGCUGCUGCUGGUGA .((((........(.((((((...((((((((.((......))))))))))......)))))).)((((((((((.((((.(((...))).)))).)))))))))))))). ( -61.20) >DroAna_CAF1 3121 102 + 1 AC---GUGACCCAGGUGGCCAGCGGCGGCGGCAUGGUGAUGCAGCUGCUGCUGGUGGUGCGCG------GCGGCCAGCGUGUGGUGAUUCAGGGCCGGCUGCUGCUGGUGG ..---....((((..(.((((((((((((.((((....)))).)))))))))))).)...(((------((((((.((.(.(((....))).))).)))))))))))).). ( -54.50) >DroPer_CAF1 1157 99 + 1 AC---AUGGCCCAGGUGGCCGGCGGCA---GCGUGAUGGUGCAACUGCUGCUGAUGAUGUGCA------GCGGCCAGCGUAUGAUGGUUCAGCGCCGGCUGCUGCUGGUGA ((---((((((.....))))(((((((---(..((......)).))))))))....))))(((------((((((.((((.(((....))))))).)))))))))...... ( -48.30) >consensus AC___AUGGCCCAGGUGGCCAGCGGCAGCGGCAUGAUGGUGCAGCUGCUGCUGAUGAUGCGCA______GCGGCCAGCGUAUGAUGGUUCAGCGCCGGCUGCUGCUGGUGA ..........((((.....((((((((((.((((....)))).))))))))))................((((((.((((.(((....))))))).))))))..))))... (-36.72 = -37.12 + 0.39)

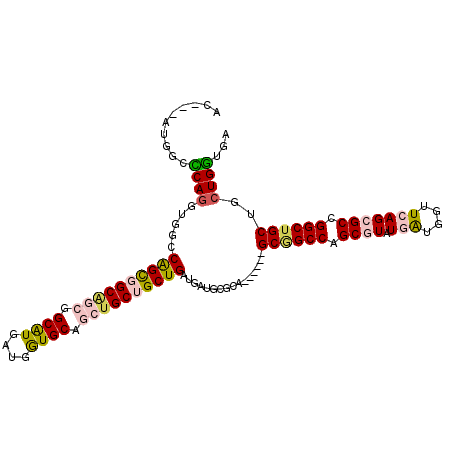
| Location | 20,032,926 – 20,033,028 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 80.78 |
| Mean single sequence MFE | -43.14 |
| Consensus MFE | -24.42 |
| Energy contribution | -25.37 |
| Covariance contribution | 0.95 |
| Combinations/Pair | 1.21 |
| Mean z-score | -3.10 |
| Structure conservation index | 0.57 |
| SVM decision value | 1.30 |
| SVM RNA-class probability | 0.938967 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
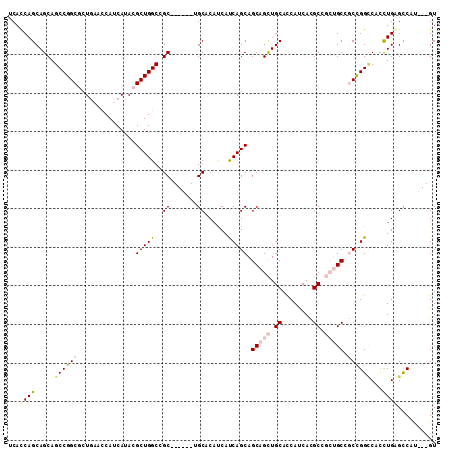
>2R_DroMel_CAF1 20032926 102 - 20766785 CCACCAGCAGCAGCCGGCCCUGAACCACCACACGCUGGCAGC------GGCGCACCACCAGCAGCAGCUGCACCACCACGCCGCUGCAGCUGGUCACCUGAGCCAC---GU .........((.((((((..((......))...)))))).))------(((.((..((((((.(((((.((........)).))))).))))))....)).)))..---.. ( -41.40) >DroVir_CAF1 1112 111 - 1 UCAUCAGCAGCAGCCGGCGCUUAAUCAUCAUACGCUGGCCGCCGCCGCGGCCCAUCAUCAGCAGCAGCUGCAUCAUCAUGCCGCGGCCACCGGCCAUUUGAGCCAUGGCGU ............(((((((.............)))))))((((((((((((..((.((((((....)))).)).))...))))))))....((((....).)))..)))). ( -45.12) >DroPse_CAF1 1157 99 - 1 UCACCAGCAGCAGCCGGCGCUGAACCAUCAUACGCUGGCCGC------UGCACAUCAUCAGCAGCAGUUGCACCAUCACGC---UGCCGCCGGCCACCUGGGCCAU---GU ......(((((.(((((((.(((....)))..))))))).))------)))((((.....((.(((((((......)).))---))).)).((((.....))))))---)) ( -40.80) >DroMoj_CAF1 3423 111 - 1 UCACCAGCAGCAGCCGGCGCUUAACCAUCAUACGCUGGCUGCCGCUGCUGCCCAUCAUCAGCAGCAGCUGCACCAUCAUGCUGCUGCAACUGGGCACCUGAGCCAUGGCGU (((.((((.((((((((((.............)))))))))).)))).((((((......((((((((((......)).))))))))...))))))..)))((....)).. ( -52.52) >DroAna_CAF1 3121 102 - 1 CCACCAGCAGCAGCCGGCCCUGAAUCACCACACGCUGGCCGC------CGCGCACCACCAGCAGCAGCUGCAUCACCAUGCCGCCGCCGCUGGCCACCUGGGUCAC---GU ...(((((.((.((((((..((......))...)))))).))------.))......(((((.((.((.((((....)))).)).)).))))).....))).....---.. ( -38.20) >DroPer_CAF1 1157 99 - 1 UCACCAGCAGCAGCCGGCGCUGAACCAUCAUACGCUGGCCGC------UGCACAUCAUCAGCAGCAGUUGCACCAUCACGC---UGCCGCCGGCCACCUGGGCCAU---GU ......(((((.(((((((.(((....)))..))))))).))------)))((((.....((.(((((((......)).))---))).)).((((.....))))))---)) ( -40.80) >consensus UCACCAGCAGCAGCCGGCGCUGAACCAUCAUACGCUGGCCGC______UGCACAUCAUCAGCAGCAGCUGCACCAUCACGCCGCUGCCGCCGGCCACCUGAGCCAU___GU ...((((.....((((((...............(((((..((.......))......))))).(((((.((........)).))))).))))))...)))).......... (-24.42 = -25.37 + 0.95)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:10:13 2006