
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 19,916,549 – 19,916,708 |
| Length | 159 |
| Max. P | 0.903618 |

| Location | 19,916,549 – 19,916,668 |
|---|---|
| Length | 119 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.11 |
| Mean single sequence MFE | -42.73 |
| Consensus MFE | -33.01 |
| Energy contribution | -31.35 |
| Covariance contribution | -1.66 |
| Combinations/Pair | 1.40 |
| Mean z-score | -1.93 |
| Structure conservation index | 0.77 |
| SVM decision value | 0.83 |
| SVM RNA-class probability | 0.860506 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
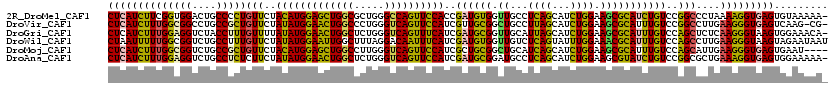
>2R_DroMel_CAF1 19916549 119 - 20766785 CUCAUCUUCGGUGGACUGCCCCUGUUCUACAUGGAGCUGGCGCUGGGCCAGUUCCACCGAUGUGGUUGCCUCAGCAUCUGGAAGCGCAUCUGUCCGGCCCUAAAAGGUGAGUGUAAAAA- ((((((((.(((((((...............((((((((((.....))))))))))..((((((.((.((.........)))).)))))).)))).)))....))))))))........- ( -45.50) >DroVir_CAF1 8318 118 - 1 CUCAUCUUUGGCGGCCUGCCGCUGUUCUAUAUGGAACUGGCCCUGGGUCAGUUCCAUCGUUGCGGCUGCCUUAGCAUCUGGAAGCGCAUUUGUCCGGCCUUGAAGGGUGAGUCAAG-CG- (((((((((.(.((((.(((((........((((((((((((...))))))))))))....)))))((((((..(....).))).))).......)))).).))))))))).....-..- ( -49.50) >DroGri_CAF1 7368 119 - 1 CUCAUCUUUGGAGGUCUACCUUUGUUUUAUAUGGAACUGGCUCUGGGUCAGUUUCAUCGAUGCGGUUGCAUUAGCAUCUGGAAGCGCAUUUGUCCAGCUCUCAAGGGUAAGUGGAAACA- ((.(((((((..(((...............(((((((((((.....))))))))))).((((((.((.((........)).)).))))))......)))..))))))).))((....))- ( -35.40) >DroWil_CAF1 29019 120 - 1 CUAAUUUUUGGCGGUCUGCCUUUGUUCUAUAUGGAAUUGGCUUUAGGACAAUUUCAUCGAUGUGGUUGUCUCAGUAUUUGGAAACGCAUUUGUCCAGCCUUGAAGGGUAAGUAGAAUAAU .........(((.....))).(((((((((.((((....((....(((((((..((....))..)))))))........(....))).....))))((((....))))..))))))))). ( -30.90) >DroMoj_CAF1 14310 116 - 1 CUCAUCUUUGGCGGUCUGCCGCUGUUCUACAUGGAGCUGGCCUUGGGUCAGUUCCAUCGCUGCGGCUGCAUCAGCAUCUGGAAGCGCAUUUGUCCAGCAUUGAAGGGUGAGUGAAU---- ((((((((((((.....)))((((..(...((((((((((((...))))))))))))((((.(((.(((....))).)))..)))).....)..))))....))))))))).....---- ( -48.80) >DroAna_CAF1 5843 119 - 1 CUCAUCUUUGGAGGUCUGCCUCUCUUCUAUAUGGAACUGGCUCUGGGUCAGUUCCAUCGAUGCGGAUGCCUCAGCAUCUGGAAGCGUAUCUGUCCGGCGCUGAAAGGUGAGUGGAAAAA- ((((((((((((((....))))).......(((((((((((.....)))))))))))...(.(((((((....))))))).)(((((.........))))).)))))))))........- ( -46.30) >consensus CUCAUCUUUGGCGGUCUGCCUCUGUUCUAUAUGGAACUGGCUCUGGGUCAGUUCCAUCGAUGCGGUUGCCUCAGCAUCUGGAAGCGCAUUUGUCCAGCCCUGAAGGGUGAGUGGAAAAA_ ((((((((((((((....)))))(((..(((((((((((((.....))))))))))..((((((.((...((((...)))).)))))))))))..)))....)))))))))......... (-33.01 = -31.35 + -1.66)
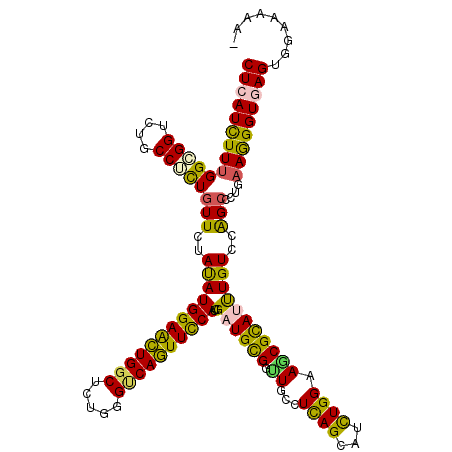
| Location | 19,916,588 – 19,916,708 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.22 |
| Mean single sequence MFE | -46.42 |
| Consensus MFE | -35.38 |
| Energy contribution | -34.64 |
| Covariance contribution | -0.74 |
| Combinations/Pair | 1.43 |
| Mean z-score | -2.61 |
| Structure conservation index | 0.76 |
| SVM decision value | 1.03 |
| SVM RNA-class probability | 0.903618 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
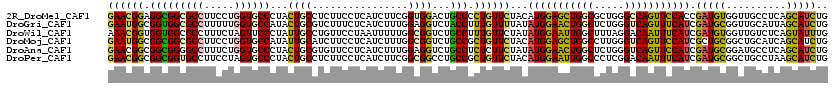
>2R_DroMel_CAF1 19916588 120 - 20766785 GAACGGAGGCGGCGCCUUCCUGGUGCCCUACUGCCUCUUCCUCAUCUUCGGUGGACUGCCCCUGUUCUACAUGGAGCUGGCGCUGGGCCAGUUCCACCGAUGUGGUUGCCUCAGCAUCUG ((((((.(((((((((.....))))))...(..((..............))..)...))).))))))....((((((((((.....))))))))))..(((((.(......).))))).. ( -51.64) >DroGri_CAF1 7407 120 - 1 GAAUGGCGGUGGCGCCUUUUUGGUGCCAUACUGCGUCUUUCUCAUCUUUGGAGGUCUACCUUUGUUUUAUAUGGAACUGGCUCUGGGUCAGUUUCAUCGAUGCGGUUGCAUUAGCAUCUG .....(((((((((((.....))))))))((((((((............(((((....))))).......(((((((((((.....))))))))))).)))))))))))........... ( -45.00) >DroWil_CAF1 29059 120 - 1 AAACGGUGGUGGCGCCUUUCUAGUUCCCUAUUGCCUGUUCCUAAUUUUUGGCGGUCUGCCUUUGUUCUAUAUGGAAUUGGCUUUAGGACAAUUUCAUCGAUGUGGUUGUCUCAGUAUUUG ....((((....))))...........(((..(((.(((((........(((.....)))............))))).)))..)))((((((..((....))..)))))).......... ( -28.25) >DroMoj_CAF1 14346 120 - 1 GAAUGGCGGCGGCGCCUUCCUGGUGCCAUAUUGCAUCUUCCUCAUCUUUGGCGGUCUGCCGCUGUUCUACAUGGAGCUGGCCUUGGGUCAGUUCCAUCGCUGCGGCUGCAUCAGCAUCUG ((((((((((((((((.....))))))............(((((....))).))...))))))))))...((((((((((((...)))))))))))).....(((.(((....))).))) ( -52.70) >DroAna_CAF1 5882 120 - 1 GAACGGCGGCGGGGCCUUUCUGGUGCCCUACUGCGUGUUCCUCAUCUUUGGAGGUCUGCCUCUCUUCUAUAUGGAACUGGCUCUGGGUCAGUUCCAUCGAUGCGGAUGCCUCAGCAUCUG (((((((((..((((((....)).))))..)))).)))))..((((...(((((....))))).......(((((((((((.....))))))))))).))))(((((((....))))))) ( -54.90) >DroPer_CAF1 6164 120 - 1 GAACGGCGGCGGUGCCUUCCUAGUGCCCUACUGCCUCUUCCUCAUCUUCGGCGGCCUGCCGCUGUUCUACAUGGAAUUGGCCCUCGGACAAUUUCAUCGAUGCGGCUGCCUAAGCAUCUG ((((((((((((.(((.....((((...))))(((..............))))))))))))))))))...(((((((((.((...)).))))))))).(((((..........))))).. ( -46.04) >consensus GAACGGCGGCGGCGCCUUCCUGGUGCCCUACUGCCUCUUCCUCAUCUUUGGCGGUCUGCCUCUGUUCUACAUGGAACUGGCUCUGGGUCAGUUCCAUCGAUGCGGUUGCCUCAGCAUCUG ((((((.(((((((((.....))))))...((((................))))...))).))))))...(((((((((((.....))))))))))).(((((..........))))).. (-35.38 = -34.64 + -0.74)
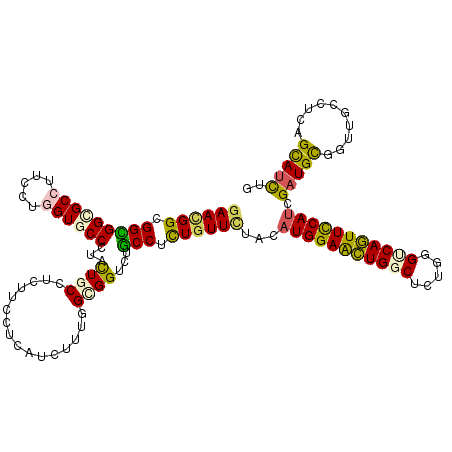
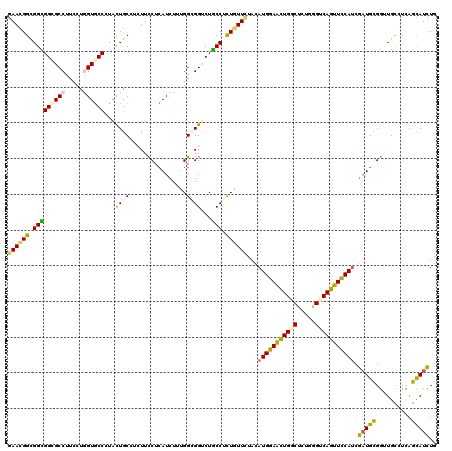
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:08:58 2006