| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 19,856,198 – 19,856,299 |
| Length | 101 |
| Max. P | 0.948024 |

| Location | 19,856,198 – 19,856,299 |
|---|---|
| Length | 101 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 79.73 |
| Mean single sequence MFE | -43.20 |
| Consensus MFE | -28.28 |
| Energy contribution | -28.82 |
| Covariance contribution | 0.53 |
| Combinations/Pair | 1.20 |
| Mean z-score | -2.13 |
| Structure conservation index | 0.65 |
| SVM decision value | 1.01 |
| SVM RNA-class probability | 0.900455 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
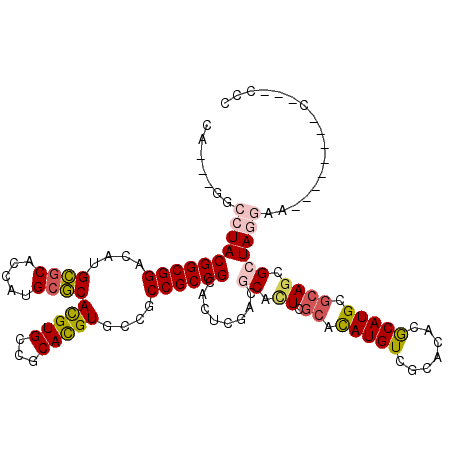
>2R_DroMel_CAF1 19856198 101 + 20766785 CA---G---UACGGCGGACACGGGCACCACGCCCACGUGCCGCACGUGCCGCCGCCGCAUUCGACGCACUCGCACAUGUCCCACACGCAUGCGCAGCGCUAGG----------C---UUC ..---(---((((((((.(((((((.....))))..))))))).))))).(((..(((....((.....))((.(((((.......))))).)).)))...))----------)---... ( -41.60) >DroVir_CAF1 130355 110 + 1 CA---GGCCUACGGCGGACAUGCGCACCAUGCGCAUGUGCCGCAUGUGCCGCCGCCGCACUCUACACAUUCACACAUGUCCCAUACGCAUGCCCAGCGCUAGGGCC--AU--GC---CCC ..---(((((..(((((((((((((.....)))))))).))(((((((..((....)).......((((......))))......))))))).....))).)))))--..--..---... ( -40.70) >DroPse_CAF1 102038 110 + 1 CA---GGCCUACGGCGGACAUGCGCACCAUGCGCAUGUGGCCCACGUGCCGCCGCCGCAUUCGACGCACUCGCACAUGUCGCAUACGCAUGCGCAGCGCUAGGCAA--AU--CC---UUC ..---.(((((((((((((((((((.....))))))))(((......))).))))))........((.((.((.((((.((....)))))).)))).)))))))..--..--..---... ( -49.30) >DroGri_CAF1 128972 117 + 1 CA---GGCCUACGGCGGGCAUGCGCAUCAUGCGCAUGUGCCGCACGUGCCACCGCCGCACUCCACACACUCGCACAUGUCGCACACGCAUGCGCAACGCUAGGAAAUUUCAAAUUUCCCC ..---(((...((((((((((((((.....))))))))...((....))..))))))..............((.((((.((....)))))).))...))).((((((.....)))))).. ( -42.80) >DroMoj_CAF1 180743 115 + 1 CA---GGCCUACGGCGGACAUGCGCACCACGCGCACGUGCCACACGUGCCGCCGCCGCACUCCACGCACUCGCACAUGUCGCAUACGCAUGCGCAGCGCUAGGGAC--UCAGACCUCCCC ..---((....((((((.(..(((.....)))(((((((...))))))).)))))))....))..((.((.((.((((.((....)))))).)))).))..((((.--.......)))). ( -44.40) >DroAna_CAF1 94720 105 + 1 CAGGCG---UACGGCGGGCAUGCCCACCAUGCGCACGUGCCCCAUGUGCCGCCGCCGCACUCGACGCACUCGCAUAUGUCGCACACACAUGCGCAGCGCUAGGA---------C---AUU ..((((---(.(((((((((((.....)))))(((((((...)))))))..)))))).)).....((...(((((.(((.....))).)))))..)))))....---------.---... ( -40.40) >consensus CA___GGCCUACGGCGGACAUGCGCACCAUGCGCACGUGCCGCACGUGCCGCCGCCGCACUCGACGCACUCGCACAUGUCGCACACGCAUGCGCAGCGCUAGGAA________C___CCC .......((((((((((....((((.....))))(((((...)))))....))))))........((.((.((.(((((.......))))).)))).))))))................. (-28.28 = -28.82 + 0.53)


| Location | 19,856,198 – 19,856,299 |
|---|---|
| Length | 101 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.73 |
| Mean single sequence MFE | -55.70 |
| Consensus MFE | -39.95 |
| Energy contribution | -39.98 |
| Covariance contribution | 0.03 |
| Combinations/Pair | 1.28 |
| Mean z-score | -2.42 |
| Structure conservation index | 0.72 |
| SVM decision value | 1.38 |
| SVM RNA-class probability | 0.948024 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
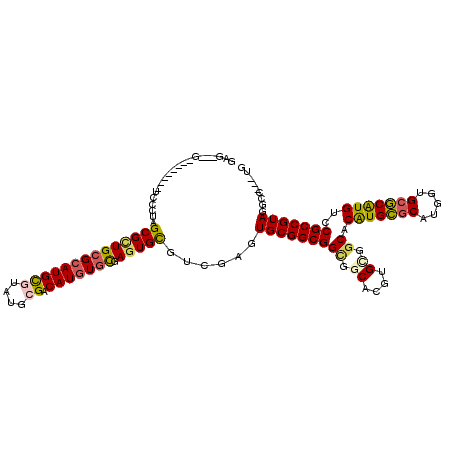
>2R_DroMel_CAF1 19856198 101 - 20766785 GAA---G----------CCUAGCGCUGCGCAUGCGUGUGGGACAUGUGCGAGUGCGUCGAAUGCGGCGGCGGCACGUGCGGCACGUGGGCGUGGUGCCCGUGUCCGCCGUA---C---UG ...---(----------((..(((((((((((((......).))))))).)))))((.....)))))(((((((((.(((.((((....)))).))).)))).)))))...---.---.. ( -51.70) >DroVir_CAF1 130355 110 - 1 GGG---GC--AU--GGCCCUAGCGCUGGGCAUGCGUAUGGGACAUGUGUGAAUGUGUAGAGUGCGGCGGCGGCACAUGCGGCACAUGCGCAUGGUGCGCAUGUCCGCCGUAGGCC---UG .((---((--((--(((....((((.......))))...(((((((((((.(((((((..((((.(((........))).)))).)))))))..))))))))))))))))..)))---). ( -54.10) >DroPse_CAF1 102038 110 - 1 GAA---GG--AU--UUGCCUAGCGCUGCGCAUGCGUAUGCGACAUGUGCGAGUGCGUCGAAUGCGGCGGCGGCACGUGGGCCACAUGCGCAUGGUGCGCAUGUCCGCCGUAGGCC---UG ...---..--..--..(((..((((((((((((((....)).))))))).)))))......((((((((.(((......)))((((((((.....))))))))))))))))))).---.. ( -57.40) >DroGri_CAF1 128972 117 - 1 GGGGAAAUUUGAAAUUUCCUAGCGUUGCGCAUGCGUGUGCGACAUGUGCGAGUGUGUGGAGUGCGGCGGUGGCACGUGCGGCACAUGCGCAUGAUGCGCAUGCCCGCCGUAGGCC---UG .(((((((.....))))))).((((((((((((((....)).))))))))).)))..((..(((((((((.((....)).)).(((((((.....)))))))..)))))))..))---.. ( -54.80) >DroMoj_CAF1 180743 115 - 1 GGGGAGGUCUGA--GUCCCUAGCGCUGCGCAUGCGUAUGCGACAUGUGCGAGUGCGUGGAGUGCGGCGGCGGCACGUGUGGCACGUGCGCGUGGUGCGCAUGUCCGCCGUAGGCC---UG ....((((((.(--.(((...((((((((((((((....)).))))))).)))))..))).)((((((((((((((((...)))))))(((.....))).)).))))))))))))---). ( -61.80) >DroAna_CAF1 94720 105 - 1 AAU---G---------UCCUAGCGCUGCGCAUGUGUGUGCGACAUAUGCGAGUGCGUCGAGUGCGGCGGCGGCACAUGGGGCACGUGCGCAUGGUGGGCAUGCCCGCCGUA---CGCCUG ..(---(---------((((((.(((((((((((((.....)))))))).....(((((....)))))))))).).))))))).(.(((.((((((((....)))))))).---)))).. ( -54.40) >consensus GAG___G________UCCCUAGCGCUGCGCAUGCGUAUGCGACAUGUGCGAGUGCGUCGAGUGCGGCGGCGGCACGUGCGGCACAUGCGCAUGGUGCGCAUGUCCGCCGUAGGCC___UG .....................((((((((((((((....)).))))))).)))))......(((((((((.((....)).)).(((((((.....)))))))..)))))))......... (-39.95 = -39.98 + 0.03)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:08:38 2006