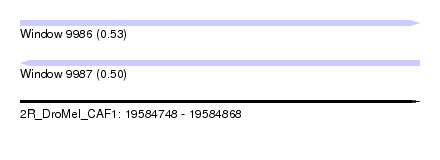
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 19,584,748 – 19,584,868 |
| Length | 120 |
| Max. P | 0.527176 |

| Location | 19,584,748 – 19,584,868 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.11 |
| Mean single sequence MFE | -52.12 |
| Consensus MFE | -31.20 |
| Energy contribution | -31.35 |
| Covariance contribution | 0.15 |
| Combinations/Pair | 1.41 |
| Mean z-score | -1.92 |
| Structure conservation index | 0.60 |
| SVM decision value | -0.01 |
| SVM RNA-class probability | 0.527176 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
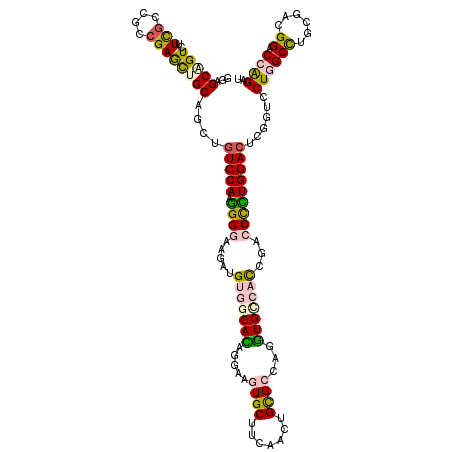
>2R_DroMel_CAF1 19584748 120 + 20766785 GGAGCGGUCUUUGCGGCCGAGCUGCAGCUGUCCAAGGGGAAGAUGUACCACAGGAAGUGCUUCAACUGCGCCAGGUGCACCCGACCCCUGGACUCGGUGCUCGCCUGUGACGGACCGGAU ....(((((((..(((.(((((.((....(((((.((((..(.((((((...((.(((......)))...)).))))))..)..)))))))))...))))))).)))..).))))))... ( -53.90) >DroPse_CAF1 90 120 + 1 GGAGCCGUUUUCGCCGCCGAGCUGCAGCUGUCCAAGGGGAAGAUGUGGCACAGGCAGUGCUUCAGCUGUGCCAGGUGCCACCGACCCCUGGACUCGGUCCUGGCCUGCGACGGUCCAGAU (((.(((((...((((((((((....)).(((((.((((..(..(((((((.((((..((....))..))))..))))))))..))))))))))))))...)))....)))))))).... ( -59.20) >DroWil_CAF1 96 120 + 1 GGGGCAGUAUUCGCUGCCGAAUUGCAAUUGUCCAAAGGAAAGAUGUGGCAUCAGAAGUGCUUCAAUUGUGCAAAAUGUCAUCGUCCUUUGGACUCUGUACUGGCUUGUGAUGGACCAGAU ..((((((....))))))....((((...((((((((((.....(((((((.....(..(.......)..)...)))))))..))))))))))..))))((((((......)).)))).. ( -42.80) >DroYak_CAF1 94 120 + 1 GGCGCGGUCUUUGCGGCCGAGCUGCAGUUGUCCAAGGGGAAGAUGUACCACCGGAAGUGCUUCAAAUGCGAGAGGUGCACCCGACCCUUGGACUCGGUGCUCGCCUGCGACGGACCGGAU ....((((((((((((.(((((.((....(((((((((...(.((((((..(....)(((.......)))...)))))).)...)))))))))...))))))).)))))).))))))... ( -57.30) >DroAna_CAF1 146 120 + 1 GGUGCAGUCUUUGCUGCCGAACUGCAGCUCUCCAAGGGAAAAAUGUGGCACAGGAAGUGCUUCAAUUGCGCCAGGUGCCACCGACCUCUGGACUCUGUCCUGGCCUGCGACGGUCCGGAU (.((((((..(((....))))))))).)..((((..((......(((((((..(..((((.......)))))..)))))))...))..))))....((((.((((......)))).)))) ( -42.50) >DroPer_CAF1 90 120 + 1 GGAGCCGUAUUCGCCGCAGAGCUGCAGCUGUCCAAGGGGAAGAUGUGGCAUAGGCAAUGCUUCAGCUGUGCCAGGUGCCACCGACCCCUGGACUCGGUCCUGGCCUGCGACGGUCCAGAU (((.((((......(((((.((((..((((((((.((((..(..(((((((.((((..((....))..))))..))))))))..)))))))))..)))..)))))))))))))))).... ( -57.00) >consensus GGAGCAGUCUUCGCCGCCGAGCUGCAGCUGUCCAAGGGGAAGAUGUGGCACAGGAAGUGCUUCAACUGCGCCAGGUGCCACCGACCCCUGGACUCGGUCCUGGCCUGCGACGGACCAGAU ...(((((..(((....))))))))....(((((.((((.....(((((((.....((((.......))))...)))))))...)))))))))......((((((......)).)))).. (-31.20 = -31.35 + 0.15)

| Location | 19,584,748 – 19,584,868 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.11 |
| Mean single sequence MFE | -48.35 |
| Consensus MFE | -28.47 |
| Energy contribution | -29.58 |
| Covariance contribution | 1.12 |
| Combinations/Pair | 1.37 |
| Mean z-score | -1.93 |
| Structure conservation index | 0.59 |
| SVM decision value | -0.06 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
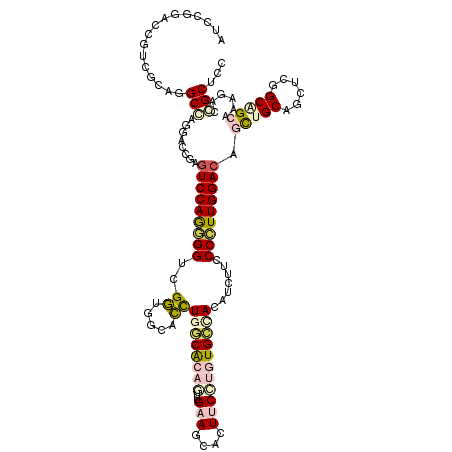
>2R_DroMel_CAF1 19584748 120 - 20766785 AUCCGGUCCGUCACAGGCGAGCACCGAGUCCAGGGGUCGGGUGCACCUGGCGCAGUUGAAGCACUUCCUGUGGUACAUCUUCCCCUUGGACAGCUGCAGCUCGGCCGCAAAGACCGCUCC ...((((((((.....))).((.(((((((((((((..(((((.(((....((((..(((....)))))))))).)))))..))))))))).((....))))))..))...))))).... ( -48.70) >DroPse_CAF1 90 120 - 1 AUCUGGACCGUCGCAGGCCAGGACCGAGUCCAGGGGUCGGUGGCACCUGGCACAGCUGAAGCACUGCCUGUGCCACAUCUUCCCCUUGGACAGCUGCAGCUCGGCGGCGAAAACGGCUCC .(((((.((......))))))).(((.(((((((((...(((((((..((((..((....))..)))).))))))).....)))).))))).(((((......))))).....))).... ( -59.70) >DroWil_CAF1 96 120 - 1 AUCUGGUCCAUCACAAGCCAGUACAGAGUCCAAAGGACGAUGACAUUUUGCACAAUUGAAGCACUUCUGAUGCCACAUCUUUCCUUUGGACAAUUGCAAUUCGGCAGCGAAUACUGCCCC ..(((((.........)))))......((((((((((.((((.((((.(((.........))).....))))...))))..))))))))))...........(((((......))))).. ( -34.00) >DroYak_CAF1 94 120 - 1 AUCCGGUCCGUCGCAGGCGAGCACCGAGUCCAAGGGUCGGGUGCACCUCUCGCAUUUGAAGCACUUCCGGUGGUACAUCUUCCCCUUGGACAACUGCAGCUCGGCCGCAAAGACCGCGCC ...(((((..(.((.(.(((((..((.(((((((((..(((((.(((.(.((.....(((....)))))).))).)))))..)))))))))...))..))))).).)))..))))).... ( -47.60) >DroAna_CAF1 146 120 - 1 AUCCGGACCGUCGCAGGCCAGGACAGAGUCCAGAGGUCGGUGGCACCUGGCGCAAUUGAAGCACUUCCUGUGCCACAUUUUUCCCUUGGAGAGCUGCAGUUCGGCAGCAAAGACUGCACC ...(((((.((.((.((((.((((...))))...)))).(((((((..((.((.......)).))....)))))))...(((((...))))))).)).)))))((((......))))... ( -41.90) >DroPer_CAF1 90 120 - 1 AUCUGGACCGUCGCAGGCCAGGACCGAGUCCAGGGGUCGGUGGCACCUGGCACAGCUGAAGCAUUGCCUAUGCCACAUCUUCCCCUUGGACAGCUGCAGCUCUGCGGCGAAUACGGCUCC .(((((.((......))))))).(((.(((((((((...((((((...((((..((....))..))))..)))))).....)))).))))).((((((....)))))).....))).... ( -58.20) >consensus AUCCGGACCGUCGCAGGCCAGGACCGAGUCCAGGGGUCGGUGGCACCUGGCACAGUUGAAGCACUUCCUGUGCCACAUCUUCCCCUUGGACAGCUGCAGCUCGGCAGCAAAGACCGCUCC ................(((........(((((((((..((.....))((((((((..(((....))))))))))).......))))))))).(((((......)))))......)))... (-28.47 = -29.58 + 1.12)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:06:19 2006