
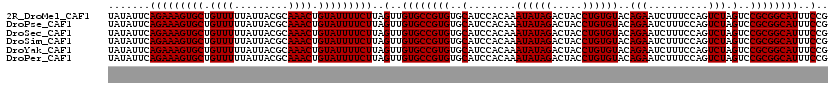
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 19,582,515 – 19,582,635 |
| Length | 120 |
| Max. P | 0.984611 |

| Location | 19,582,515 – 19,582,635 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 100.00 |
| Mean single sequence MFE | -27.50 |
| Consensus MFE | -27.50 |
| Energy contribution | -27.50 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.28 |
| Structure conservation index | 1.00 |
| SVM decision value | 1.98 |
| SVM RNA-class probability | 0.984611 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
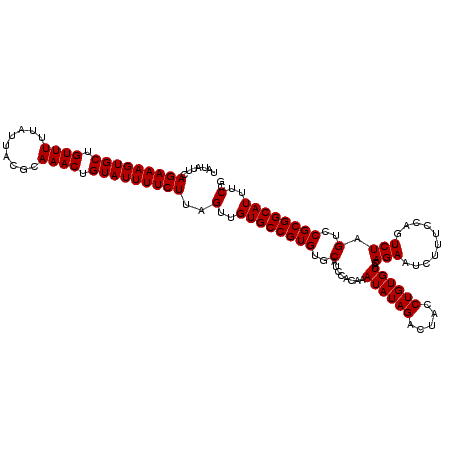
>2R_DroMel_CAF1 19582515 120 + 20766785 UAUAUUCAGAAAGUGCUGUUUUUAUUACGCAAACUGUAUUUUCUUAGUUGUGCCGUGUGCAUCCACAAAUAUAGACUACCUGUGUACAGAAUCUUUCCAGUCUAGUCCGCGGCAUUUCCG .......(((((((((.((((.........)))).)))))))))..(..((((((((..(........((((((.....))))))..(((..........))).)..))))))))..).. ( -27.50) >DroPse_CAF1 3485 120 + 1 UAUAUUCAGAAAGUGCUGUUUUUAUUACGCAAACUGUAUUUUCUUAGUUGUGCCGUGUGCAUCCACAAAUAUAGACUACCUGUGUACAGAAUCUUUCCAGUCUAGUCCGCGGCAUUUCCG .......(((((((((.((((.........)))).)))))))))..(..((((((((..(........((((((.....))))))..(((..........))).)..))))))))..).. ( -27.50) >DroSec_CAF1 5666 120 + 1 UAUAUUCAGAAAGUGCUGUUUUUAUUACGCAAACUGUAUUUUCUUAGUUGUGCCGUGUGCAUCCACAAAUAUAGACUACCUGUGUACAGAAUCUUUCCAGUCUAGUCCGCGGCAUUUCCG .......(((((((((.((((.........)))).)))))))))..(..((((((((..(........((((((.....))))))..(((..........))).)..))))))))..).. ( -27.50) >DroSim_CAF1 4861 120 + 1 UAUAUUCAGAAAGUGCUGUUUUUAUUACGCAAACUGUAUUUUCUUAGUUGUGCCGUGUGCAUCCACAAAUAUAGACUACCUGUGUACAGAAUCUUUCCAGUCUAGUCCGCGGCAUUUCCG .......(((((((((.((((.........)))).)))))))))..(..((((((((..(........((((((.....))))))..(((..........))).)..))))))))..).. ( -27.50) >DroYak_CAF1 5205 120 + 1 UAUAUUCAGAAAGUGCUGUUUUUAUUACGCAAACUGUAUUUUCUUAGUUGUGCCGUGUGCAUCCACAAAUAUAGACUACCUGUGUACAGAAUCUUUCCAGUCUAGUCCGCGGCAUUUCCG .......(((((((((.((((.........)))).)))))))))..(..((((((((..(........((((((.....))))))..(((..........))).)..))))))))..).. ( -27.50) >DroPer_CAF1 6557 120 + 1 UAUAUUCAGAAAGUGCUGUUUUUAUUACGCAAACUGUAUUUUCUUAGUUGUGCCGUGUGCAUCCACAAAUAUAGACUACCUGUGUACAGAAUCUUUCCAGUCUAGUCCGCGGCAUUUCCG .......(((((((((.((((.........)))).)))))))))..(..((((((((..(........((((((.....))))))..(((..........))).)..))))))))..).. ( -27.50) >consensus UAUAUUCAGAAAGUGCUGUUUUUAUUACGCAAACUGUAUUUUCUUAGUUGUGCCGUGUGCAUCCACAAAUAUAGACUACCUGUGUACAGAAUCUUUCCAGUCUAGUCCGCGGCAUUUCCG .......(((((((((.((((.........)))).)))))))))..(..((((((((..(........((((((.....))))))..(((..........))).)..))))))))..).. (-27.50 = -27.50 + 0.00)
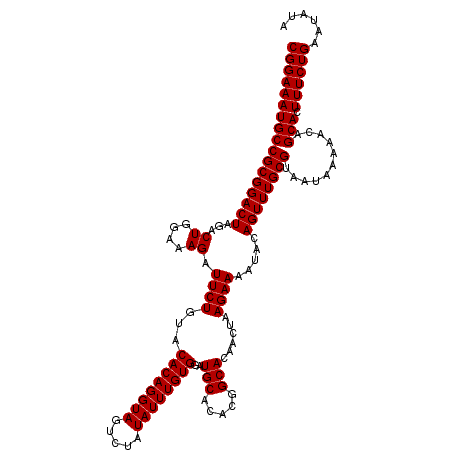
| Location | 19,582,515 – 19,582,635 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 100.00 |
| Mean single sequence MFE | -27.80 |
| Consensus MFE | -27.80 |
| Energy contribution | -27.80 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.22 |
| Structure conservation index | 1.00 |
| SVM decision value | 1.80 |
| SVM RNA-class probability | 0.977974 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 19582515 120 - 20766785 CGGAAAUGCCGCGGACUAGACUGGAAAGAUUCUGUACACAGGUAGUCUAUAUUUGUGGAUGCACACGGCACAACUAAGAAAAUACAGUUUGCGUAAUAAAAACAGCACUUUCUGAAUAUA (((((((((((((((((...((....)).((((...((((((((.....))))))))..(((.....)))......)))).....))))))))...........))).))))))...... ( -27.80) >DroPse_CAF1 3485 120 - 1 CGGAAAUGCCGCGGACUAGACUGGAAAGAUUCUGUACACAGGUAGUCUAUAUUUGUGGAUGCACACGGCACAACUAAGAAAAUACAGUUUGCGUAAUAAAAACAGCACUUUCUGAAUAUA (((((((((((((((((...((....)).((((...((((((((.....))))))))..(((.....)))......)))).....))))))))...........))).))))))...... ( -27.80) >DroSec_CAF1 5666 120 - 1 CGGAAAUGCCGCGGACUAGACUGGAAAGAUUCUGUACACAGGUAGUCUAUAUUUGUGGAUGCACACGGCACAACUAAGAAAAUACAGUUUGCGUAAUAAAAACAGCACUUUCUGAAUAUA (((((((((((((((((...((....)).((((...((((((((.....))))))))..(((.....)))......)))).....))))))))...........))).))))))...... ( -27.80) >DroSim_CAF1 4861 120 - 1 CGGAAAUGCCGCGGACUAGACUGGAAAGAUUCUGUACACAGGUAGUCUAUAUUUGUGGAUGCACACGGCACAACUAAGAAAAUACAGUUUGCGUAAUAAAAACAGCACUUUCUGAAUAUA (((((((((((((((((...((....)).((((...((((((((.....))))))))..(((.....)))......)))).....))))))))...........))).))))))...... ( -27.80) >DroYak_CAF1 5205 120 - 1 CGGAAAUGCCGCGGACUAGACUGGAAAGAUUCUGUACACAGGUAGUCUAUAUUUGUGGAUGCACACGGCACAACUAAGAAAAUACAGUUUGCGUAAUAAAAACAGCACUUUCUGAAUAUA (((((((((((((((((...((....)).((((...((((((((.....))))))))..(((.....)))......)))).....))))))))...........))).))))))...... ( -27.80) >DroPer_CAF1 6557 120 - 1 CGGAAAUGCCGCGGACUAGACUGGAAAGAUUCUGUACACAGGUAGUCUAUAUUUGUGGAUGCACACGGCACAACUAAGAAAAUACAGUUUGCGUAAUAAAAACAGCACUUUCUGAAUAUA (((((((((((((((((...((....)).((((...((((((((.....))))))))..(((.....)))......)))).....))))))))...........))).))))))...... ( -27.80) >consensus CGGAAAUGCCGCGGACUAGACUGGAAAGAUUCUGUACACAGGUAGUCUAUAUUUGUGGAUGCACACGGCACAACUAAGAAAAUACAGUUUGCGUAAUAAAAACAGCACUUUCUGAAUAUA (((((((((((((((((...((....)).((((...((((((((.....))))))))..(((.....)))......)))).....))))))))...........))).))))))...... (-27.80 = -27.80 + 0.00)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:06:17 2006