
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 19,565,800 – 19,565,953 |
| Length | 153 |
| Max. P | 0.999789 |

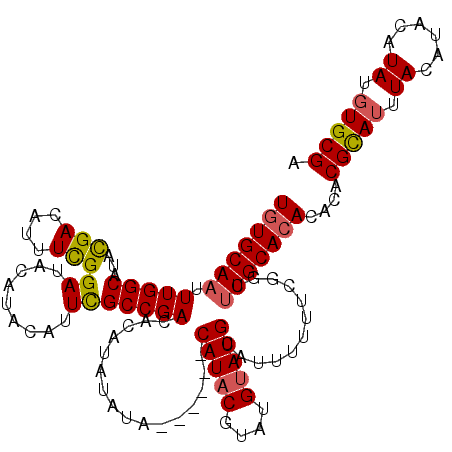
| Location | 19,565,800 – 19,565,914 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 89.86 |
| Mean single sequence MFE | -26.04 |
| Consensus MFE | -19.06 |
| Energy contribution | -19.14 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.14 |
| Mean z-score | -1.76 |
| Structure conservation index | 0.73 |
| SVM decision value | 0.14 |
| SVM RNA-class probability | 0.601859 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 19565800 114 + 20766785 UAUGCAAUUUGGCAUAUGACAUUUCGGAUACAUACAUUUGCCGACACAUAUAUA------CAUACGUAUGUAUGCAUUUUUUCGGCUUGCACACACACGUAUUUACAUACAUAUGUGCGA ..(((((.((((((.(((................))).))))))..((((((((------(....)))))))))............)))))..(.(((((((........))))))).). ( -26.19) >DroSec_CAF1 5213 113 + 1 UGUGCAAUUUGGCAUACGACAUUUCGGAUACAAACAUUCGCCGACACAUAUAGA------CAUACGUAUGUAUGCAUUU-UUCUGCUUGCACACACACGCAUUUACGUACAUAUGUGCGA (((((((.(((((...(((....)))((.........))))))).((((((...------.....))))))..(((...-...))))))))))............(((((....))))). ( -25.80) >DroSim_CAF1 5196 113 + 1 UGUGCA-UUUGGCAUACGACAUUUCGGAUACAAACAUUCGCCGACACAUAUAGA------CAUACGUAUGUAUGCAUUUUUUCGGCUUGCACACACACGCAUUUACGUACAUAUGUGCGA ((((((-((((.....(((....)))....)))).....(((((..((((((.(------(....)).)))))).......))))).))))))............(((((....))))). ( -27.10) >DroEre_CAF1 5245 114 + 1 UGUGCAAUUUGGCAUACGACAUUUUGGAUACAUACAUUCGCCGACACGUAUAUA------CAUACAUAUGUAUGCAUUUUUUCGGUUUGCACACGCUCGUAUUUACAUACAUAUGUGCGA (((((((..................((((......))))(((((...((.((((------(((....))))))).))....))))))))))))(((.(((((........))))).))). ( -26.20) >DroYak_CAF1 5382 120 + 1 UGUGCAAUUUGGCAUACGACAUUUCGGAUACAUACAUUCGCCGAUACGUAUAUACAUAUACAUACAUAUGAAUGCAUUUUUUCGGCUUGCACACACUCGCACUUACAUACAUAUUUGCGA (((((((.........(((....))).............(((((...((.(((.(((((......))))).))).))....))))))))))))...(((((..((......))..))))) ( -24.90) >consensus UGUGCAAUUUGGCAUACGACAUUUCGGAUACAUACAUUCGCCGACACAUAUAUA______CAUACGUAUGUAUGCAUUUUUUCGGCUUGCACACACACGCAUUUACAUACAUAUGUGCGA (((((((.(((((...(((....)))((.........)))))))................(((((....)))))............)))))))....(((((.((......)).))))). (-19.06 = -19.14 + 0.08)

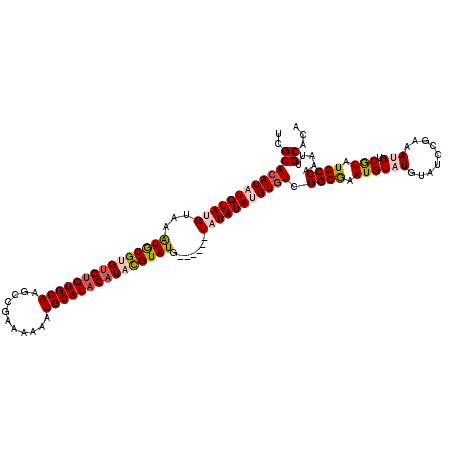
| Location | 19,565,800 – 19,565,914 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 89.86 |
| Mean single sequence MFE | -36.70 |
| Consensus MFE | -31.00 |
| Energy contribution | -32.24 |
| Covariance contribution | 1.24 |
| Combinations/Pair | 1.13 |
| Mean z-score | -3.95 |
| Structure conservation index | 0.84 |
| SVM decision value | 4.08 |
| SVM RNA-class probability | 0.999789 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 19565800 114 - 20766785 UCGCACAUAUGUAUGUAAAUACGUGUGUGUGCAAGCCGAAAAAAUGCAUACAUACGUAUG------UAUAUAUGUGUCGGCAAAUGUAUGUAUCCGAAAUGUCAUAUGCCAAAUUGCAUA ..(((((((((((((...(((((((((((((((...........))))))))))))))).------))))))))))).((((.((((((.........))).))).)))).....))... ( -40.70) >DroSec_CAF1 5213 113 - 1 UCGCACAUAUGUACGUAAAUGCGUGUGUGUGCAAGCAGAA-AAAUGCAUACAUACGUAUG------UCUAUAUGUGUCGGCGAAUGUUUGUAUCCGAAAUGUCGUAUGCCAAAUUGCACA .((.(((((((((.(...(((((((((((((((.......-...))))))))))))))).------.))))))))))))((((...((.((((.(((....))).)))).)).))))... ( -38.30) >DroSim_CAF1 5196 113 - 1 UCGCACAUAUGUACGUAAAUGCGUGUGUGUGCAAGCCGAAAAAAUGCAUACAUACGUAUG------UCUAUAUGUGUCGGCGAAUGUUUGUAUCCGAAAUGUCGUAUGCCAAA-UGCACA (((((((((((((.(...(((((((((((((((...........))))))))))))))).------.))))))))))..))))..((((((((.(((....))).))).))))-)..... ( -38.30) >DroEre_CAF1 5245 114 - 1 UCGCACAUAUGUAUGUAAAUACGAGCGUGUGCAAACCGAAAAAAUGCAUACAUAUGUAUG------UAUAUACGUGUCGGCGAAUGUAUGUAUCCAAAAUGUCGUAUGCCAAAUUGCACA ..(((.(((((((((((.(((((...(((((((...........)))))))...))))).------))))))))))).((((.((((((.........))).))).))))....)))... ( -31.00) >DroYak_CAF1 5382 120 - 1 UCGCAAAUAUGUAUGUAAGUGCGAGUGUGUGCAAGCCGAAAAAAUGCAUUCAUAUGUAUGUAUAUGUAUAUACGUAUCGGCGAAUGUAUGUAUCCGAAAUGUCGUAUGCCAAAUUGCACA (((((..(((....)))..)))))...(((((((.........(((((((((((((((((((....)))))))))))....))))))))((((.(((....))).))))....))))))) ( -35.20) >consensus UCGCACAUAUGUAUGUAAAUGCGUGUGUGUGCAAGCCGAAAAAAUGCAUACAUACGUAUG______UAUAUAUGUGUCGGCGAAUGUAUGUAUCCGAAAUGUCGUAUGCCAAAUUGCACA ..(((((((((((((...(((((((((((((((...........))))))))))))))).......))))))))))).((((.((((((.........))).))).)))).....))... (-31.00 = -32.24 + 1.24)


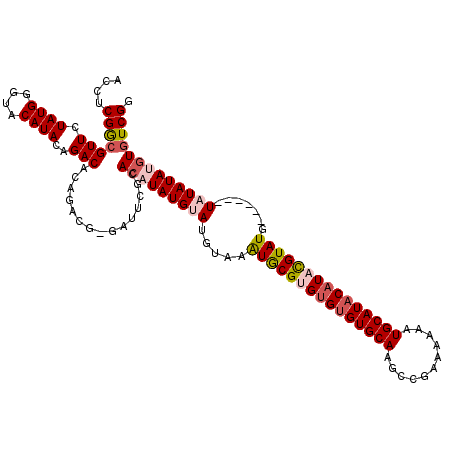
| Location | 19,565,840 – 19,565,953 |
|---|---|
| Length | 113 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 88.72 |
| Mean single sequence MFE | -35.60 |
| Consensus MFE | -27.86 |
| Energy contribution | -29.06 |
| Covariance contribution | 1.20 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.95 |
| Structure conservation index | 0.78 |
| SVM decision value | 2.41 |
| SVM RNA-class probability | 0.993558 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 19565840 113 - 20766785 ACCUCGGCGUUCUAUGGGUACAUACAGACACAGACG-GAUUCGCACAUAUGUAUGUAAAUACGUGUGUGUGCAAGCCGAAAAAAUGCAUACAUACGUAUG------UAUAUAUGUGUCGG .((.........((((....))))..((((((..((-(.(((((((((((((((....))))))))))))).)).))).....((((((((....)))))------)))...)))))))) ( -40.30) >DroSec_CAF1 5253 113 - 1 ACAUCGGCGUUCUAUGCAUACAUACAGACACAGGCGAGAUUCGCACAUAUGUACGUAAAUGCGUGUGUGUGCAAGCAGAA-AAAUGCAUACAUACGUAUG------UCUAUAUGUGUCGG ....((((((.....))..(((((.(((((((.(((.....))).....)))......(((((((((((((((.......-...))))))))))))))))------))).))))))))). ( -38.60) >DroSim_CAF1 5235 114 - 1 ACCUCGACGUUCUAUGCAUACAUACAGACACAGACGGGAUUCGCACAUAUGUACGUAAAUGCGUGUGUGUGCAAGCCGAAAAAAUGCAUACAUACGUAUG------UCUAUAUGUGUCGG .((((...(((.((((....))))..)))...)).))....((.(((((((((.(...(((((((((((((((...........))))))))))))))).------.)))))))))))). ( -35.30) >DroEre_CAF1 5285 113 - 1 ACCUCGGCGUUCUAUGGGUACAUACAGACACAGACC-AAUUCGCACAUAUGUAUGUAAAUACGAGCGUGUGCAAACCGAAAAAAUGCAUACAUAUGUAUG------UAUAUACGUGUCGG .((.........((((....))))..(((((.....-.....((((((.(((((....)))))...))))))...........((((((((....)))))------)))....))))))) ( -29.20) >DroYak_CAF1 5422 119 - 1 ACCUCGGCGUUCUAUGGGUACAUACAGACACAGACG-GAUUCGCAAAUAUGUAUGUAAGUGCGAGUGUGUGCAAGCCGAAAAAAUGCAUUCAUAUGUAUGUAUAUGUAUAUACGUAUCGG .((((((((((.((((....))))..)))....(((-.(((((((..(((....)))..))))))).)))....)))))............(((((((((((....))))))))))).)) ( -34.60) >consensus ACCUCGGCGUUCUAUGGGUACAUACAGACACAGACG_GAUUCGCACAUAUGUAUGUAAAUGCGUGUGUGUGCAAGCCGAAAAAAUGCAUACAUACGUAUG______UAUAUAUGUGUCGG ....(((((((.((((....))))..)))...............(((((((((.....(((((((((((((((...........))))))))))))))).......))))))))))))). (-27.86 = -29.06 + 1.20)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:06:07 2006