| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 19,421,332 – 19,421,435 |
| Length | 103 |
| Max. P | 0.937885 |

| Location | 19,421,332 – 19,421,435 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 110 |
| Reading direction | forward |
| Mean pairwise identity | 80.39 |
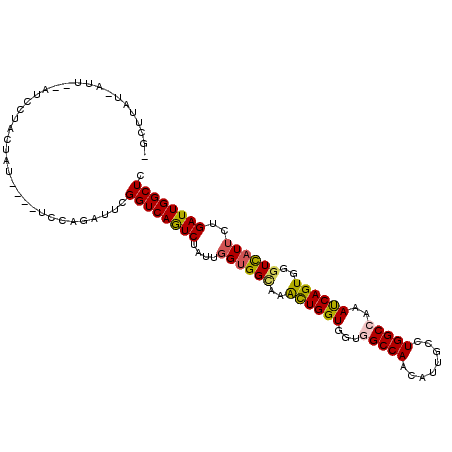
| Mean single sequence MFE | -32.22 |
| Consensus MFE | -27.53 |
| Energy contribution | -27.32 |
| Covariance contribution | -0.22 |
| Combinations/Pair | 1.24 |
| Mean z-score | -2.14 |
| Structure conservation index | 0.85 |
| SVM decision value | 1.26 |
| SVM RNA-class probability | 0.937885 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 19421332 103 + 20766785 -GCUUAUCAUC--AUCCAACUAU----UCCAGAUUCGGUCAGUCUAUUGGUGGCAAGCUGGUGGUGGCCAAUAUUGCCUGGCCAAAUCAGUGGGUCAUUCUGAUUGGCUC -..........--..........----.........((((((((....((((((..((((((..((((((........)))))).))))))..))))))..)))))))). ( -34.30) >DroSim_CAF1 4029 104 + 1 UGCUUAUCAUU--AACCUUCUAU----UCCAGAUUCGGUCAGUCUAUCGGUGGCAAGCUGGUGGUGGCCAAUAUCGCCUGGCCAAAUCAGUGGGUCAUUCUGAUUGGCUC ...........--..........----.........((((((((....((((((..((((((..((((((........)))))).))))))..))))))..)))))))). ( -34.30) >DroEre_CAF1 4147 100 + 1 -GCUUAU---U--ACGCUACUAU----UCCAGAUUCGGUCAGUCUAUUGGUGGCAAACUGGUGGUGGCCAACAUAGCCUGGCCAAAUCAGUGGGUCAUUCUGAUUGGCUC -((....---.--..))......----.........((((((((....((((((..((((((..((((((........)))))).))))))..))))))..)))))))). ( -34.10) >DroWil_CAF1 5643 99 + 1 UGUUUGUUAUU--AUAC-----U----UAUAGAUUUGGUCAAUCGAUUGGAGGUAAAUUGGUUGUCGCCAAUAUUGCGUGGCCCAAUCAGUGGGUUAUAUUGAUUGGCUC ...........--....-----.----.........(((((((((((.....((((((((((....)))))).))))((((((((.....))))))))))))))))))). ( -28.30) >DroYak_CAF1 4174 100 + 1 -UCUUAU---U--UCCCUGCUAU----UCCAGAUUCGGUCAGUCUAUUGGUGGCAAACUGGUAGUAGCCAACAUUGCCUGGCCAAAUCAGUGGGUCAUUCUGAUUGGCUC -......---.--...(((....----..)))....((((((((....((((((..((((((....((((........))))...))))))..))))))..)))))))). ( -28.00) >DroAna_CAF1 4002 109 + 1 -GCUUACUUAUAUUUUCAAAUAUCCCAUUCAGAUUCGGUCAAUCCAUUGGUGGAAAAUUGGUGGUGGCCAACAUUGCCUGGCCCAACCAGUGGGUCAUUCUGAUUGGCUC -.......................(((.(((((...(((((..((((..((.....))..))))))))).........(((((((.....))))))).))))).)))... ( -34.30) >consensus _GCUUAU_AUU__AUCCUACUAU____UCCAGAUUCGGUCAGUCUAUUGGUGGCAAACUGGUGGUGGCCAACAUUGCCUGGCCAAAUCAGUGGGUCAUUCUGAUUGGCUC ....................................((((((((....((((((..((((((...(((((........)))))..))))))..))))))..)))))))). (-27.53 = -27.32 + -0.22)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:04:40 2006