
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 19,021,619 – 19,021,745 |
| Length | 126 |
| Max. P | 0.748868 |

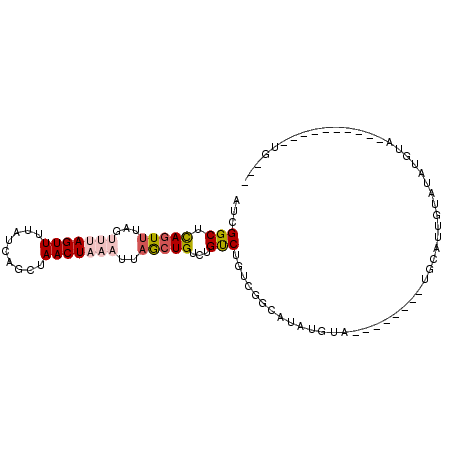
| Location | 19,021,619 – 19,021,714 |
|---|---|
| Length | 95 |
| Sequences | 6 |
| Columns | 103 |
| Reading direction | reverse |
| Mean pairwise identity | 70.29 |
| Mean single sequence MFE | -21.27 |
| Consensus MFE | -8.90 |
| Energy contribution | -9.73 |
| Covariance contribution | 0.83 |
| Combinations/Pair | 1.20 |
| Mean z-score | -1.81 |
| Structure conservation index | 0.42 |
| SVM decision value | 0.40 |
| SVM RNA-class probability | 0.721571 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
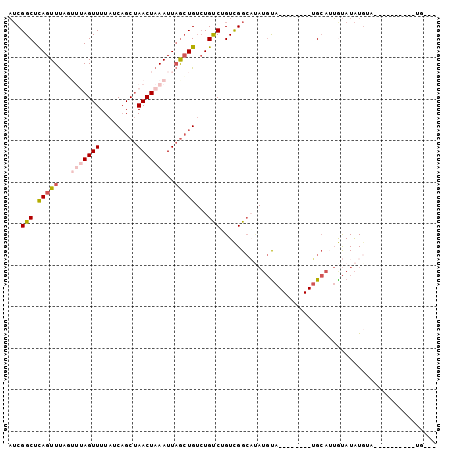
>2R_DroMel_CAF1 19021619 95 - 20766785 AUCGGCUCAGUUUAGUUUAGUUUUAUCAGCUAACUAAAUUAGCUGUCUGCCUGUCGGCAUAUGUA--------UGCAUUGUAUAUGUAUAUGAAUGGCUGUGU ..(((((.(((((((((.((((.....))))))))))))))))))...(((..(((..(((((((--------(((...)))))))))).)))..)))..... ( -27.60) >DroSec_CAF1 119511 77 - 1 AUCGGCUCAGUUUAGUUUAGUUUUAUCCGCUAACUAAAUUAGCUGUCUGUCUGUCGGCAUAUGCA--------UGCAUUGUAUAU------------------ ..(((((.(((((((((.(((.......)))))))))))))))))..((((....))))((((((--------.....)))))).------------------ ( -19.80) >DroSim_CAF1 124118 92 - 1 AUCGGCUCAGUUUAGUUUAGUUUUAUCAGCUAACUAAAUUAGCUGUCUGUCUGUCGGCAUAUGUA--------UGCAUUGUAUAUGUAUAUGAACGGAAG--- ..(((((.(((((((((.((((.....))))))))))))))))))....(((((((..(((((((--------(((...)))))))))).)).)))))..--- ( -26.30) >DroEre_CAF1 123003 78 - 1 AUCGGCUCAGUUU-----AGUUUUAUCAGCUAACUAAAUUAGCUGUCUGUCUGUUGGCUCAUGUA--------UGCAGUAUAUAUG------------UAAGU ..(((((.(((((-----((((.........))))))))))))))...(((....))).((((((--------((...))))))))------------..... ( -17.00) >DroYak_CAF1 124156 94 - 1 AUCGGCUCAGUUU-----AGUUUUAUCAGCUAACUAAAUUAGCUGUCUGCCGGUUGGCAUAUGUAUGUACAUAUGCAGUAUAUAUGAAUGUG----UCUGAGU ((((((.((((((-----((((.........))))))....))))...)))))).(((((((((((((((.......))))))))..)))))----))..... ( -26.50) >DroPer_CAF1 123420 77 - 1 UACGGCUUAGUUCACUUUAGUUUUAUCAGCCAACUAAAUUUAGUG--UGUCAGUCGGAU---CUA--------GGAGUAGCAUGUGUG----------UG--- ..(((((.....((((((((((.........))))))....))))--....)))))...---...--------......(((....))----------).--- ( -10.40) >consensus AUCGGCUCAGUUUAGUUUAGUUUUAUCAGCUAACUAAAUUAGCUGUCUGUCUGUCGGCAUAUGUA________UGCAUUGUAUAUGUA__________UG___ ...(((.(((((...(((((((.........)))))))..)))))...))).................................................... ( -8.90 = -9.73 + 0.83)

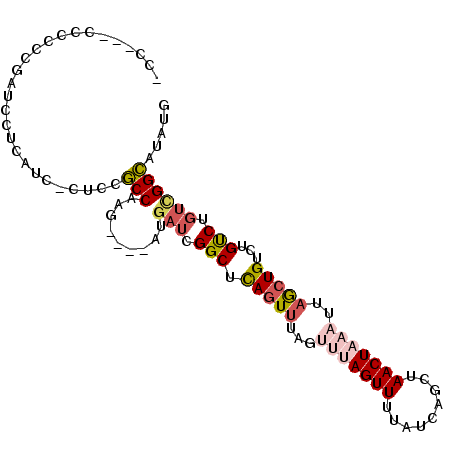
| Location | 19,021,651 – 19,021,745 |
|---|---|
| Length | 94 |
| Sequences | 6 |
| Columns | 103 |
| Reading direction | reverse |
| Mean pairwise identity | 72.56 |
| Mean single sequence MFE | -21.85 |
| Consensus MFE | -11.98 |
| Energy contribution | -12.87 |
| Covariance contribution | 0.89 |
| Combinations/Pair | 1.24 |
| Mean z-score | -1.59 |
| Structure conservation index | 0.55 |
| SVM decision value | 0.47 |
| SVM RNA-class probability | 0.748868 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
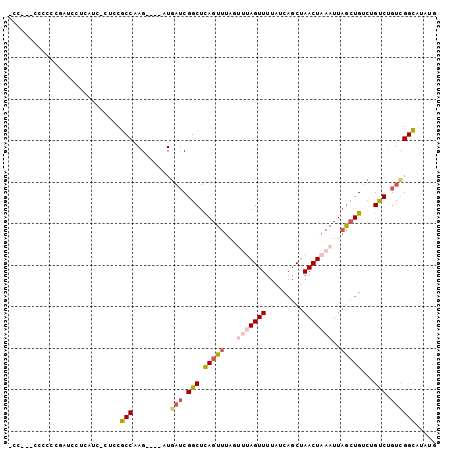
>2R_DroMel_CAF1 19021651 94 - 20766785 -CC---CCCCCCGUUCCACAUC-CUUCGCCAAG----AUGAUCGGCUCAGUUUAGUUUAGUUUUAUCAGCUAACUAAAUUAGCUGUCUGCCUGUCGGCAUAUG -..---................-....(((...----..(((.(((.(((((.(((((((((.........))))))))))))))...))).))))))..... ( -22.30) >DroSec_CAF1 119525 96 - 1 -CCGA-CCCCCUUAUCCUUGUC-CUUCGCCAAG----AUGAUCGGCUCAGUUUAGUUUAGUUUUAUCCGCUAACUAAAUUAGCUGUCUGUCUGUCGGCAUAUG -....-................-....(((.((----(((..(((((.(((((((((.(((.......)))))))))))))))))..)))))...)))..... ( -24.10) >DroSim_CAF1 124147 96 - 1 -CCGA-CCCCCCUACCCUCGUC-CUUCGCCAAG----AUGAUCGGCUCAGUUUAGUUUAGUUUUAUCAGCUAACUAAAUUAGCUGUCUGUCUGUCGGCAUAUG -....-................-....(((.((----(((..(((((.(((((((((.((((.....))))))))))))))))))..)))))...)))..... ( -25.00) >DroEre_CAF1 123023 76 - 1 ---------------C--CGAC-UUCCGCCAAG----AUGAUCGGCUCAGUUU-----AGUUUUAUCAGCUAACUAAAUUAGCUGUCUGUCUGUUGGCUCAUG ---------------.--....-....((((((----(((..(((((.(((((-----((((.........))))))))))))))..)))))..))))..... ( -22.30) >DroYak_CAF1 124192 91 - 1 ACC-G-CCCCCGCCCCCGCAUC-CACCGCCAAG----AUGAUCGGCUCAGUUU-----AGUUUUAUCAGCUAACUAAAUUAGCUGUCUGCCGGUUGGCAUAUG ...-(-((...((....))...-.((((...((----(....(((((.(((((-----((((.........)))))))))))))))))..)))).)))..... ( -23.10) >DroPer_CAF1 123439 96 - 1 -U-GUACUAUCUGUUCUCCAUCGCUCCUCCUAGCAGCCUCUACGGCUUAGUUCACUUUAGUUUUAUCAGCCAACUAAAUUUAGUG--UGUCAGUCGGAU---C -.-.....................(((..((..(((((.....)))......((((((((((.........))))))....))))--))..))..))).---. ( -14.30) >consensus _CC___CCCCCCGAUCCUCAUC_CUCCGCCAAG____AUGAUCGGCUCAGUUUAGUUUAGUUUUAUCAGCUAACUAAAUUAGCUGUCUGUCUGUCGGCAUAUG ...........................(((.........(((.(((.(((((...(((((((.........)))))))..)))))...))).))))))..... (-11.98 = -12.87 + 0.89)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:01:12 2006