
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 3,069,869 – 3,069,999 |
| Length | 130 |
| Max. P | 0.836540 |

| Location | 3,069,869 – 3,069,962 |
|---|---|
| Length | 93 |
| Sequences | 6 |
| Columns | 93 |
| Reading direction | forward |
| Mean pairwise identity | 85.59 |
| Mean single sequence MFE | -37.13 |
| Consensus MFE | -27.90 |
| Energy contribution | -30.40 |
| Covariance contribution | 2.50 |
| Combinations/Pair | 1.27 |
| Mean z-score | -2.62 |
| Structure conservation index | 0.75 |
| SVM decision value | 0.34 |
| SVM RNA-class probability | 0.697137 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 3069869 93 + 20766785 AUCAGCUCCAGUCCGGGCUACAACUCCAAGCUUAUGGAAGCGCCACUGCUGGGUUCCCAGCAGGGGGGACGUAAGUCUAAUGCACCCGGCAGC ....((((((....(((((.........))))).))).)))(((.(((((((....)))))))(((((((....))))......))))))... ( -39.00) >DroVir_CAF1 20030 93 + 1 AUUAGCUCCAGUCCCGGCUAUAAUUCCAAGCUCAUGGAGACGCCGUUGCUGGCAUCGGCGCAGAGCGCACGCAAGAGCCAAGCUCCCAGCGUG ....((((((((..((((.....(((((......)))))..))))..))))((......)).)))).(((((..((((...))))...))))) ( -33.30) >DroSec_CAF1 2459 93 + 1 AUCAGCUCCAGUCCGGGCUACAACUCUAAGCUUAUGGAAGCACCACUGCUGGGAUCCCAGCAGGGCGGACGUAAGUCCAAUGCACCCGGCAGC ....(((...(.(((((.......((((......)))).(((((.(((((((....))))))))).((((....))))..))).))))))))) ( -38.70) >DroSim_CAF1 2957 93 + 1 AUCAGCUCCAGUCCGGGCUACAACUCUAAGCUUAUGGAAGCACCACUGCUGGGAUCCCAGCAGGGCGGACGUAAGUCCAAUGCACCCGGCAGC ....(((...(.(((((.......((((......)))).(((((.(((((((....))))))))).((((....))))..))).))))))))) ( -38.70) >DroEre_CAF1 2908 93 + 1 AUCAGCUCCAGUCCGGGCUACAACUCCAAGCUCAUGGAAGCGCCACUGCUGGGCUCCCAACAAGGAGGGCGUAAGUCCAACGCACCCGGCACC ....((((((....(((((.........))))).))).))).....(((((((.(((......)))((((....))))......))))))).. ( -33.20) >DroYak_CAF1 2984 93 + 1 AUCAGCUCCAGUCCGGGCUACAACUCCAAGCUUAUGGAAGCGCCACUGCUGGGUUCCCAGCAAGGGGGACGUAAGUCCAACGCACCCGGCACC ..........(((((((.......((((......)))).(((((..((((((....))))))..))((((....))))..))).))))).)). ( -39.90) >consensus AUCAGCUCCAGUCCGGGCUACAACUCCAAGCUUAUGGAAGCGCCACUGCUGGGAUCCCAGCAGGGCGGACGUAAGUCCAAUGCACCCGGCAGC ..........(.(((((.......((((......)))).(((((.(((((((....))))))))).((((....))))..))).))))))... (-27.90 = -30.40 + 2.50)

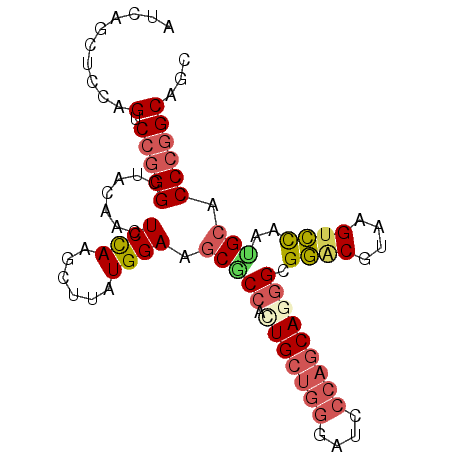
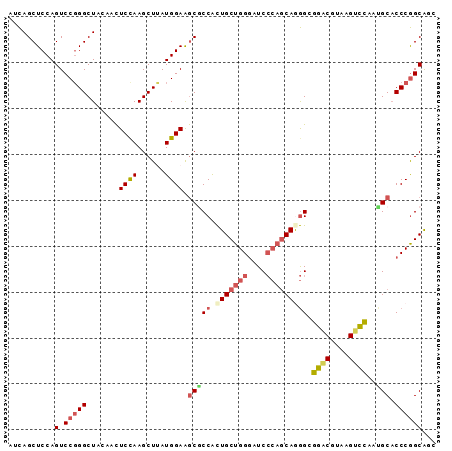
| Location | 3,069,909 – 3,069,999 |
|---|---|
| Length | 90 |
| Sequences | 6 |
| Columns | 90 |
| Reading direction | forward |
| Mean pairwise identity | 81.78 |
| Mean single sequence MFE | -37.65 |
| Consensus MFE | -24.90 |
| Energy contribution | -26.07 |
| Covariance contribution | 1.17 |
| Combinations/Pair | 1.33 |
| Mean z-score | -2.42 |
| Structure conservation index | 0.66 |
| SVM decision value | 0.73 |
| SVM RNA-class probability | 0.836540 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 3069909 90 + 20766785 CGCCACUGCUGGGUUCCCAGCAGGGGGGACGUAAGUCUAAUGCACCCGGCAGCGCACAGCGCCAGACUCCGCUCAUGGACUUGGCCCCCA .....(((((((....)))))))(((((.(.((((((((..((....(((.((.....))))).(....)))...)))))))))))))). ( -42.30) >DroVir_CAF1 20070 87 + 1 CGCCGUUGCUGGCAUCGGCGCAGAGCGCACGCAAGAGCCAAGCUCCCAGCGUGCUGCAGCGUCAG---CCUCUGAUGGACUUGGCACCAA ..((((((..(((...(((((...((((((((..((((...))))...)))))).)).))))).)---))..))))))............ ( -34.00) >DroSec_CAF1 2499 90 + 1 CACCACUGCUGGGAUCCCAGCAGGGCGGACGUAAGUCCAAUGCACCCGGCAGCGCACAGCGCCAGACUCCACUAAUGGACUUGGCCCCCA ..((.(((((((....))))))))).((((....))))..(((.....)))((.....))(((((..((((....)))).)))))..... ( -37.50) >DroSim_CAF1 2997 90 + 1 CACCACUGCUGGGAUCCCAGCAGGGCGGACGUAAGUCCAAUGCACCCGGCAGCGCACAGCGCCAGACUCCACUAAUGGACUUGGCCCCCA ..((.(((((((....))))))))).((((....))))..(((.....)))((.....))(((((..((((....)))).)))))..... ( -37.50) >DroEre_CAF1 2948 90 + 1 CGCCACUGCUGGGCUCCCAACAAGGAGGGCGUAAGUCCAACGCACCCGGCACCGCGCAGCGCCAAACUCCGCUCAUGGAUUUGGCCCCCA (((...(((((((.(((......)))((((....))))......)))))))..)))....((((((.((((....))))))))))..... ( -35.20) >DroYak_CAF1 3024 90 + 1 CGCCACUGCUGGGUUCCCAGCAAGGGGGACGUAAGUCCAACGCACCCGGCACCGCGCAGCGCCAAACUCCGCUCAUGGAUUUGGCUCCCA (((...((((((((.(((.....)))((((....)))).....))))))))..)))....((((((.((((....))))))))))..... ( -39.40) >consensus CGCCACUGCUGGGAUCCCAGCAGGGCGGACGUAAGUCCAAUGCACCCGGCAGCGCACAGCGCCAGACUCCGCUCAUGGACUUGGCCCCCA ......(((((((..((......)).((((....))))......))))))).........(((((..(((......))).)))))..... (-24.90 = -26.07 + 1.17)

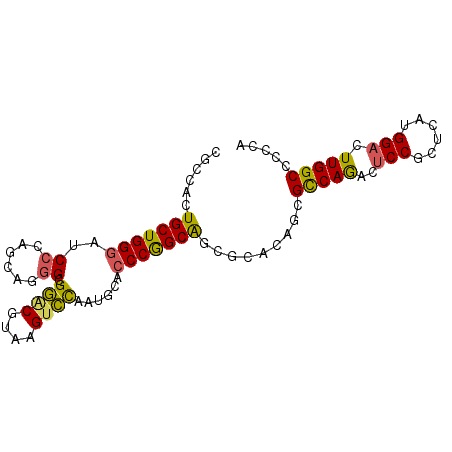
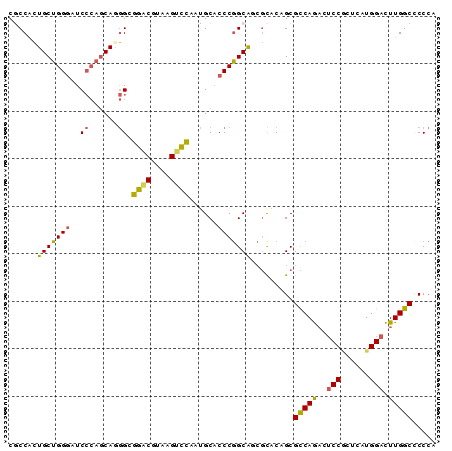
| Location | 3,069,909 – 3,069,999 |
|---|---|
| Length | 90 |
| Sequences | 6 |
| Columns | 90 |
| Reading direction | reverse |
| Mean pairwise identity | 81.78 |
| Mean single sequence MFE | -41.13 |
| Consensus MFE | -27.65 |
| Energy contribution | -28.40 |
| Covariance contribution | 0.75 |
| Combinations/Pair | 1.32 |
| Mean z-score | -1.45 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.01 |
| SVM RNA-class probability | 0.537056 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 3069909 90 - 20766785 UGGGGGCCAAGUCCAUGAGCGGAGUCUGGCGCUGUGCGCUGCCGGGUGCAUUAGACUUACGUCCCCCCUGCUGGGAACCCAGCAGUGGCG .((((((.(((((....((((........))))(((((((....)))))))..)))))..))))))((((((((....))))))).)... ( -40.90) >DroVir_CAF1 20070 87 - 1 UUGGUGCCAAGUCCAUCAGAGG---CUGACGCUGCAGCACGCUGGGAGCUUGGCUCUUGCGUGCGCUCUGCGCCGAUGCCAGCAACGGCG .(((........))).((((((---(.......)).((((((.((((((...)))))))))))).)))))(((((.((....)).))))) ( -36.20) >DroSec_CAF1 2499 90 - 1 UGGGGGCCAAGUCCAUUAGUGGAGUCUGGCGCUGUGCGCUGCCGGGUGCAUUGGACUUACGUCCGCCCUGCUGGGAUCCCAGCAGUGGUG ...((((.(((((((...(((.(.((((((((.....)).))))))).))))))))))..))))((((((((((....))))))).))). ( -44.60) >DroSim_CAF1 2997 90 - 1 UGGGGGCCAAGUCCAUUAGUGGAGUCUGGCGCUGUGCGCUGCCGGGUGCAUUGGACUUACGUCCGCCCUGCUGGGAUCCCAGCAGUGGUG ...((((.(((((((...(((.(.((((((((.....)).))))))).))))))))))..))))((((((((((....))))))).))). ( -44.60) >DroEre_CAF1 2948 90 - 1 UGGGGGCCAAAUCCAUGAGCGGAGUUUGGCGCUGCGCGGUGCCGGGUGCGUUGGACUUACGCCCUCCUUGUUGGGAGCCCAGCAGUGGCG .((((((....(((......)))(((..((((.((.((....)).))))))..)))....)))))).(((((((....)))))))..... ( -39.60) >DroYak_CAF1 3024 90 - 1 UGGGAGCCAAAUCCAUGAGCGGAGUUUGGCGCUGCGCGGUGCCGGGUGCGUUGGACUUACGUCCCCCUUGCUGGGAACCCAGCAGUGGCG .((((((((......)).)).(((((..((((.((.((....)).))))))..)))))...))))(((((((((....))))))).)).. ( -40.90) >consensus UGGGGGCCAAGUCCAUGAGCGGAGUCUGGCGCUGCGCGCUGCCGGGUGCAUUGGACUUACGUCCGCCCUGCUGGGAUCCCAGCAGUGGCG ...((((.(((((((...(((.(.(((((((........)))))))).))))))))))..)))).(((((((((....))))))).)).. (-27.65 = -28.40 + 0.75)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:42:37 2006