
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 3,066,268 – 3,066,443 |
| Length | 175 |
| Max. P | 0.938354 |

| Location | 3,066,268 – 3,066,376 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.71 |
| Mean single sequence MFE | -35.59 |
| Consensus MFE | -24.59 |
| Energy contribution | -24.98 |
| Covariance contribution | 0.39 |
| Combinations/Pair | 1.08 |
| Mean z-score | -1.79 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.18 |
| SVM RNA-class probability | 0.619524 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 3066268 108 - 20766785 CCGGGAUA---UACGU---------AUACGUGUAGCAUGUGCGGUGGGCAUAUUGAUGCCAUCGGAAUCGGGCCAAGCAAUUUAGUCUUUUGUGUCUAAUGGAUGCCAUUAGUCGGCUCG ((((..((---((((.---------...))))))((....))....(((((....))))).))))...((((((..((((.........))))(.(((((((...))))))).))))))) ( -35.60) >DroSec_CAF1 23051 107 - 1 CCGGGAUA---UACAU---------AUACG-AUGGCAUGUGCGGUGGGCAUAUUGAUGCCAUCGGAAUCGGGCCAAGCAAUUUGGUCUUUUGUGUCUAAUGGAUGCCAUUAGUCGGCUCG ........---.....---------...((-(((((((((((.....))))....)))))))))....((((((..((((.........))))(.(((((((...))))))).))))))) ( -36.40) >DroSim_CAF1 17724 108 - 1 CCGGGAUA---UACGU---------AUACGUAUGGCAUGUGCGGUGGGCAUAUUGAUGCCAUCGGAAUCGGGCCAAGCAAUUUGGUCUUUUGUGUCUAAUGGAUGCCAUUAGUCGGCUCG ((((...(---((((.---------...)))))(((((((((.....))))....))))).))))...((((((..((((.........))))(.(((((((...))))))).))))))) ( -36.30) >DroEre_CAF1 17542 100 - 1 --------------------CGUACAUACGUAUAGUAUGUGCGGUGGGCAUAUUGAUGCCAUCGGAAUCGGGCCAAGCAAUUUAGUCUUUUGUGUCUAAUGGAUGCCAUUAGUCGGCUCG --------------------(((((((((.....)))))))))...(((((....)))))........((((((..((((.........))))(.(((((((...))))))).))))))) ( -37.70) >DroYak_CAF1 17351 117 - 1 CCGGGAUA---UACAUAGUACAUACAUGCGUUUAGUAUGUGCGGUGGGCAUAUUGAUGCCAUCGGAAUCGGGCUAAGCAAUUUGGUCUUUUGUGUCUAAUGGAUGCCAUUAGUCGGCUCA ((((....---..(((.((((((((.........)))))))).)))(((((....))))).))))....(((((..((((.........))))(.(((((((...))))))).)))))). ( -37.80) >DroAna_CAF1 13757 106 - 1 CCGGCAUUGGGUAGCU---------AU-CUUCUAACAC--ACA--GUAUAUAUCGAUGCCAUCGGAAUCGGGCCAGGCAAUUUGGUCUUUUGUGUCUAAUGGAUGCCAUUAGUCGGCUCA ..((((((((...(((---------..-..........--..)--)).....)))))))).........(((((((((......)))).....(.(((((((...))))))).)))))). ( -29.74) >consensus CCGGGAUA___UACAU_________AUACGUAUAGCAUGUGCGGUGGGCAUAUUGAUGCCAUCGGAAUCGGGCCAAGCAAUUUGGUCUUUUGUGUCUAAUGGAUGCCAUUAGUCGGCUCG .........................................((((((.(((....))))))))).....(((((..((((.........))))(.(((((((...))))))).)))))). (-24.59 = -24.98 + 0.39)

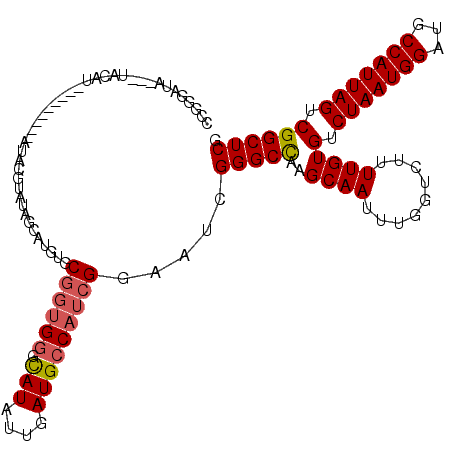
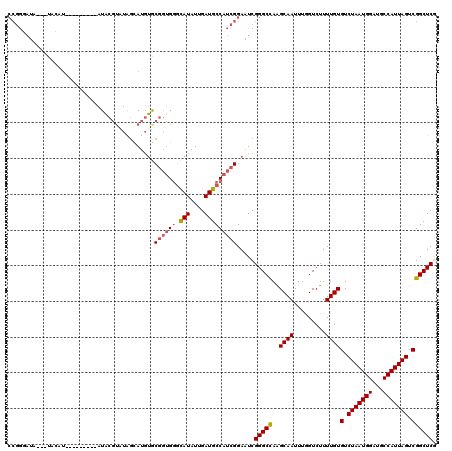
| Location | 3,066,308 – 3,066,410 |
|---|---|
| Length | 102 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 73.14 |
| Mean single sequence MFE | -33.49 |
| Consensus MFE | -12.28 |
| Energy contribution | -13.12 |
| Covariance contribution | 0.84 |
| Combinations/Pair | 1.25 |
| Mean z-score | -2.04 |
| Structure conservation index | 0.37 |
| SVM decision value | 1.16 |
| SVM RNA-class probability | 0.924869 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 3066308 102 - 20766785 AAAUAAAUGCAGUGUG------UGUCUGUGUGUCUGUGUGCCGGGAUA---UACGU---------AUACGUGUAGCAUGUGCGGUGGGCAUAUUGAUGCCAUCGGAAUCGGGCCAAGCAA ......((((.(..((------(((.(((((((((........)))))---)))).---------)))))..).)))).((((((((.(((....))))))))((.......))..))). ( -32.10) >DroSec_CAF1 23091 99 - 1 AAAUAAAUGCAGUGUGUCUGU--------GUGUCUGUGUACCGGGAUA---UACAU---------AUACG-AUGGCAUGUGCGGUGGGCAUAUUGAUGCCAUCGGAAUCGGGCCAAGCAA .......(((..(((((.(((--------((((((........)))))---)))).---------)))))-.((((...(.((((((.(((....))))))))).).....)))).))). ( -33.60) >DroSim_CAF1 17764 108 - 1 AAAUAAAUGCAGUGUGUCUGUGUGUCUGUGUGUCUGUGUACCGGGAUA---UACGU---------AUACGUAUGGCAUGUGCGGUGGGCAUAUUGAUGCCAUCGGAAUCGGGCCAAGCAA ........((((.....))))((((.(((((((((........)))))---)))).---------))))((.((((...(.((((((.(((....))))))))).).....)))).)).. ( -34.50) >DroYak_CAF1 17391 109 - 1 AAAUAAAUGCAGUGUGUCUGU--------GUGCUUUUGUGCCGGGAUA---UACAUAGUACAUACAUGCGUUUAGUAUGUGCGGUGGGCAUAUUGAUGCCAUCGGAAUCGGGCUAAGCAA ........(((((((((((((--------(((..(((......)))..---))))))).)))))).)))(((((((...(.((((((.(((....))))))))).).....))))))).. ( -37.00) >DroAna_CAF1 13797 103 - 1 AAAUAUGUGCAGGAACCCA---GGAAUGGGUUUCCAUAUGCCGGCAUUGGGUAGCU---------AU-CUUCUAACAC--ACA--GUAUAUAUCGAUGCCAUCGGAAUCGGGCCAGGCAA ....(((((..((((((((---....)))))))))))))(((((((((((...(((---------..-..........--..)--)).....))))))))...((.......)).))).. ( -30.24) >consensus AAAUAAAUGCAGUGUGUCUGU__G__UG_GUGUCUGUGUGCCGGGAUA___UACAU_________AUACGUAUAGCAUGUGCGGUGGGCAUAUUGAUGCCAUCGGAAUCGGGCCAAGCAA .......(((.(.....).............(((((....((((..............................((....))....(((((....))))).))))...)))))...))). (-12.28 = -13.12 + 0.84)

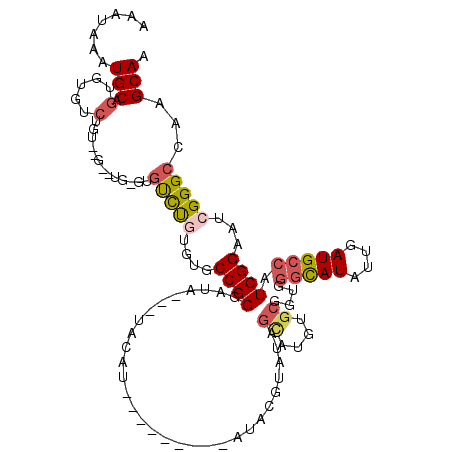
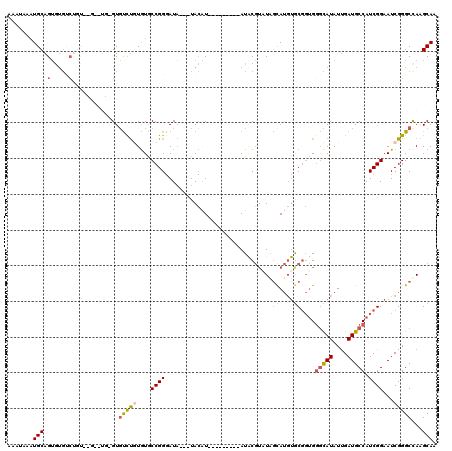
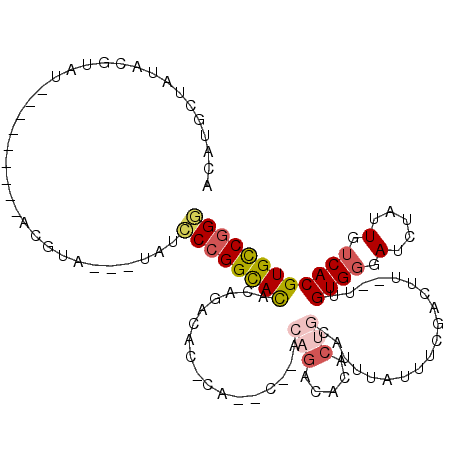
| Location | 3,066,348 – 3,066,443 |
|---|---|
| Length | 95 |
| Sequences | 5 |
| Columns | 115 |
| Reading direction | forward |
| Mean pairwise identity | 70.88 |
| Mean single sequence MFE | -25.67 |
| Consensus MFE | -13.50 |
| Energy contribution | -14.30 |
| Covariance contribution | 0.80 |
| Combinations/Pair | 1.25 |
| Mean z-score | -1.94 |
| Structure conservation index | 0.53 |
| SVM decision value | 1.27 |
| SVM RNA-class probability | 0.938354 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 3066348 95 + 20766785 ACAUGCUACACGUAU---------ACGUA---UAUCCCGGCACACAGACACACAGACA------CACACUGCAUUUAUUUUGACUU--UUGUGGGAUCUAUUGUCACGUGCCGGG .........(((...---------.))).---...((((((((...((((...(((..------((((..(((.......)).)..--.))))...)))..))))..)))))))) ( -25.80) >DroSec_CAF1 23131 92 + 1 ACAUGCCAU-CGUAU---------AUGUA---UAUCCCGGUACACAGACAC--------ACAGACACACUGCAUUUAUUUCGACUU--UUGUGGGAUCUAUUGUCACGUGCCGGG (((((....-....)---------)))).---...((((((((...((((.--------..(((....(..((.............--.))..)..)))..))))..)))))))) ( -22.54) >DroSim_CAF1 17804 101 + 1 ACAUGCCAUACGUAU---------ACGUA---UAUCCCGGUACACAGACACACAGACACACAGACACACUGCAUUUAUUUCGACUU--UUGUGGGAUCUAUUGUCACGUGCCGGG .......(((((...---------.))))---)..((((((((...((((...(((..((((((.....................)--)))))...)))..))))..)))))))) ( -26.40) >DroYak_CAF1 17431 102 + 1 ACAUACUAAACGCAUGUAUGUACUAUGUA---UAUCCCGGCACAAAAGCAC--------ACAGACACACUGCAUUUAUUUCAACUU--UUGUGGCAUCUAUUGUCACGUGCCGGA ((((((.........))))))........---....(((((((....(((.--------..........)))..............--..((((((.....))))))))))))). ( -28.80) >DroAna_CAF1 13835 100 + 1 --GUGUUAGAAG-AU---------AGCUACCCAAUGCCGGCAUAUGGAAACCCAUUCC---UGGGUUCCUGCACAUAUUUUGAUUUUUUUGUGCCAUCUAUUGUCACGUGUCCGU --(((.(((.((-((---------.((........)).((((((.((((.((((....---))))))))....((.....)).......)))))))))).))).)))........ ( -24.80) >consensus ACAUGCUAUACGUAU_________ACGUA___UAUCCCGGCACACAGACAC_CA__C__ACAGACACACUGCAUUUAUUUCGACUU__UUGUGGGAUCUAUUGUCACGUGCCGGG ...................................((((((((.................(((.....)))...................((((.(.....).)))))))))))) (-13.50 = -14.30 + 0.80)

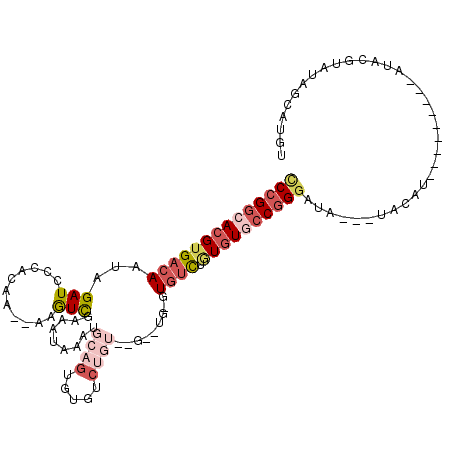
| Location | 3,066,348 – 3,066,443 |
|---|---|
| Length | 95 |
| Sequences | 5 |
| Columns | 115 |
| Reading direction | reverse |
| Mean pairwise identity | 70.88 |
| Mean single sequence MFE | -28.62 |
| Consensus MFE | -9.66 |
| Energy contribution | -12.14 |
| Covariance contribution | 2.48 |
| Combinations/Pair | 1.24 |
| Mean z-score | -1.82 |
| Structure conservation index | 0.34 |
| SVM decision value | 0.44 |
| SVM RNA-class probability | 0.735440 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 3066348 95 - 20766785 CCCGGCACGUGACAAUAGAUCCCACAA--AAGUCAAAAUAAAUGCAGUGUG------UGUCUGUGUGUCUGUGUGCCGGGAUA---UACGU---------AUACGUGUAGCAUGU ((((((((..((((((((((..((((.--..(.((.......)))..))))------.)))))).))))...)))))))).((---((((.---------...))))))...... ( -34.40) >DroSec_CAF1 23131 92 - 1 CCCGGCACGUGACAAUAGAUCCCACAA--AAGUCGAAAUAAAUGCAGUGUGUCUGU--------GUGUCUGUGUACCGGGAUA---UACAU---------AUACG-AUGGCAUGU ..((((..(((...........)))..--..))))......((((..(((((.(((--------((((((........)))))---)))).---------)))))-...)))).. ( -22.90) >DroSim_CAF1 17804 101 - 1 CCCGGCACGUGACAAUAGAUCCCACAA--AAGUCGAAAUAAAUGCAGUGUGUCUGUGUGUCUGUGUGUCUGUGUACCGGGAUA---UACGU---------AUACGUAUGGCAUGU (((((((((.((((((((((.(.((..--..)).)......((((((.....)))))))))))).))))))))..))))).((---((((.---------...))))))...... ( -29.40) >DroYak_CAF1 17431 102 - 1 UCCGGCACGUGACAAUAGAUGCCACAA--AAGUUGAAAUAAAUGCAGUGUGUCUGU--------GUGCUUUUGUGCCGGGAUA---UACAUAGUACAUACAUGCGUUUAGUAUGU (((((((((.(...((((((((.((..--..(((......)))...))))))))))--------...)...)))))))))...---............((((((.....)))))) ( -29.60) >DroAna_CAF1 13835 100 - 1 ACGGACACGUGACAAUAGAUGGCACAAAAAAAUCAAAAUAUGUGCAGGAACCCA---GGAAUGGGUUUCCAUAUGCCGGCAUUGGGUAGCU---------AU-CUUCUAACAC-- ........(((.....(((((((.................((((..((((((((---....))))))))))))((((.......)))))))---------))-)).....)))-- ( -26.80) >consensus CCCGGCACGUGACAAUAGAUCCCACAA__AAGUCGAAAUAAAUGCAGUGUGUCUGU__G__UG_GUGUCUGUGUGCCGGGAUA___UACAU_________AUACGUAUAGCAUGU ((((((((((((((...(((...........))).........((((.....)))).........)))).))))))))))................................... ( -9.66 = -12.14 + 2.48)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:42:35 2006