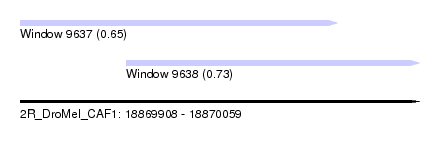
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 18,869,908 – 18,870,059 |
| Length | 151 |
| Max. P | 0.732610 |

| Location | 18,869,908 – 18,870,028 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 81.94 |
| Mean single sequence MFE | -49.83 |
| Consensus MFE | -31.52 |
| Energy contribution | -30.33 |
| Covariance contribution | -1.19 |
| Combinations/Pair | 1.46 |
| Mean z-score | -1.98 |
| Structure conservation index | 0.63 |
| SVM decision value | 0.25 |
| SVM RNA-class probability | 0.654273 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
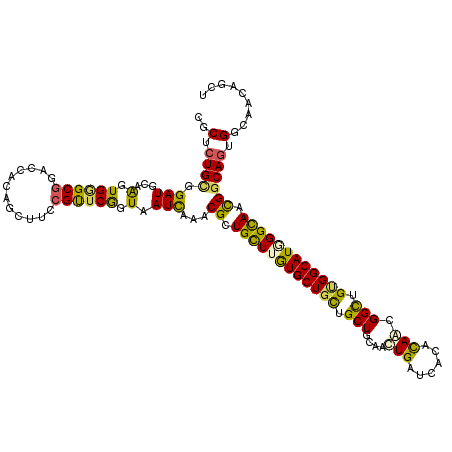
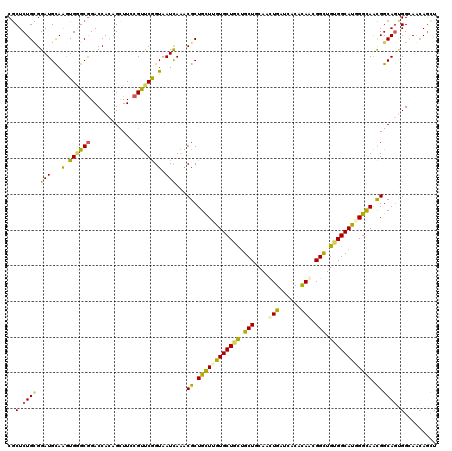
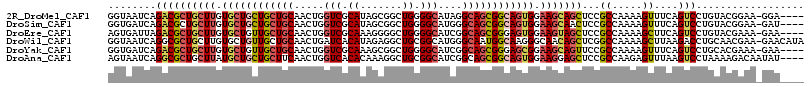
>2R_DroMel_CAF1 18869908 120 + 20766785 CGCUCUGGGGAUGCAAGUGGGCGGACCACAGUUUCCGCUCGGUAAUCAGACGCUGCUUGUGCUGCUGCUGCAACUGGUCGCAUAGCGGCUGGGGCAUAGGCAGCGGCAGUGGAAGCAGCU .(.(((((...(((....(((((((........))))))).))).))))))(((((((.((((((((((((..(..(((((...)))))..).......)))))))))))).))))))). ( -62.30) >DroVir_CAF1 3852 120 + 1 CGCUCUGCGGAUGUAAGUGAGCGGACCACAGUUUCCGUUCGGUGAUUAAACGCUGUUUGUGCUGCUGCUGCAAUUGAUCGCUCAACGGCUGUGGCAUGGGUAACGGCAGCGGCAGCAGUU .((.(.(((..((..(.((((((((........)))))))).)...))..))).)...))(((((((((((..((((....))))..(((((.((....)).))))).))))))))))). ( -45.60) >DroGri_CAF1 4355 120 + 1 CGCUCUGUGGAUGCAGAUGUGCGGACCAAAGCUUGCGUUCGGUAAUCAAACGCUGCUUGUGCUGCAGCUUCAGCUGUUCACUCAACGGUUGUGGCAUCGGCAGCGGCAGUGGCAACAGUU .(((((((.(.(((.(((.(((((....((((..(((((.(.....).))))).))))...)))))(((.((((((((.....)))))))).)))))).))).).)))).)))....... ( -45.80) >DroWil_CAF1 1952 120 + 1 CACUCUGUGGAUGCAGAUGGGCAGACCAAAGCUUACGCUCGGUAAUCAGGCGCUGCUUGUGCUGUUGCUGCAACUGAUCACAUAGAGGCUGCGGCAUGGGCAAUGGCAAGGGCAACAGCU ..((((((((((.(((.((((((((((..(((....))).))).....(((((.....))))).))))).)).)))))).)))))))((((..((....((....))....))..)))). ( -44.10) >DroMoj_CAF1 2264 120 + 1 CGCUCUGCGGAUGCAAGUGAGCGGACCACAGCUUGCGUUCAGUGAUCAAACGCUGCUUGUGCUGCUGCUUCAGCUGAUCGCUCAGUGGCUGUGGCAUGGGCAAUGGCAGUGGCAACAAUU .(((((((((((((((((............))))))))))..........((.((((..(((..(.(((...(((((....)))))))).)..)))..)))).)))))).)))....... ( -48.80) >DroAna_CAF1 2337 120 + 1 CGCUCUGCGGAUGCAAGUGGGCGGACCACAGUUUCCGUUCAGUAAUCAGGCGCUGCUUAUGCUGCUGCUUCAACUGGUCACACAAAGGCUGCGGCAUCGGCAGCGGCAGUGGAAGGAGCU .((((((((((.((..((((.....)))).)).)))))..........(.(((((((.(((((((.((((....((....))...)))).))))))).))))))).).......))))). ( -52.40) >consensus CGCUCUGCGGAUGCAAGUGGGCGGACCACAGCUUCCGUUCGGUAAUCAAACGCUGCUUGUGCUGCUGCUGCAACUGAUCACACAACGGCUGUGGCAUGGGCAACGGCAGUGGCAACAGCU ..(.((((.(((...(.((((((............)))))).).)))...((.((((((((((((.(((....(((......))).))).)))))))))))).)))))).)......... (-31.52 = -30.33 + -1.19)

| Location | 18,869,948 – 18,870,059 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 116 |
| Reading direction | forward |
| Mean pairwise identity | 83.08 |
| Mean single sequence MFE | -44.28 |
| Consensus MFE | -30.73 |
| Energy contribution | -31.65 |
| Covariance contribution | 0.92 |
| Combinations/Pair | 1.24 |
| Mean z-score | -2.32 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.43 |
| SVM RNA-class probability | 0.732610 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 18869948 111 + 20766785 GGUAAUCAGACGCUGCUUGUGCUGCUGCUGCAACUGGUCGCAUAGCGGCUGGGGCAUAGGCAGCGGCAGUGGAAGCAGCUCCGCCAAAAGUUUCAGUCCUGUACGGAA-GGA---- (((.....((.(((((((.((((((((((((..(..(((((...)))))..).......)))))))))))).))))))))).))).......((..(((.....))).-.))---- ( -51.60) >DroSim_CAF1 2378 111 + 1 GGUGAUCAGACGCUGCUUGUGCUGCUGCUGCAACUGGUCGCAUAGCGGCUGGGGCAUGGGCAGCGGCAGUGGAAGCAACUCCGCCAAAAGUUUCAGUCCUGUACGGAA-GAU---- ((((....((.(.(((((.((((((((((((..(..(((((...)))))..).......)))))))))))).))))).)))))))...........(((.....))).-...---- ( -45.70) >DroEre_CAF1 2330 111 + 1 AGUGAUUAGACGCUGCUUGUGCUGUUGCUGCAACUGGUCGCAAAGGGGCUGGGGCAUCGGCAGCGGGAGUGGAAGUAGCUCCGCCAAAAGCUUCAGUCCUGUACGAAA-GAA---- .((((((((..((.((..........)).))..))))))))...(((((((((((...(((....(((((.......))))))))....)))))))))))...(....-)..---- ( -42.90) >DroWil_CAF1 1992 115 + 1 GGUAAUCAGGCGCUGCUUGUGCUGUUGCUGCAACUGAUCACAUAGAGGCUGCGGCAUGGGCAAUGGCAAGGGCAACAGCUCGGCCAAAAGCUUAAGACCUGCAACGAA-GAACAUA (((....((((..((((..((((((.(((....(((......))).))).))))))..)))).((((..((((....)))).))))...))))...))).........-....... ( -38.70) >DroYak_CAF1 2382 111 + 1 GGUGAUCAGACGCUGCUUGUGCUGUUGCUGCAACUGGUCGCAAAGCGGCUGGGGCAUCGGCAGCGGGAGCGGAAGCAGUUCCGCCAAAAGUUUCAGUCCUGCACGAAA-GAA---- ((((....((.(((((((.((((.(((((((..(..(((((...)))))..).......))))))).)))).)))))))))))))..................(....-)..---- ( -45.00) >DroAna_CAF1 2377 112 + 1 AGUAAUCAGGCGCUGCUUAUGCUGCUGCUUCAACUGGUCACACAAAGGCUGCGGCAUCGGCAGCGGCAGUGGAAGGAGCUCCGCCAAGAGUUUAAGUCCUAAAAGACAAUAU---- .((.....(.(((((((.(((((((.((((....((....))...)))).))))))).))))))).)...(((..((((((......))))))...)))......)).....---- ( -41.80) >consensus GGUAAUCAGACGCUGCUUGUGCUGCUGCUGCAACUGGUCGCAUAGCGGCUGGGGCAUCGGCAGCGGCAGUGGAAGCAGCUCCGCCAAAAGUUUCAGUCCUGUACGAAA_GAA____ ........((((((((((.((((((((((((.....(((.((.......)).)))....)))))))))))).)))))))...((.....))....))).................. (-30.73 = -31.65 + 0.92)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:00:19 2006