
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 18,864,020 – 18,864,218 |
| Length | 198 |
| Max. P | 0.996468 |

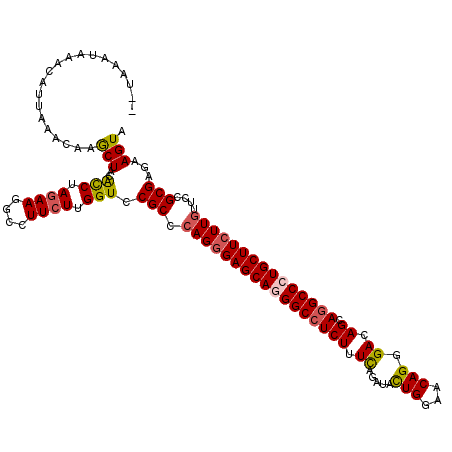
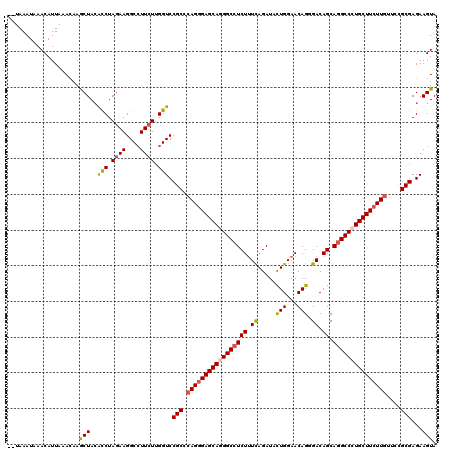
| Location | 18,864,020 – 18,864,138 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.58 |
| Mean single sequence MFE | -38.68 |
| Consensus MFE | -32.78 |
| Energy contribution | -33.10 |
| Covariance contribution | 0.32 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.38 |
| Structure conservation index | 0.85 |
| SVM decision value | 2.70 |
| SVM RNA-class probability | 0.996468 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 18864020 118 + 20766785 --UAAAUAAACAUUAAACAAGCUAGAUCUAGAAGGCCUUCUUGGUCCGCCCAGGGAGCAGGGCCUCUUUCAGAUACUGGAACAGCGACAGCAGGCCCUGCUUCUUGUUACGCGAGAAGUA --..................(((.((((.((((....)))).))))(((.(((((((((((((((.((((((...))))))..((....)))))))))))))))))....)))...))). ( -43.30) >DroSec_CAF1 39428 118 + 1 --UAAAUAAACAUUAAACAAGCUACACCUAGAAGGCCUUCUUGGUCCGCCCAGGGAGCAAGGCCUCUUUCAGAUACUGGAACAGCGACAGCAGGCCCUGCUUCUUGUUGCGCGAGAAGUA --..................(((..........)))(((((((...(((.(((((((((.(((((.((((((...))))))..((....))))))).)))))))))..)))))))))).. ( -37.70) >DroSim_CAF1 38893 118 + 1 --UAAAUUAACAUUAAACAAGCUACACCUAGAAGGCGUUGUGGGUCCGCCCAGAGAGCAAGGCAUCUAUUAGAUAUUGGAACAGGGACAGCAGGCCCUGCUUGUUUUUGUGCGAGAAGUA --.......(((..((((((((....(((.....((.((.((((....)))).)).)).)))..((((........))))..((((........)))))))))))).))).......... ( -28.60) >DroEre_CAF1 41604 120 + 1 AAUAAAUAAACAUUAAAUAAACUACGCCUAGAAGGCCUUCUUGGUCCGCCCAGGGAGCAGGGCCUCUUUCAGAUACUGGAACAGGGACAGCAGGCCCUGCUUCUUGUUCCGCGAGAAGUA .........................(((.....)))(((((((....(..(((((((((((((((((.((.....(((...))).)).)).)))))))))))))))..)..))))))).. ( -42.80) >DroYak_CAF1 40825 118 + 1 --UAAAUAAACAUUAAAUAAACUACACCUAGAAGGCCUUCUUGGUCCGCCCAGGGAGCAGGGCCUCUUUCAGAUACUGGAACAGGGACAGCAGGCCCUGCUUCUUGUUCCGCGAGAAGUA --..................(((.((((.((((....)))).))).(((.(((((((((((((((((.((.....(((...))).)).)).)))))))))))))))....))).).))). ( -41.00) >consensus __UAAAUAAACAUUAAACAAGCUACACCUAGAAGGCCUUCUUGGUCCGCCCAGGGAGCAGGGCCUCUUUCAGAUACUGGAACAGGGACAGCAGGCCCUGCUUCUUGUUCCGCGAGAAGUA ....................(((..(((.((((....)))).))).(((.(((((((((((((((((.((.....(((...))).)).)).)))))))))))))))....)))...))). (-32.78 = -33.10 + 0.32)

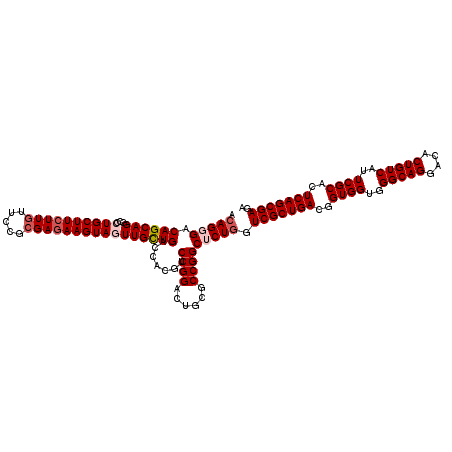
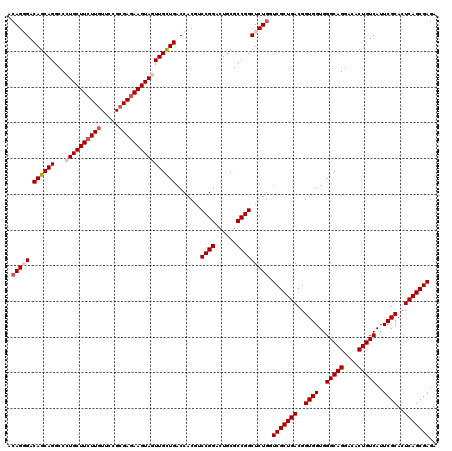
| Location | 18,864,098 – 18,864,218 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.00 |
| Mean single sequence MFE | -51.32 |
| Consensus MFE | -47.94 |
| Energy contribution | -49.18 |
| Covariance contribution | 1.24 |
| Combinations/Pair | 1.02 |
| Mean z-score | -1.76 |
| Structure conservation index | 0.93 |
| SVM decision value | 2.45 |
| SVM RNA-class probability | 0.994143 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 18864098 120 + 20766785 ACAGCGACAGCAGGCCCUGCUUCUUGUUACGCGAGAAGUAGUUGCUGACCACGUCCGGACUGCGCCGGCUCUGGUCGCUGACGGUGGUGGGCAGGACACUGUCAUUCGCACUCAGCGAGA .((.((.((((.(((.((((((((((.....))))))))))..)))(((((...((((......))))...))))))))).)).))...(((((....))))).(((((.....))))). ( -53.60) >DroSec_CAF1 39506 120 + 1 ACAGCGACAGCAGGCCCUGCUUCUUGUUGCGCGAGAAGUAGUUGCUGACCACGUCCGGACUGCGCCGGCUCUGGUCGCUGACGGUGGUGGGCAGGACACUGUCAUUCGCACUCAGCGAGA .((.((.((((.(((.((((((((((.....))))))))))..)))(((((...((((......))))...))))))))).)).))...(((((....))))).(((((.....))))). ( -53.60) >DroSim_CAF1 38971 119 + 1 ACAGGGACAGCAGGCCCUGCUUGUUUUUGUGCGAGAAGUAGUUGUUGACCACGUCCGGAGUGCGCCGGCUCU-GUCGCUGACGGUGGUGGGCAGGACACUGUCAUUCGCAAUCAGCGAGA .(((((.((((((...((.(((((......))))).))...)))))).......((((......))))))))-)(((((((..((((..(((((....)))))..))))..))))))).. ( -44.60) >DroEre_CAF1 41684 120 + 1 ACAGGGACAGCAGGCCCUGCUUCUUGUUCCGCGAGAAGUAAUUGCUGACCACAUCCGGACUGCGCCGGCUCUGAUCGCUGACGGUGGUGGGCAGGACACUGUCAUUCGCACUCAGCGAGA .(((((.((((((....(((((((((.....))))))))).)))))).......((((......))))))))).(((((((..((((..(((((....)))))..))))..))))))).. ( -52.40) >DroYak_CAF1 40903 120 + 1 ACAGGGACAGCAGGCCCUGCUUCUUGUUCCGCGAGAAGUAAUUGCUGACCACAUCCGGACUGCGCCGGCUCUGAUCGCUGACGGUGGUGGGCAGGACACUGUCAUUCGCACUCAGCGAGA .(((((.((((((....(((((((((.....))))))))).)))))).......((((......))))))))).(((((((..((((..(((((....)))))..))))..))))))).. ( -52.40) >consensus ACAGGGACAGCAGGCCCUGCUUCUUGUUCCGCGAGAAGUAGUUGCUGACCACGUCCGGACUGCGCCGGCUCUGGUCGCUGACGGUGGUGGGCAGGACACUGUCAUUCGCACUCAGCGAGA .(((((.((((((...((((((((((.....)))))))))))))))).......((((......))))))))).(((((((..((((..(((((....)))))..))))..))))))).. (-47.94 = -49.18 + 1.24)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:00:07 2006