
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
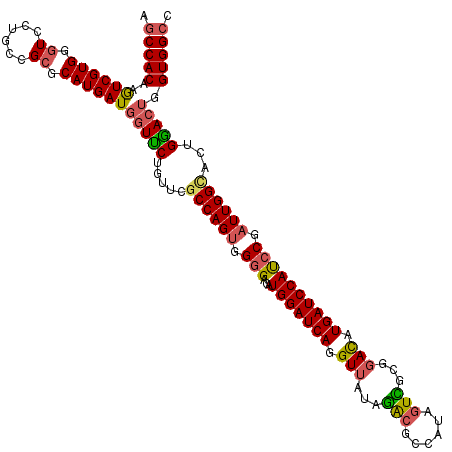
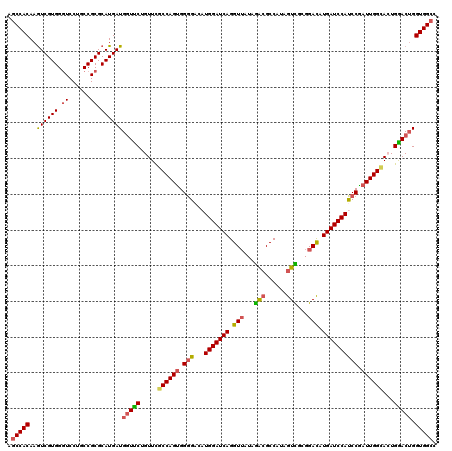
| Location | 18,692,165 – 18,692,325 |
| Length | 160 |
| Max. P | 0.999329 |

| Location | 18,692,165 – 18,692,285 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 89.50 |
| Mean single sequence MFE | -51.68 |
| Consensus MFE | -44.16 |
| Energy contribution | -44.24 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.23 |
| Mean z-score | -2.91 |
| Structure conservation index | 0.85 |
| SVM decision value | 3.52 |
| SVM RNA-class probability | 0.999329 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 18692165 120 + 20766785 AGCCACAAGUCGUGGAUCCUUCCGCGCAUGAUGGUUCUGUUCGCCAGUGGGGAUAUGGAUCAGGUUAUAGAGGCCAUAGUCUCGGACAUGAUCCAUCCGAUUGGUACUGGACUGGUGGCC .(((((..(.((((((....))))))).....(((((.((..((((((.((...((((((((.(((...(((((....))))).))).)))))))))).)))))))).))))).))))). ( -53.70) >DroSec_CAF1 55694 120 + 1 AGCCACAAGUCGUGGGUCCUGCCGCGCAUGAUGGUUCUGUUCGCCAGUGGGGACAUGGAUCAGGUUAUAGACGCCAUAGUCGCGGACAUGAUCCACCCGAUUGGCACUGAACUGGUGGCC .(((((..((((((.((......)).))))))(((((.((..((((((.(((...(((((((.(((...(((......)))...))).)))))))))).)))))))).))))).))))). ( -55.10) >DroSim_CAF1 56093 120 + 1 AGCCACAAGUCGUGGGUCCUGCCGCGCAUGAUGGUUCUGUUCGCCAGUGGGGACAUGGAUCAGGUUAUAGACGCCAUAGUCACGGACAUGAUCCACCCAAUUGGCACUGAACUGGUGGCC .(((((..((((((.((......)).))))))(((((.((..((((((.(((...(((((((.(((...(((......)))...))).)))))))))).)))))))).))))).))))). ( -51.90) >DroEre_CAF1 56581 120 + 1 ACCCACAAGUCGUGGGUCCUUCCGCGCAUGAUGAUCCUGUUUGCCAGCGGCGAUAUGGAUCAGGUUGUAAGCGCCAUAGUUGAGGAUAUGAUCCAUCCGAUUGGGAGUGGACAGGUGGCC ..((((..(((.((((((..(((((((.((.((((((...(((((...)))))...)))))).....)).))))((....)).)))...))))))(((.....)))...)))..)))).. ( -43.40) >DroYak_CAF1 56505 120 + 1 AGCCACAAAUCGUGGGUCCUGCCGCGCAUGAUGGUCCUGUUUGCCAGUGGGGACAUGGAUCAGGUGAUAGACGCCAUAGUUGCGGACAUGAUCCAUCCGAUUGGCAGUGGACUGGUGGCU ((((((..((((((.((......)).))))))(((((...((((((((.((...((((((((((((.....))))...(((...))).)))))))))).)))))))).))))).)))))) ( -54.30) >consensus AGCCACAAGUCGUGGGUCCUGCCGCGCAUGAUGGUUCUGUUCGCCAGUGGGGACAUGGAUCAGGUUAUAGACGCCAUAGUCGCGGACAUGAUCCAUCCGAUUGGCACUGGACUGGUGGCC .(((((..((((((.((......)).))))))(((((.....((((((.(((...(((((((.(((...(((......)))...))).)))))))))).))))))...))))).))))). (-44.16 = -44.24 + 0.08)

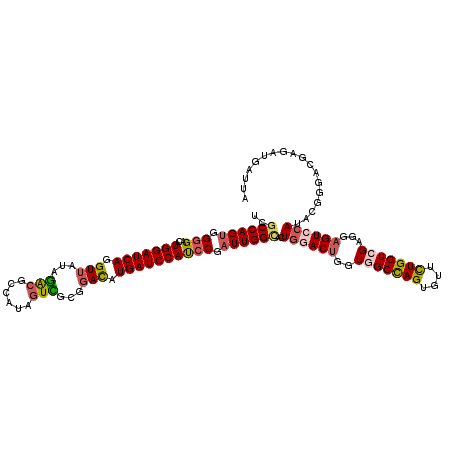
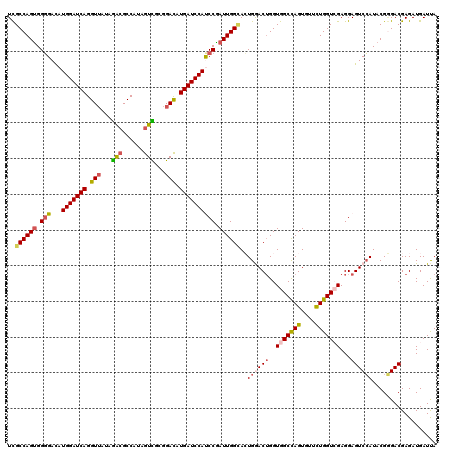
| Location | 18,692,205 – 18,692,325 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 89.42 |
| Mean single sequence MFE | -44.56 |
| Consensus MFE | -36.86 |
| Energy contribution | -37.66 |
| Covariance contribution | 0.80 |
| Combinations/Pair | 1.23 |
| Mean z-score | -2.34 |
| Structure conservation index | 0.83 |
| SVM decision value | 2.18 |
| SVM RNA-class probability | 0.989803 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 18692205 120 + 20766785 UCGCCAGUGGGGAUAUGGAUCAGGUUAUAGAGGCCAUAGUCUCGGACAUGAUCCAUCCGAUUGGUACUGGACUGGUGGCCAGUGUUCUGGUCGAGGAGUCCAUACGGGACGAGAUGAUUA ((((((((.((...((((((((.(((...(((((....))))).))).)))))))))).))))))..((((((..(((((((....)))))))...))))))........))........ ( -49.20) >DroSec_CAF1 55734 120 + 1 UCGCCAGUGGGGACAUGGAUCAGGUUAUAGACGCCAUAGUCGCGGACAUGAUCCACCCGAUUGGCACUGAACUGGUGGCCAGUGUUCUGGUCGAGGAGUCCAUACGGGACGAGAUGAUUA ..((((((.(((...(((((((.(((...(((......)))...))).)))))))))).))))))......((..(((((((....))))))).)).((((.....)))).......... ( -49.60) >DroSim_CAF1 56133 120 + 1 UCGCCAGUGGGGACAUGGAUCAGGUUAUAGACGCCAUAGUCACGGACAUGAUCCACCCAAUUGGCACUGAACUGGUGGCCAGUGUUCUGGUCGAGGAGUCCAUACGGGACGAGAUGAUCA ..((((((.(((...(((((((.(((...(((......)))...))).)))))))))).))))))...((.((..(((((((....))))))).)).((((.....)))).......)). ( -46.50) >DroEre_CAF1 56621 120 + 1 UUGCCAGCGGCGAUAUGGAUCAGGUUGUAAGCGCCAUAGUUGAGGAUAUGAUCCAUCCGAUUGGGAGUGGACAGGUGGCCAGUGUGUUGGUAGAGGAGUCGAUACGCGACGAGAUGAUUA (((((...)))))....(((((..((((..(.(((((...........((.(((((((.....))).)))))).)))))).(((((((((........))))))))).))))..))))). ( -34.80) >DroYak_CAF1 56545 120 + 1 UUGCCAGUGGGGACAUGGAUCAGGUGAUAGACGCCAUAGUUGCGGACAUGAUCCAUCCGAUUGGCAGUGGACUGGUGGCUAGUGUUCUGGUAGAGGAGUCCAUACGAGACGAGAUGAUUA .(((((((.((...((((((((((((.....))))...(((...))).)))))))))).)))))))(((((((....(((((....))))).....)))))))................. ( -42.70) >consensus UCGCCAGUGGGGACAUGGAUCAGGUUAUAGACGCCAUAGUCGCGGACAUGAUCCAUCCGAUUGGCACUGGACUGGUGGCCAGUGUUCUGGUCGAGGAGUCCAUACGGGACGAGAUGAUUA ..((((((.(((...(((((((.(((...(((......)))...))).)))))))))).))))))..((((((..(((((((....)))))))...)))))).................. (-36.86 = -37.66 + 0.80)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:58:17 2006