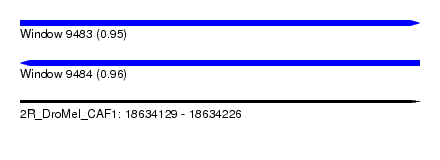
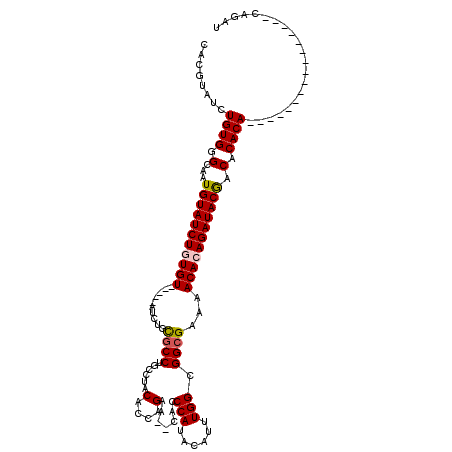
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 18,634,129 – 18,634,226 |
| Length | 97 |
| Max. P | 0.963222 |

| Location | 18,634,129 – 18,634,226 |
|---|---|
| Length | 97 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 84.74 |
| Mean single sequence MFE | -39.88 |
| Consensus MFE | -22.80 |
| Energy contribution | -22.68 |
| Covariance contribution | -0.12 |
| Combinations/Pair | 1.11 |
| Mean z-score | -3.48 |
| Structure conservation index | 0.57 |
| SVM decision value | 1.35 |
| SVM RNA-class probability | 0.945543 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 18634129 97 + 20766785 AUCUG----------------UGUGUGUCGUAUCUGUGUUUUCGCCGCCAAACGUAUGGGUG--UUCUAGUGUAGGCAGGCGCAGAU-----ACACAGAUACAUUGCCCACAGAUACGUG (((((----------------((.(((..((((((((((((((((((((..((...(((...--..)))..)).))).)))).))).-----))))))))))..))).)))))))..... ( -38.10) >DroSec_CAF1 44798 97 + 1 AUCUG----------------UGUGCGUCGUAUCUGUGUUUUCGCCGCCAAACGUAUGGGUG--UUCUGGUGUAGGCAGGCGCAGAU-----ACACAGAUACAUUGCCCACAGAUACGUG (((((----------------((.(((..((((((((((((((((((((.....(((.((..--..)).)))..))).)))).))).-----))))))))))..))).)))))))..... ( -41.30) >DroSim_CAF1 41214 97 + 1 AUCUG----------------UGUGCGUCGUAUCUGUGUUUUCGCCGCCAAGUGUAUGGGUG--UUCUGGUGUAGGCAGGUGCAGAU-----ACACAGAUACAUUGCCCACAGAUACGUG (((((----------------((.(((..((((((((((((((((((((...(((((.((..--..)).)))))))).)))).))).-----))))))))))..))).)))))))..... ( -39.80) >DroEre_CAF1 47107 104 + 1 AUUUG--------------UGUGUGUGUCGUAUCUGUGUUUUCGCCGCCAAAUGUAUGGGUG--CUCUGGUGUAUGAAGGCACCGAUACGAUACAUAGAUACAUUGCCCACAGCUACGUG ...((--------------(.(((((((((((((.(((((((((.(((((...(((....))--)..)))))..))))))))).))))))))))))).))).......(((......))) ( -39.00) >DroYak_CAF1 43715 115 + 1 AUCUGUGUACCAGAGUGUGUGUGUGUGUUGUAUCUGUGUUUUCGCCGCCAAAUGUAUGGGUGGGUUCUGGUGUAGGAAGGCACAGAU-----ACAAAGAUACAUUGCCCACAGAUACGUG ((((((((((....)))...((((((.((((((((((((((((..((((((((.((....)).))).)))))...))))))))))))-----))))..))))))....)))))))..... ( -41.20) >consensus AUCUG________________UGUGUGUCGUAUCUGUGUUUUCGCCGCCAAAUGUAUGGGUG__UUCUGGUGUAGGCAGGCGCAGAU_____ACACAGAUACAUUGCCCACAGAUACGUG .....................((((.((.((((((((((......((((.........))))...(((.((((......)))))))......))))))))))...)).))))........ (-22.80 = -22.68 + -0.12)

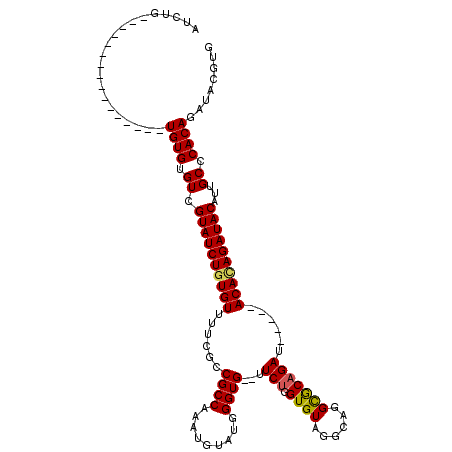
| Location | 18,634,129 – 18,634,226 |
|---|---|
| Length | 97 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 84.74 |
| Mean single sequence MFE | -32.83 |
| Consensus MFE | -19.20 |
| Energy contribution | -19.40 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.08 |
| Mean z-score | -3.58 |
| Structure conservation index | 0.58 |
| SVM decision value | 1.55 |
| SVM RNA-class probability | 0.963222 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 18634129 97 - 20766785 CACGUAUCUGUGGGCAAUGUAUCUGUGU-----AUCUGCGCCUGCCUACACUAGAA--CACCCAUACGUUUGGCGGCGAAAACACAGAUACGACACACA----------------CAGAU .....(((((((.....(((((((((((-----.....((((.(((.......(((--(........)))))))))))...))))))))))).....))----------------))))) ( -35.41) >DroSec_CAF1 44798 97 - 1 CACGUAUCUGUGGGCAAUGUAUCUGUGU-----AUCUGCGCCUGCCUACACCAGAA--CACCCAUACGUUUGGCGGCGAAAACACAGAUACGACGCACA----------------CAGAU .....(((((((.((..(((((((((((-----.....((((.(((.......(((--(........)))))))))))...)))))))))))..)).))----------------))))) ( -38.11) >DroSim_CAF1 41214 97 - 1 CACGUAUCUGUGGGCAAUGUAUCUGUGU-----AUCUGCACCUGCCUACACCAGAA--CACCCAUACACUUGGCGGCGAAAACACAGAUACGACGCACA----------------CAGAU .....(((((((.((..(((((((((((-----.(.(((..(((.......)))..--..((((......))).)))).).)))))))))))..)).))----------------))))) ( -32.70) >DroEre_CAF1 47107 104 - 1 CACGUAGCUGUGGGCAAUGUAUCUAUGUAUCGUAUCGGUGCCUUCAUACACCAGAG--CACCCAUACAUUUGGCGGCGAAAACACAGAUACGACACACACA--------------CAAAU .((((.((.....)).)))).....(((.(((((((((((........))))...(--(..(((......)))..)).........))))))).)))....--------------..... ( -28.10) >DroYak_CAF1 43715 115 - 1 CACGUAUCUGUGGGCAAUGUAUCUUUGU-----AUCUGUGCCUUCCUACACCAGAACCCACCCAUACAUUUGGCGGCGAAAACACAGAUACAACACACACACACACUCUGGUACACAGAU .....(((((((..(..(((....((((-----(((((((..(((...(.(((((.............))))).)..)))..)))))))))))...)))..((.....)))..))))))) ( -29.82) >consensus CACGUAUCUGUGGGCAAUGUAUCUGUGU_____AUCUGCGCCUGCCUACACCAGAA__CACCCAUACAUUUGGCGGCGAAAACACAGAUACGACACACA________________CAGAU ........((((.(...(((((((((((..........((((......(....).......(((......))).))))...))))))))))).).))))..................... (-19.20 = -19.40 + 0.20)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:57:50 2006