
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 18,491,325 – 18,491,485 |
| Length | 160 |
| Max. P | 0.993868 |

| Location | 18,491,325 – 18,491,445 |
|---|---|
| Length | 120 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.50 |
| Mean single sequence MFE | -59.90 |
| Consensus MFE | -53.81 |
| Energy contribution | -55.88 |
| Covariance contribution | 2.06 |
| Combinations/Pair | 1.09 |
| Mean z-score | -3.51 |
| Structure conservation index | 0.90 |
| SVM decision value | 2.43 |
| SVM RNA-class probability | 0.993868 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 18491325 120 - 20766785 GCACUGUCUCCGGCUGGGGAUCCACCAGUUGGUGGGGCAGCUGGUGGGACAGGUGCUUCGGCAGUCUGCCGGACUAUCUGCAGAUGGCAUGGGUCAGCGUCUACAAUCGGGAGCAGUGCG (((((((..(((..((..(((((((((((((......))))))))))(((..((((((((((.....)))))).((((....))))))))..)))...)))..))..)))..))))))). ( -62.80) >DroSec_CAF1 9932 120 - 1 GCACUGUUUCCGGCUGGGGAUCCACCAGUUGGUGGGGCAGCUGGUGGGACAGGUGCUUCGGCAGUCUGCCAGACCAUCUGCAGAUGGCAUGGGUCAGCGUCUACAAUCGGGAGCAGUGCG ((((((((((((..((..(((((((((((((......))))))))))(((((((((....))).))).(((..(((((....)))))..))))))...)))..))..)))))))))))). ( -67.60) >DroEre_CAF1 15395 120 - 1 GCUCUGUCUCCGGCUGGGGAUCCACCAGUUGGUGGGGCAGCUGGUGGGACAGGUGCUUCGGCAGUCUGCCAGACUACCUCCAGAUGACAUGGGUCAGCGUCUACAAUCGGAAGCAGUGCG ((.(((..((((..((..(((((((((((((......))))))))))(((((((((....))((((.....))))))))(((.......))))))...)))..))..))))..))).)). ( -56.50) >DroYak_CAF1 17208 120 - 1 GCUAUGUUUCCGGCUGGGGAUCCACCAGUUGGUGGGCCAGCUGGUGGGACAGGUGCUUCGGUAGUCUGCCCGACUAUCUUCAACUGGUAUGGGUCAGUGUCUACAAUAGGGAGCAGUGCG ((..((((((((((....(.((((((((((((....)))))))))))).)....)))...((((.(((((((((((........)))).)))).)))...))))....)))))))..)). ( -52.70) >consensus GCACUGUCUCCGGCUGGGGAUCCACCAGUUGGUGGGGCAGCUGGUGGGACAGGUGCUUCGGCAGUCUGCCAGACUAUCUGCAGAUGGCAUGGGUCAGCGUCUACAAUCGGGAGCAGUGCG ((((((((((((..((..(((((((((((((......))))))))))(((((((((....))).))).(((..(((((....)))))..))))))...)))..))..)))))))))))). (-53.81 = -55.88 + 2.06)

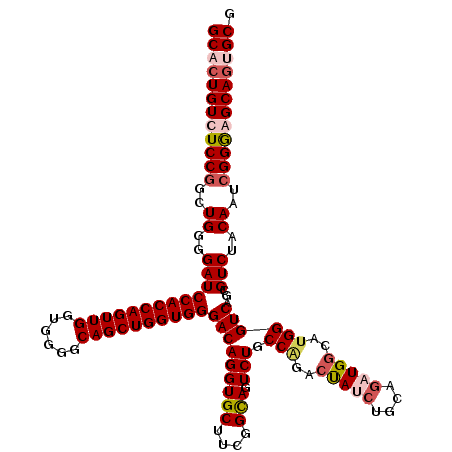
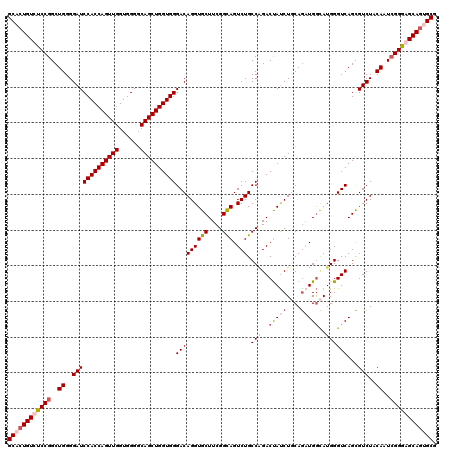
| Location | 18,491,365 – 18,491,485 |
|---|---|
| Length | 120 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 90.56 |
| Mean single sequence MFE | -44.06 |
| Consensus MFE | -36.29 |
| Energy contribution | -38.48 |
| Covariance contribution | 2.19 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.39 |
| Structure conservation index | 0.82 |
| SVM decision value | 1.40 |
| SVM RNA-class probability | 0.950026 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 18491365 120 + 20766785 CAGAUAGUCCGGCAGACUGCCGAAGCACCUGUCCCACCAGCUGCCCCACCAACUGGUGGAUCCCCAGCCGGAGACAGUGCACCACGAACCAUCGUCCGGUUCAUCCUCUGCUAUGCUGAU ((((((((.((((.....))))..((((.((((...((.((((..(((((....))))).....)))).)).)))))))).....(((((.......))))).......))))).))).. ( -44.60) >DroSec_CAF1 9972 120 + 1 CAGAUGGUCUGGCAGACUGCCGAAGCACCUGUCCCACCAGCUGCCCCACCAACUGGUGGAUCCCCAGCCGGAAACAGUGCACCAGGAACCAUCGUCCGGCUCAUCCUCUGCUAUGCUGAU .....(((.(((((((..(((...((((.(((....((.((((..(((((....))))).....)))).))..)))))))....(((.......))))))......))))))).)))... ( -40.60) >DroEre_CAF1 15435 120 + 1 GAGGUAGUCUGGCAGACUGCCGAAGCACCUGUCCCACCAGCUGCCCCACCAACUGGUGGAUCCCCAGCCGGAGACAGAGCACAUGGUAUCAUCAUCCGGCUCAUCGUCUGCUAUGCUGAU ....((((.((((((((.((((..((..(((((...((.((((..(((((....))))).....)))).)).))))).))..(((((...))))).)))).....)))))))).)))).. ( -45.20) >DroYak_CAF1 17248 120 + 1 AAGAUAGUCGGGCAGACUACCGAAGCACCUGUCCCACCAGCUGGCCCACCAACUGGUGGAUCCCCAGCCGGAAACAUAGCACCAGGAAUCAUCGUCCGGCACUUCGUCUGCUAGGCUGAU ....(((((.(((((((....((((........(((((((.(((....))).))))))).......((((((.......(.....)........)))))).))))))))))).))))).. ( -45.86) >consensus CAGAUAGUCUGGCAGACUGCCGAAGCACCUGUCCCACCAGCUGCCCCACCAACUGGUGGAUCCCCAGCCGGAAACAGAGCACCAGGAACCAUCGUCCGGCUCAUCCUCUGCUAUGCUGAU ....((((.((((((((.((((..((((.(((((((((((.((......)).))))))).(((......))))))))))).....(......)...)))).....)))))))).)))).. (-36.29 = -38.48 + 2.19)

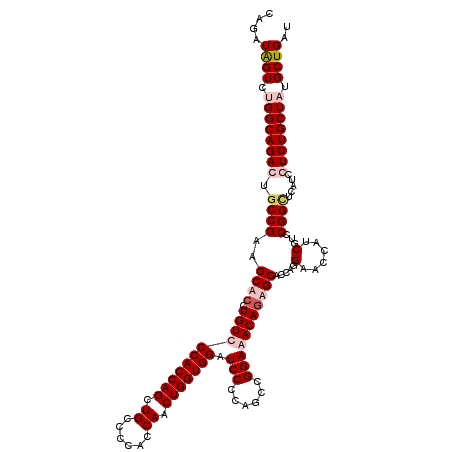
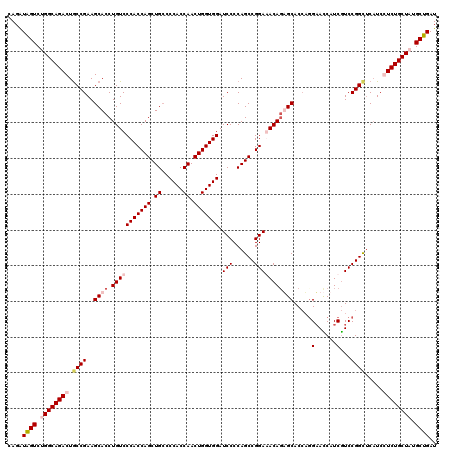
| Location | 18,491,365 – 18,491,485 |
|---|---|
| Length | 120 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.56 |
| Mean single sequence MFE | -53.00 |
| Consensus MFE | -42.42 |
| Energy contribution | -43.30 |
| Covariance contribution | 0.88 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.18 |
| Structure conservation index | 0.80 |
| SVM decision value | 0.99 |
| SVM RNA-class probability | 0.896798 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 18491365 120 - 20766785 AUCAGCAUAGCAGAGGAUGAACCGGACGAUGGUUCGUGGUGCACUGUCUCCGGCUGGGGAUCCACCAGUUGGUGGGGCAGCUGGUGGGACAGGUGCUUCGGCAGUCUGCCGGACUAUCUG ..(((.((((......((((((((.....)))))))).(..(.(((((((((((((....((((((....)))))).)))))))..)))))))..)((((((.....))))))))))))) ( -53.70) >DroSec_CAF1 9972 120 - 1 AUCAGCAUAGCAGAGGAUGAGCCGGACGAUGGUUCCUGGUGCACUGUUUCCGGCUGGGGAUCCACCAGUUGGUGGGGCAGCUGGUGGGACAGGUGCUUCGGCAGUCUGCCAGACCAUCUG ..........((((((..((((((.....))))))((((((.((((((.(((((((((....).)))))))).((((((.(((......))).)))))))))))).)))))).)).)))) ( -52.00) >DroEre_CAF1 15435 120 - 1 AUCAGCAUAGCAGACGAUGAGCCGGAUGAUGAUACCAUGUGCUCUGUCUCCGGCUGGGGAUCCACCAGUUGGUGGGGCAGCUGGUGGGACAGGUGCUUCGGCAGUCUGCCAGACUACCUC .........((((((.....((((((..(((....)))(..(.(((((((((((((....((((((....)))))).)))))))..)))))))..))))))).))))))........... ( -50.80) >DroYak_CAF1 17248 120 - 1 AUCAGCCUAGCAGACGAAGUGCCGGACGAUGAUUCCUGGUGCUAUGUUUCCGGCUGGGGAUCCACCAGUUGGUGGGCCAGCUGGUGGGACAGGUGCUUCGGUAGUCUGCCCGACUAUCUU ..(((((....(((((.((..((((..((....))))))..)).)))))..)))))(((.((((((((((((....)))))))))))).(((((((....))).)))))))......... ( -55.50) >consensus AUCAGCAUAGCAGACGAUGAGCCGGACGAUGAUUCCUGGUGCACUGUCUCCGGCUGGGGAUCCACCAGUUGGUGGGGCAGCUGGUGGGACAGGUGCUUCGGCAGUCUGCCAGACUAUCUG .........((((((.....((((((.((....))...(..(.(((((((((((((....((((((....)))))).)))))))..)))))))..))))))).))))))........... (-42.42 = -43.30 + 0.88)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:55:43 2006