
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 18,392,649 – 18,392,760 |
| Length | 111 |
| Max. P | 0.567735 |

| Location | 18,392,649 – 18,392,760 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.76 |
| Mean single sequence MFE | -39.46 |
| Consensus MFE | -26.32 |
| Energy contribution | -26.72 |
| Covariance contribution | 0.39 |
| Combinations/Pair | 1.41 |
| Mean z-score | -1.39 |
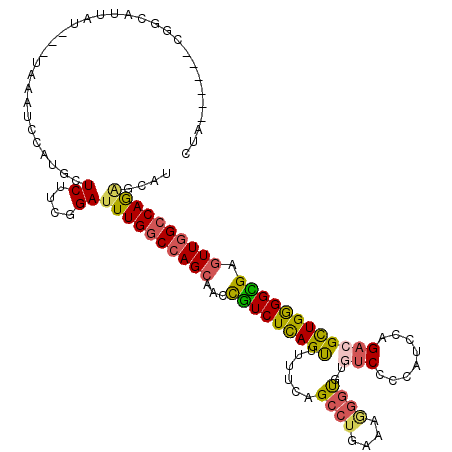
| Structure conservation index | 0.67 |
| SVM decision value | 0.07 |
| SVM RNA-class probability | 0.567735 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
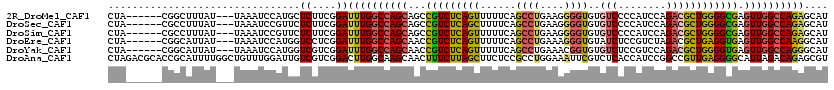
>2R_DroMel_CAF1 18392649 111 + 20766785 CUA------CGGCUUUAU---UAAAUCCAUGCUCUUCGGAUUUGGCCAGCAGCCGUCUCAGUUUUUCAGCCUGAAGGGGUGUGUCCCCAUCCAGACGCUGGGGCGAGUUGGCCAGAGCAU ...------..(((((..---.((((((.........))))))(((((((...(((((((((..(((.....)))((((.....))))........))))))))).)))))))))))).. ( -43.00) >DroSec_CAF1 29431 111 + 1 CUA------CGCCUUUAU---UAAAUCCGUUCUCUUCGGAUUUGGCCAGCAGCCGUCUCAGCUUUUCAGCCUGAAGGGGUGUGUCCCCAUCCAGACGCUGGGGCGAGUUGGCCAGAGCAU ...------.((((....---.(((((((.......)))))))(((((((...(((((((((..(((.....)))((((.....))))........))))))))).))))))))).)).. ( -45.70) >DroSim_CAF1 32179 111 + 1 CUA------CGCCUUUAU---UAAAUCCGUUCUCUUCGGAUUUGGCCAGCAGCCGUCUCAGUUUUUCAGCCUGAAGGGGUGUGUCCCCAUCCAGACGCUGGGGCGAGUUGGCCAGAGCAU ...------.((((....---.(((((((.......)))))))(((((((...(((((((((..(((.....)))((((.....))))........))))))))).))))))))).)).. ( -43.20) >DroEre_CAF1 30004 111 + 1 CUA------CGGCAUUAU---UAAAUCCAUGGUCCUCGGAUUUGGCCAGCAACCGUCUCAGUUUUUCAGCCUGAAAGGGUGUAUCUCCGUCUAGACGCUGAGGUGAGUUGGCCAAGGCAU ...------..((.....---.((((((.........))))))(((((((...(..((((((......((((....))))........((....))))))))..).)))))))...)).. ( -34.80) >DroYak_CAF1 30127 111 + 1 CUA------CGGCAUUAU---UAAAUCCAUGGUCGUCGGAUUUGGCCAGCAACCGUCUCAGUUUUUCAGCCUGAAACGGUGUGUCUCCGUCCAGACGCUGGGGUGAGUUGGCCAGGGCAU ...------(((((((((---.......))))).))))..((((((((((.(((((.((((.........)))).)))))((((((......))))))........)))))))))).... ( -37.60) >DroAna_CAF1 27713 120 + 1 CUAGACGCACCGCAUUUUGGCUGUUUGGAUUGUCGUCGGACUUGGCAAGCAACUUUCUUAGCUUCUCCGCCUGGAAAUUCGUCUCACCAUCCGGCCGUUGAGGGGCAUUAGACAGAGCGU ....((((.(((.....)))((((((((.((((((.......))))))............(((((((((.(((((..............))))).))..))))))).)))))))).)))) ( -32.44) >consensus CUA______CGGCAUUAU___UAAAUCCAUGCUCUUCGGAUUUGGCCAGCAACCGUCUCAGUUUUUCAGCCUGAAAGGGUGUGUCCCCAUCCAGACGCUGGGGCGAGUUGGCCAGAGCAU ......................................(.((((((((((...(((((((((......((((....))))..(((........)))))))))))).)))))))))).).. (-26.32 = -26.72 + 0.39)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:54:55 2006