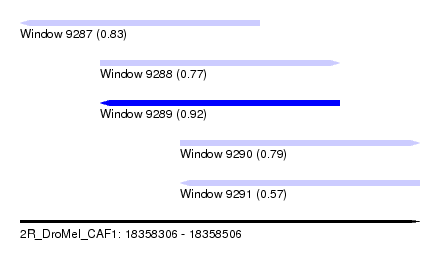
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 18,358,306 – 18,358,506 |
| Length | 200 |
| Max. P | 0.922080 |

| Location | 18,358,306 – 18,358,426 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 78.72 |
| Mean single sequence MFE | -38.77 |
| Consensus MFE | -21.83 |
| Energy contribution | -22.20 |
| Covariance contribution | 0.37 |
| Combinations/Pair | 1.37 |
| Mean z-score | -2.08 |
| Structure conservation index | 0.56 |
| SVM decision value | 0.73 |
| SVM RNA-class probability | 0.834986 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 18358306 120 - 20766785 AAUUGCAGUCGAAUUUUGACUGCAUACUGCAGCAGCAGCGCUUCCUUGAGUGCAUUUGUCGUUCGGAUGUGGGCGAUAUUUGUGAUCUUCUGGAUUUUUGCCUUCUGCAUAAAGUUUUCG ...(((((((((...)))))))))...(((((..((((....(((..(((.(((..(((((((((....)))))))))..)))...)))..)))...))))...)))))........... ( -38.70) >DroPse_CAF1 4322 120 - 1 AGUUGCAGUCGAACUUUGACUGCAUUCUGCAGCAACAGAGAUUUCUGGACUGCAUCUGCCGCUCAGAUGUAGGCGACAUUUGCGAUCUCCUUGACUUUUGCCUGCUGCACAAGGUCUUUG ...(((((((((...)))))))))...(((((((.(((((.((...((((((((((((.....)))))))))((((...))))....)))..)).)))))..)))))))........... ( -41.40) >DroGri_CAF1 3259 120 - 1 AGCUGCAAUCCAAUUUCGAUUGCAUUAUGCAGCAACAGAAAUUUCUCGAGUGCAUUUGUCGCUCGGAUGUGGGCGACAUUUGUGAUCUGCUGGACUUUUGCCUGCUGCAUAGGAUCUUUG ...(((((((.......)))))))((((((((((...((((.(((..((..(((..(((((((((....)))))))))..)))..))....))).))))...))))))))))........ ( -42.40) >DroWil_CAF1 2703 120 - 1 AAUUGGAAAAGAAUUUCGAUUGCAUUCUGCAACAAGAAAGAUUUCUUAAUUGCAUUUGUCGCUCAGAUGUGGGUGAUAUUUGUGAUCUUUUGGAUUUUUGCCUCUUGGACAAGGUCUUUG ......((((((.((((..((((.....))))...))))..........(..((..(((((((((....)))))))))..))..)))))))(((((((..((....))..)))))))... ( -29.80) >DroMoj_CAF1 3457 120 - 1 AGCUGCAGACGAACUUCGACUGCGUUCUGCAGCAGCAGAAGUUUCUGGAGUGCAUCUGCCGCUCGGAUGUGGGCGACAUUUGCGAUCUGCUGGACUUUUGUCUGCUGCAUCGCAUCUUUG .((.((((.((.....)).))))....(((((((((((((((((..(((.((((..((.((((((....)))))).))..)))).)))...))))))))).))))))))..))....... ( -49.50) >DroAna_CAF1 3716 120 - 1 AACUCCAGAGCAACUUCGACUGCAUCCUGCAACAGCAACGCUUCCUCGAUUGCAUCUGCCGCUCGGAUGUGGGCGAUAUUUGUGAUCUGUUGGACUUCUGUUUGCUCCACAAGGUCUUCG .......((((((..(((.(..(((((.(((...((((((......)).))))...))).....)))))..).)))...))))(((((..(((((........).))))..))))).)). ( -30.80) >consensus AACUGCAGACGAACUUCGACUGCAUUCUGCAGCAACAGAGAUUUCUCGAGUGCAUCUGCCGCUCGGAUGUGGGCGACAUUUGUGAUCUGCUGGACUUUUGCCUGCUGCACAAGGUCUUUG ...(((((.((.....)).)))))...(((((((...((((.(((..((.((((..((.((((((....)))))).))..)))).))....))).))))...)))))))........... (-21.83 = -22.20 + 0.37)


| Location | 18,358,346 – 18,358,466 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.44 |
| Mean single sequence MFE | -43.21 |
| Consensus MFE | -20.23 |
| Energy contribution | -20.68 |
| Covariance contribution | 0.45 |
| Combinations/Pair | 1.32 |
| Mean z-score | -2.70 |
| Structure conservation index | 0.47 |
| SVM decision value | 0.53 |
| SVM RNA-class probability | 0.771293 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
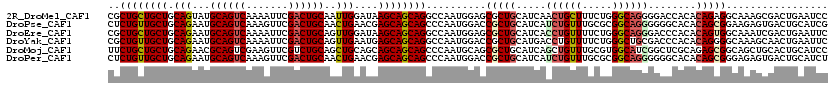
>2R_DroMel_CAF1 18358346 120 + 20766785 AAUAUCGCCCACAUCCGAACGACAAAUGCACUCAAGGAAGCGCUGCUGCUGCAGUAUGCAGUCAAAAUUCGACUGCAAUUGGAUAAGCAGCAGGCCAAUGGAGCGCUGCAUCAACUGCUU .....((((((..(((...................))).((.((((((((.((((.(((((((.......)))))))))))....))))))))))...))).)))..(((.....))).. ( -39.01) >DroVir_CAF1 2852 120 + 1 AAUGUCGCCCACAUCCGAGCGGCAGAUGCAGUCGAGAAACUUUUGCUGCUGCAUAAUGCAGUCGAAGUUGGCUUCGAGCUGAAUGAGCAGCAGUCCAAUGCAGCGCUGCAGCAGCUGUUU ..((((((.(......).))))))............((((....(((((((((...((((.((((((....))))))((((......)))).......))))....))))))))).)))) ( -44.30) >DroPse_CAF1 4362 120 + 1 AAUGUCGCCUACAUCUGAGCGGCAGAUGCAGUCCAGAAAUCUCUGUUGCUGCAGAAUGCAGUCAAAGUUCGACUGCAACUGAACGAGCAGCAGCCCAAUGGACCGCUGCAUCAUCUGUUU ..(((((((.......).))))))(((((((((((.......((((((((.(((..(((((((.......))))))).)))....)))))))).....)))...))))))))........ ( -47.00) >DroGri_CAF1 3299 120 + 1 AAUGUCGCCCACAUCCGAGCGACAAAUGCACUCGAGAAAUUUCUGUUGCUGCAUAAUGCAAUCGAAAUUGGAUUGCAGCUGCAUCAGGAGUAGUCCAAUGCAGCGUUGCAUCAAAUGCUU ..((((((.(......).))))))..((((.((....((((((.(((((........))))).)))))).)).))))(((((((..(((....))).)))))))...((((...)))).. ( -39.30) >DroMoj_CAF1 3497 120 + 1 AAUGUCGCCCACAUCCGAGCGGCAGAUGCACUCCAGAAACUUCUGCUGCUGCAGAACGCAGUCGAAGUUCGUCUGCAGCUGCAGCAGCAGCAGCCCAAUGCAGCGCUGCAUCAGCUGUUU ..((((((.(......).))))))((((((((.((....((.((((((((((((...((((.((.....)).))))..)))))))))))).)).....)).))...))))))........ ( -52.20) >DroAna_CAF1 3756 120 + 1 AAUAUCGCCCACAUCCGAGCGGCAGAUGCAAUCGAGGAAGCGUUGCUGUUGCAGGAUGCAGUCGAAGUUGCUCUGGAGUUGAAUAAGCAGCAGUGCUAUGGAGCGCUGAAUGACGUGCUU ......((.(.(((((..(((((((((((..........))))..))))))).)))))..((((..((((((.............))))))((((((....))))))...))))).)).. ( -37.42) >consensus AAUGUCGCCCACAUCCGAGCGGCAGAUGCACUCGAGAAACCUCUGCUGCUGCAGAAUGCAGUCGAAGUUCGACUGCAGCUGAAUAAGCAGCAGUCCAAUGCAGCGCUGCAUCAACUGCUU ..((((((.(......).)))))).(((((............((((((((.(((...((((.(.......).))))..)))....)))))))).............)))))......... (-20.23 = -20.68 + 0.45)


| Location | 18,358,346 – 18,358,466 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.44 |
| Mean single sequence MFE | -46.87 |
| Consensus MFE | -19.72 |
| Energy contribution | -21.31 |
| Covariance contribution | 1.59 |
| Combinations/Pair | 1.38 |
| Mean z-score | -3.46 |
| Structure conservation index | 0.42 |
| SVM decision value | 1.15 |
| SVM RNA-class probability | 0.922080 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
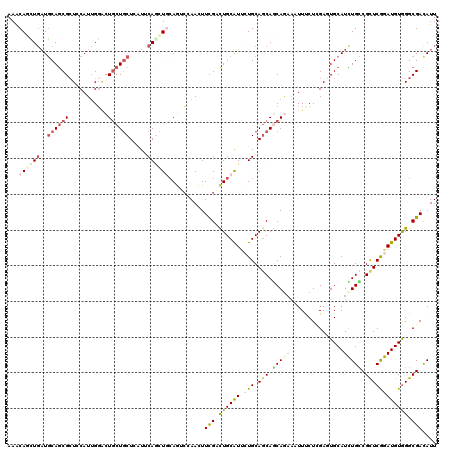
>2R_DroMel_CAF1 18358346 120 - 20766785 AAGCAGUUGAUGCAGCGCUCCAUUGGCCUGCUGCUUAUCCAAUUGCAGUCGAAUUUUGACUGCAUACUGCAGCAGCAGCGCUUCCUUGAGUGCAUUUGUCGUUCGGAUGUGGGCGAUAUU ..(((.....))).((((((....(((((((((((........(((((((((...)))))))))......)))))))).))).....))))))...(((((((((....))))))))).. ( -50.34) >DroVir_CAF1 2852 120 - 1 AAACAGCUGCUGCAGCGCUGCAUUGGACUGCUGCUCAUUCAGCUCGAAGCCAACUUCGACUGCAUUAUGCAGCAGCAAAAGUUUCUCGACUGCAUCUGCCGCUCGGAUGUGGGCGACAUU .....((((((((((.((.(((......))).)).)...(((.((((((....))))))))).....))))))))).........(((.(..((((((.....))))))..).))).... ( -45.60) >DroPse_CAF1 4362 120 - 1 AAACAGAUGAUGCAGCGGUCCAUUGGGCUGCUGCUCGUUCAGUUGCAGUCGAACUUUGACUGCAUUCUGCAGCAACAGAGAUUUCUGGACUGCAUCUGCCGCUCAGAUGUAGGCGACAUU .....(((((.((((((((((...)))))))))))))))(((.(((((((((...)))))))))..)))..((..(((......)))..(((((((((.....)))))))))))...... ( -51.50) >DroGri_CAF1 3299 120 - 1 AAGCAUUUGAUGCAACGCUGCAUUGGACUACUCCUGAUGCAGCUGCAAUCCAAUUUCGAUUGCAUUAUGCAGCAACAGAAAUUUCUCGAGUGCAUUUGUCGCUCGGAUGUGGGCGACAUU ..((((((((((((..(((((((((((....))).))))))))))))....((((((..((((........))))..))))))..))))))))...(((((((((....))))))))).. ( -48.50) >DroMoj_CAF1 3497 120 - 1 AAACAGCUGAUGCAGCGCUGCAUUGGGCUGCUGCUGCUGCAGCUGCAGACGAACUUCGACUGCGUUCUGCAGCAGCAGAAGUUUCUGGAGUGCAUCUGCCGCUCGGAUGUGGGCGACAUU .....((.(((((...(((.((..(((((.((((((((((((.(((((.((.....)).)))))..)))))))))))).))))).)).)))))))).))((((((....))))))..... ( -55.80) >DroAna_CAF1 3756 120 - 1 AAGCACGUCAUUCAGCGCUCCAUAGCACUGCUGCUUAUUCAACUCCAGAGCAACUUCGACUGCAUCCUGCAACAGCAACGCUUCCUCGAUUGCAUCUGCCGCUCGGAUGUGGGCGAUAUU ..(((.(((...(((.(((....))).))).(((((...........))))).....))))))....(((....)))........(((.(..((((((.....))))))..).))).... ( -29.50) >consensus AAACAGCUGAUGCAGCGCUCCAUUGGACUGCUGCUCAUUCAGCUGCAGUCCAACUUCGACUGCAUUCUGCAGCAGCAGAAAUUUCUCGAGUGCAUCUGCCGCUCGGAUGUGGGCGACAUU ...((((((..((((((...........)))))).....))))))..........(((.((((((.(((.(((.(((((...............))))).)))))))))))).))).... (-19.72 = -21.31 + 1.59)

| Location | 18,358,386 – 18,358,506 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 81.94 |
| Mean single sequence MFE | -47.93 |
| Consensus MFE | -29.66 |
| Energy contribution | -30.05 |
| Covariance contribution | 0.39 |
| Combinations/Pair | 1.21 |
| Mean z-score | -2.49 |
| Structure conservation index | 0.62 |
| SVM decision value | 0.59 |
| SVM RNA-class probability | 0.791655 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
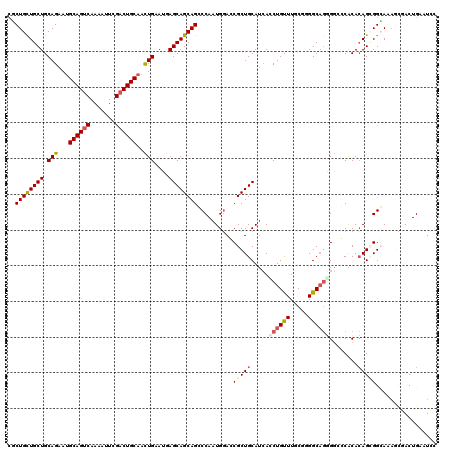
>2R_DroMel_CAF1 18358386 120 + 20766785 CGCUGCUGCUGCAGUAUGCAGUCAAAAUUCGACUGCAAUUGGAUAAGCAGCAGGCCAAUGGAGCGCUGCAUCAACUGCUUUCUGGGCAGGGGACCACACAGAGGCAAAGCGACUGAAUCC ((((((((((.((((.(((((((.......)))))))))))....))))))..(((..((..((...)).((..(((((.....)))))..)).....))..)))..))))......... ( -43.40) >DroPse_CAF1 4402 120 + 1 CUCUGUUGCUGCAGAAUGCAGUCAAAGUUCGACUGCAACUGAACGAGCAGCAGCCCAAUGGACCGCUGCAUCAUCUGUUUGCGCGGCAGGGGGGCACACAGCGGAAGAGUGACUGCAUCG (((((((((.(((((.(((((((.......))))))).......(((((((.(((....)).).))))).)).....))))))))))))))..((((((..(....).)))..))).... ( -46.40) >DroEre_CAF1 8455 120 + 1 CGCUGCUGCUGCAGAAUGCAGUCAAAAUUCGACUGCAGUUGGAUAAGCAGCAGGCCAAUGGAGCGCUGCAUCACCUGUUUUCUGGGCAGGGACCCACACAGUGGCAAAUCGACUGAAUUC ..((((((((.(....(((((((.......)))))))....)...))))))))((((.(((..(.((((....((........)))))).)..))).....))))............... ( -43.90) >DroYak_CAF1 8591 120 + 1 CGCUGUUGCUGCAGAAUGCAGUCAAAAUUCGACUGCAGUUGAAUGAGCAGCAGGCCAAUGGACCGCUGCAUGACCUGUUUUCUGGGCUGCGACCCACACAGGGGCAAAGCAACUGAAUUC ....((((((.(((((.(((((((..(((((((....)))))))..(((((.((((...)).))))))).))).)))).)))))...(((..(((.....)))))).))))))....... ( -44.20) >DroMoj_CAF1 3537 120 + 1 UUCUGCUGCUGCAGAACGCAGUCGAAGUUCGUCUGCAGCUGCAGCAGCAGCAGCCCAAUGCAGCGCUGCAUCAGCUGUUUGCGUGGCAUCGGCUCGCAGAGCGGCAGCUGCACUGCAUCC ...(((((((((((...((((.((.....)).))))..)))))))))))((((.....(((((((((((.((.((.....))(((((....)).))).))))))).)))))))))).... ( -62.60) >DroPer_CAF1 4419 120 + 1 CUCUGUUGCUGCAGAAUGCAGUCAAAGUUCGACUGCAACUGAACGAGCAGCAGCCCAAUGGACCGCUGCAUCAUCUGUUUGCGCGGCAGGGGGGCACACAGCGGGAGAGUGACUGCAUCU .(((((....)))))(((((((((...(((..((((..(((......)))..((((....(.((((.(((.........))))))))....)))).....))))..)))))))))))).. ( -47.10) >consensus CGCUGCUGCUGCAGAAUGCAGUCAAAAUUCGACUGCAACUGAAUGAGCAGCAGCCCAAUGGACCGCUGCAUCACCUGUUUGCGGGGCAGGGGCCCACACAGCGGCAAAGCGACUGAAUCC ..((((((((.(((...((((((.......))))))..)))....))))))))..........(((((.....((((((.....))))))........)))))................. (-29.66 = -30.05 + 0.39)
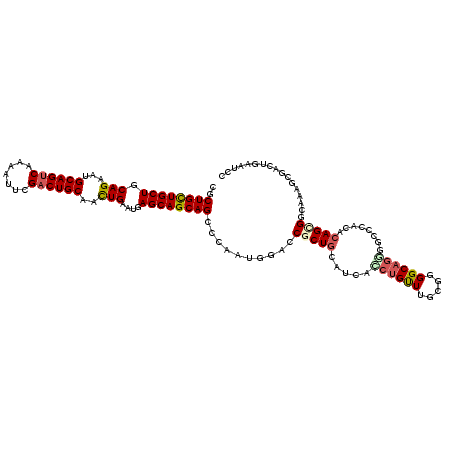
| Location | 18,358,386 – 18,358,506 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.94 |
| Mean single sequence MFE | -47.45 |
| Consensus MFE | -24.76 |
| Energy contribution | -24.82 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.22 |
| Mean z-score | -2.46 |
| Structure conservation index | 0.52 |
| SVM decision value | 0.06 |
| SVM RNA-class probability | 0.565805 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 18358386 120 - 20766785 GGAUUCAGUCGCUUUGCCUCUGUGUGGUCCCCUGCCCAGAAAGCAGUUGAUGCAGCGCUCCAUUGGCCUGCUGCUUAUCCAAUUGCAGUCGAAUUUUGACUGCAUACUGCAGCAGCAGCG ((((((((..((((....((((.(..(....)..).))))))))..)))).((((((..((...))..))))))..))))...(((((((((...)))))))))..((((....)))).. ( -41.90) >DroPse_CAF1 4402 120 - 1 CGAUGCAGUCACUCUUCCGCUGUGUGCCCCCCUGCCGCGCAAACAGAUGAUGCAGCGGUCCAUUGGGCUGCUGCUCGUUCAGUUGCAGUCGAACUUUGACUGCAUUCUGCAGCAACAGAG ..(((((((((...(((.((((((.((......)))))((((...(((((.((((((((((...)))))))))))))))...))))))).)))...)))))))))((((......)))). ( -50.50) >DroEre_CAF1 8455 120 - 1 GAAUUCAGUCGAUUUGCCACUGUGUGGGUCCCUGCCCAGAAAACAGGUGAUGCAGCGCUCCAUUGGCCUGCUGCUUAUCCAACUGCAGUCGAAUUUUGACUGCAUUCUGCAGCAGCAGCG ...............((((..(((((.(((((((.........)))).))).)).))).....))))((((((((........(((((((((...)))))))))......)))))))).. ( -40.34) >DroYak_CAF1 8591 120 - 1 GAAUUCAGUUGCUUUGCCCCUGUGUGGGUCGCAGCCCAGAAAACAGGUCAUGCAGCGGUCCAUUGGCCUGCUGCUCAUUCAACUGCAGUCGAAUUUUGACUGCAUUCUGCAGCAACAGCG .......((((((..((.(((((.(((((....)))))....)))))....(((((((((....)))).))))).........(((((((((...)))))))))....)))))))).... ( -45.60) >DroMoj_CAF1 3537 120 - 1 GGAUGCAGUGCAGCUGCCGCUCUGCGAGCCGAUGCCACGCAAACAGCUGAUGCAGCGCUGCAUUGGGCUGCUGCUGCUGCAGCUGCAGACGAACUUCGACUGCGUUCUGCAGCAGCAGAA .((((((((((.((....(((.((((.((....))..))))...)))....)).))))))))))......((((((((((((.(((((.((.....)).)))))..)))))))))))).. ( -58.90) >DroPer_CAF1 4419 120 - 1 AGAUGCAGUCACUCUCCCGCUGUGUGCCCCCCUGCCGCGCAAACAGAUGAUGCAGCGGUCCAUUGGGCUGCUGCUCGUUCAGUUGCAGUCGAACUUUGACUGCAUUCUGCAGCAACAGAG ..(((((((((.......((((((.((......)))))((((...(((((.((((((((((...)))))))))))))))...))))))).......)))))))))((((......)))). ( -47.44) >consensus GGAUGCAGUCGCUCUGCCGCUGUGUGGGCCCCUGCCCAGAAAACAGAUGAUGCAGCGCUCCAUUGGCCUGCUGCUCAUUCAACUGCAGUCGAACUUUGACUGCAUUCUGCAGCAACAGAG ...................((((........((((..........((....((((((..((...))..))))))....))...(((((((((...)))))))))....))))..)))).. (-24.76 = -24.82 + 0.06)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:54:44 2006