
| Sequence ID | 2R_DroMel_CAF1 |
|---|---|
| Location | 18,232,833 – 18,233,033 |
| Length | 200 |
| Max. P | 0.999971 |

| Location | 18,232,833 – 18,232,953 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.78 |
| Mean single sequence MFE | -49.20 |
| Consensus MFE | -40.56 |
| Energy contribution | -41.45 |
| Covariance contribution | 0.89 |
| Combinations/Pair | 1.17 |
| Mean z-score | -2.48 |
| Structure conservation index | 0.82 |
| SVM decision value | 0.88 |
| SVM RNA-class probability | 0.873510 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 18232833 120 + 20766785 CGCCAGCGAUCUUCGUGCCGUUAAUGGACGCAUCGAUCACGUGGGUGCCCACCUCGGUGGGCACGAACUCGAUGCGAGCAGCACCAUUUGCCGCCUCGUGGGUGACUCUGGCGUUCAGCU ((((((.(......((((.(((......((((((((....((..((((((((....))))))))..))))))))))))).)))).......((((.....)))).).))))))....... ( -51.80) >DroVir_CAF1 59409 120 + 1 CGCCAGCGAUCUUCGUGCCAUUGAUGGACGCAUCAAUGAUGUGCGUGCCCACUUCGGUGGGCACGAACUCAAUGCGUGCAGCACCAUUUGCCGCCUCAUGGGUGACUCUGGCAUUCAGCU .(((((.(.((.(((..(((((((((....))))))))..)..)((((((((....))))))))))(((((..(((.((((......)))))))....)))))))).)))))........ ( -53.00) >DroPse_CAF1 30317 120 + 1 CACCAGCUAUCUUCGUGCCAUUGAUGGACGCAUCGAUGACGUGCGUUCCCACUUCCGUGGGGACGAACUCGAUGCGUGCAGCACCAUUUGCCGCCUCAUGGGUGACUCUGGCGUUCAGCU ....((((......((((......((.(((((((((....((.(((((((((....))))))))).))))))))))).)))))).......((((....((....))..))))...)))) ( -48.70) >DroGri_CAF1 69371 120 + 1 CGCCAGCGAUCUUUGUGCCAUUGAUGGACGCAUCAAUGAUGUGCGUGCCCACUUCGGUGGGCACGAACUCAAUGCGUGCAGCACCAUUUGCCGCCUCAUGGGUGACUCUGGCAUUCAGCU .(((((.(.....((..(((((((....((((((...))))))(((((((((....)))))))))...)))))).)..))(((.....)))((((.....)))).).)))))........ ( -52.80) >DroWil_CAF1 38913 120 + 1 CGCCAGCUAUCUUUGUGCCAUUGAUGGACGCAUCGAUAAUGUGGGUACCCACCUCGGUGGGGACAAACUCAAUGCGUGCAGCACCAUUUGCCGCCUCAUGGGUGACUCUGGCAUUCAGUU .(((((.......((..(((((((((..(.((((((....((((....)))).)))))).)..))...)))))).)..))(((.....)))((((.....))))...)))))........ ( -42.80) >DroPer_CAF1 30250 120 + 1 CACCAGCUAUCUUCGUACCAUUGAUGGACGCAUCGAUGACGUGCGUUCCCACUUCCGUGGGGACGAACUCGAUGCGUGCAGCACCAUUUGCCGCCUCAUGGGUGACUCUGGCGUUCAGCU ....((((................((.(((((((((....((.(((((((((....))))))))).))))))))))).))(((.....)))((((....((....))..))))...)))) ( -46.10) >consensus CGCCAGCGAUCUUCGUGCCAUUGAUGGACGCAUCGAUGACGUGCGUGCCCACUUCGGUGGGCACGAACUCAAUGCGUGCAGCACCAUUUGCCGCCUCAUGGGUGACUCUGGCAUUCAGCU .(((((...((.((((((((((((((....))))))))..))))((((((((....))))))))))(((((..(((.((((......)))))))....)))))))..)))))........ (-40.56 = -41.45 + 0.89)


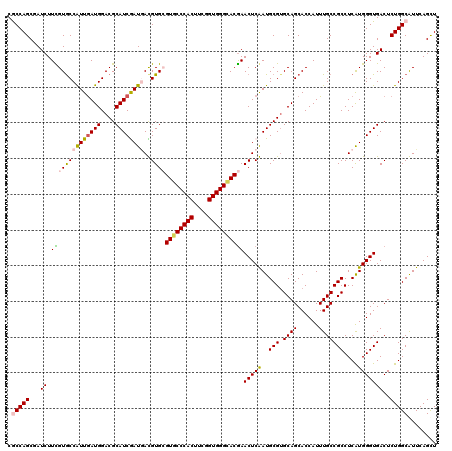
| Location | 18,232,833 – 18,232,953 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.78 |
| Mean single sequence MFE | -49.57 |
| Consensus MFE | -44.23 |
| Energy contribution | -43.98 |
| Covariance contribution | -0.25 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.62 |
| Structure conservation index | 0.89 |
| SVM decision value | 2.66 |
| SVM RNA-class probability | 0.996192 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 18232833 120 - 20766785 AGCUGAACGCCAGAGUCACCCACGAGGCGGCAAAUGGUGCUGCUCGCAUCGAGUUCGUGCCCACCGAGGUGGGCACCCACGUGAUCGAUGCGUCCAUUAACGGCACGAAGAUCGCUGGCG .......((((((.(((......(((.(((((.....))))))))(((((((((..((((((((....))))))))..))....)))))))((((......)).))...)))..)))))) ( -51.70) >DroVir_CAF1 59409 120 - 1 AGCUGAAUGCCAGAGUCACCCAUGAGGCGGCAAAUGGUGCUGCACGCAUUGAGUUCGUGCCCACCGAAGUGGGCACGCACAUCAUUGAUGCGUCCAUCAAUGGCACGAAGAUCGCUGGCG ........(((((.(((.........((((((.....))))))..((.....((.(((((((((....))))))))).))..((((((((....)))))))))).....)))..))))). ( -50.30) >DroPse_CAF1 30317 120 - 1 AGCUGAACGCCAGAGUCACCCAUGAGGCGGCAAAUGGUGCUGCACGCAUCGAGUUCGUCCCCACGGAAGUGGGAACGCACGUCAUCGAUGCGUCCAUCAAUGGCACGAAGAUAGCUGGUG .......((((((..((..((((((.((((((.....))))))(((((((((((.(((.(((((....))))).))).))....)))))))))...)).))))...))......)))))) ( -50.50) >DroGri_CAF1 69371 120 - 1 AGCUGAAUGCCAGAGUCACCCAUGAGGCGGCAAAUGGUGCUGCACGCAUUGAGUUCGUGCCCACCGAAGUGGGCACGCACAUCAUUGAUGCGUCCAUCAAUGGCACAAAGAUCGCUGGCG ........(((((.(((.........((((((.....))))))..((.....((.(((((((((....))))))))).))..((((((((....)))))))))).....)))..))))). ( -50.30) >DroWil_CAF1 38913 120 - 1 AACUGAAUGCCAGAGUCACCCAUGAGGCGGCAAAUGGUGCUGCACGCAUUGAGUUUGUCCCCACCGAGGUGGGUACCCACAUUAUCGAUGCGUCCAUCAAUGGCACAAAGAUAGCUGGCG ........(((((.(((..((((((.((((((.....))))))(((((((((...(((.(((((....))))).....)))...)))))))))...)).))))......)))..))))). ( -44.10) >DroPer_CAF1 30250 120 - 1 AGCUGAACGCCAGAGUCACCCAUGAGGCGGCAAAUGGUGCUGCACGCAUCGAGUUCGUCCCCACGGAAGUGGGAACGCACGUCAUCGAUGCGUCCAUCAAUGGUACGAAGAUAGCUGGUG .......((((((..((..((((((.((((((.....))))))(((((((((((.(((.(((((....))))).))).))....)))))))))...)).))))...))......)))))) ( -50.50) >consensus AGCUGAACGCCAGAGUCACCCAUGAGGCGGCAAAUGGUGCUGCACGCAUCGAGUUCGUCCCCACCGAAGUGGGCACGCACAUCAUCGAUGCGUCCAUCAAUGGCACGAAGAUAGCUGGCG .......((((((.(((..((((((.((((((.....))))))(((((((((((.(((.(((((....))))).))).))....)))))))))...)).))))......)))..)))))) (-44.23 = -43.98 + -0.25)

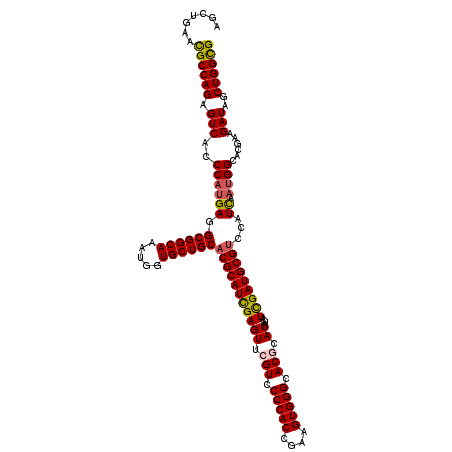
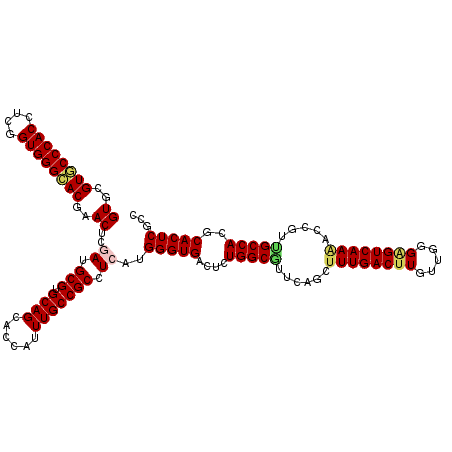
| Location | 18,232,873 – 18,232,993 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 91.50 |
| Mean single sequence MFE | -50.22 |
| Consensus MFE | -42.53 |
| Energy contribution | -42.70 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.21 |
| Structure conservation index | 0.85 |
| SVM decision value | 1.01 |
| SVM RNA-class probability | 0.900549 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 18232873 120 + 20766785 GUGGGUGCCCACCUCGGUGGGCACGAACUCGAUGCGAGCAGCACCAUUUGCCGCCUCGUGGGUGACUCUGGCGUUCAGCUUUGACUUGCUAGGAGUCAAAACUGUCGCCACGCACUCGCC ((..((((((((....))))))))..))..((.(((.((((......))))))).))(((((((....(((((..(((.((((((((.....)))))))).))).)))))..))))))). ( -58.10) >DroPse_CAF1 30357 120 + 1 GUGCGUUCCCACUUCCGUGGGGACGAACUCGAUGCGUGCAGCACCAUUUGCCGCCUCAUGGGUGACUCUGGCGUUCAGCUUUGACUUGUUGGGAGUCAAAACCGUUGCCACGCACUCGCC ((.(((((((((....))))))))).)).(((((((((((((.......((((((....((....))..))))....))((((((((.....))))))))...)))).)))))).))).. ( -49.40) >DroWil_CAF1 38953 120 + 1 GUGGGUACCCACCUCGGUGGGGACAAACUCAAUGCGUGCAGCACCAUUUGCCGCCUCAUGGGUGACUCUGGCAUUCAGUUUUGACUUGUUUGGCGUCAAUACCGAUGCCACGCACUCGCC ((((((.(((((....)))))...(((((.((((((.((.(((.....))).))).((.((....)).))))))).)))))......((.(((((((......))))))).)))))))). ( -45.30) >DroMoj_CAF1 58797 120 + 1 GUGCGUGCCCACCUCGGUGGGCACGAACUCAAUGCGUGCAGCACCAUUUGCCGCCUCAUGGGUGACUCUGGCAUUCAGCUUUGACUUGUUUGGGGUCAAGACCGUUGCCACGCACUCGCC ((.(((((((((....))))))))).))....((((((((((..........(((....((....))..))).......((((((((.....))))))))...)))).))))))...... ( -48.80) >DroAna_CAF1 30617 120 + 1 GUGGGUGCCCACCUCUGUGGGCACGAACUCGAUGCGUGCAGCACCAUUUGCCGCCUCGUGGGUGACUCUGGCGUUGAGCUUUGACUUACUGGGAGUCAAAACCGUGGCCACGCACUCGCC ((((((((........((((.((((..(((((((((.((.(((.....))).))).((.((....)).)))))))))).((((((((.....))))))))..)))).)))))))))))). ( -50.30) >DroPer_CAF1 30290 120 + 1 GUGCGUUCCCACUUCCGUGGGGACGAACUCGAUGCGUGCAGCACCAUUUGCCGCCUCAUGGGUGACUCUGGCGUUCAGCUUUGACUUGUUGGGAGUCAAAACCGUUGCCACGCACUCGCC ((.(((((((((....))))))))).)).(((((((((((((.......((((((....((....))..))))....))((((((((.....))))))))...)))).)))))).))).. ( -49.40) >consensus GUGCGUGCCCACCUCGGUGGGCACGAACUCGAUGCGUGCAGCACCAUUUGCCGCCUCAUGGGUGACUCUGGCGUUCAGCUUUGACUUGUUGGGAGUCAAAACCGUUGCCACGCACUCGCC ((..((((((((....))))))))..))..((.(((.((((......))))))).))..(((((....(((((......((((((((.....)))))))).....)))))..)))))... (-42.53 = -42.70 + 0.17)

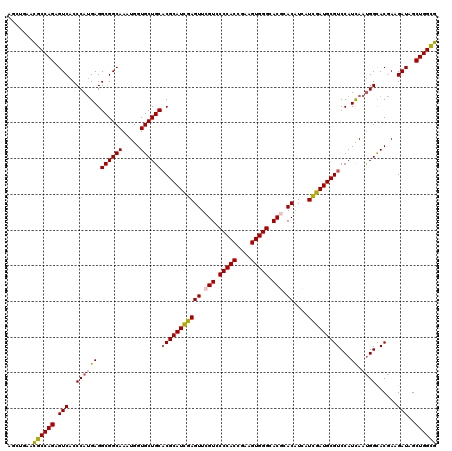
| Location | 18,232,873 – 18,232,993 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 91.50 |
| Mean single sequence MFE | -51.87 |
| Consensus MFE | -44.48 |
| Energy contribution | -44.82 |
| Covariance contribution | 0.34 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.76 |
| Structure conservation index | 0.86 |
| SVM decision value | 1.99 |
| SVM RNA-class probability | 0.985064 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 18232873 120 - 20766785 GGCGAGUGCGUGGCGACAGUUUUGACUCCUAGCAAGUCAAAGCUGAACGCCAGAGUCACCCACGAGGCGGCAAAUGGUGCUGCUCGCAUCGAGUUCGUGCCCACCGAGGUGGGCACCCAC .((((((((((((((.(((((((((((.......)))))))))))..)))))..(((.((.....)).)))......))).)))))).........((((((((....)))))))).... ( -56.10) >DroPse_CAF1 30357 120 - 1 GGCGAGUGCGUGGCAACGGUUUUGACUCCCAACAAGUCAAAGCUGAACGCCAGAGUCACCCAUGAGGCGGCAAAUGGUGCUGCACGCAUCGAGUUCGUCCCCACGGAAGUGGGAACGCAC ..(((.((((((.((.(((((((((((.......)))))))))))..(((((..(((.((.....)).)))...))))).))))))))))).((.(((.(((((....))))).))).)) ( -52.90) >DroWil_CAF1 38953 120 - 1 GGCGAGUGCGUGGCAUCGGUAUUGACGCCAAACAAGUCAAAACUGAAUGCCAGAGUCACCCAUGAGGCGGCAAAUGGUGCUGCACGCAUUGAGUUUGUCCCCACCGAGGUGGGUACCCAC ((..((((((((((((((((.(((((.........))))).)))).)))))...............((((((.....)))))))))))))......((.(((((....))))).)))).. ( -46.20) >DroMoj_CAF1 58797 120 - 1 GGCGAGUGCGUGGCAACGGUCUUGACCCCAAACAAGUCAAAGCUGAAUGCCAGAGUCACCCAUGAGGCGGCAAAUGGUGCUGCACGCAUUGAGUUCGUGCCCACCGAGGUGGGCACGCAC .((.(((((((((((.(((..(((((.........)))))..)))..))))...............((((((.....)))))))))))))......((((((((....)))))))))).. ( -52.00) >DroAna_CAF1 30617 120 - 1 GGCGAGUGCGUGGCCACGGUUUUGACUCCCAGUAAGUCAAAGCUCAACGCCAGAGUCACCCACGAGGCGGCAAAUGGUGCUGCACGCAUCGAGUUCGUGCCCACAGAGGUGGGCACCCAC (((((.((((((.....((((((((((.......))))))))))((.(((((..(((.((.....)).)))...))))).))))))))))).....((((((((....)))))))))).. ( -51.10) >DroPer_CAF1 30290 120 - 1 GGCGAGUGCGUGGCAACGGUUUUGACUCCCAACAAGUCAAAGCUGAACGCCAGAGUCACCCAUGAGGCGGCAAAUGGUGCUGCACGCAUCGAGUUCGUCCCCACGGAAGUGGGAACGCAC ..(((.((((((.((.(((((((((((.......)))))))))))..(((((..(((.((.....)).)))...))))).))))))))))).((.(((.(((((....))))).))).)) ( -52.90) >consensus GGCGAGUGCGUGGCAACGGUUUUGACUCCCAACAAGUCAAAGCUGAACGCCAGAGUCACCCAUGAGGCGGCAAAUGGUGCUGCACGCAUCGAGUUCGUCCCCACCGAGGUGGGCACCCAC ..((((((.(..((..(((((((((((.......)))))))))))...((((..(((.((.....)).)))...))))))..).))).))).....((.(((((....))))).)).... (-44.48 = -44.82 + 0.34)

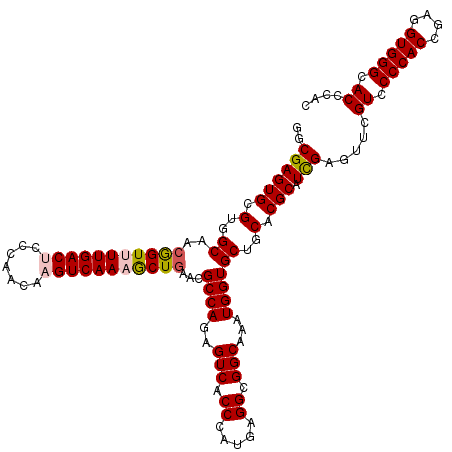
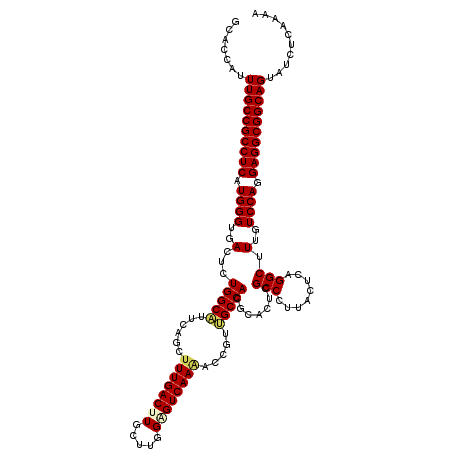
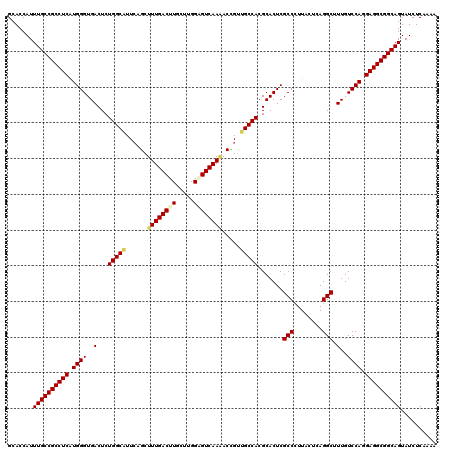
| Location | 18,232,913 – 18,233,033 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.83 |
| Mean single sequence MFE | -47.67 |
| Consensus MFE | -43.81 |
| Energy contribution | -44.00 |
| Covariance contribution | 0.19 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.99 |
| Structure conservation index | 0.92 |
| SVM decision value | 4.02 |
| SVM RNA-class probability | 0.999760 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 18232913 120 + 20766785 GCACCAUUUGCCGCCUCGUGGGUGACUCUGGCGUUCAGCUUUGACUUGCUAGGAGUCAAAACUGUCGCCACGCACUCGCCCUUACUCAGGCUUUGUCCAGGAGGCGGCAGUAUCUCAAAA .......((((((((((.((((..(...(((((..(((.((((((((.....)))))))).))).))))).......(((........))).)..)))).)))))))))).......... ( -52.70) >DroVir_CAF1 59489 120 + 1 GCACCAUUUGCCGCCUCAUGGGUGACUCUGGCAUUCAGCUUUGACUUGCUUGGAGUCAAGACCGUUGCCACGCACUCGCCUUUGCUCAGGCUUUGUCCAGGAGGCGGCAGUAUCUCAAAA .......((((((((((..(((((....(((((..(.(..(((((((.....)))))))..).).)))))..)))))((((......)))).........)))))))))).......... ( -46.20) >DroGri_CAF1 69451 120 + 1 GCACCAUUUGCCGCCUCAUGGGUGACUCUGGCAUUCAGCUUUGACUUGUUGGGUGUCAAAACCGUUGCCACGCACUCGCCUUUACUCAGGCUUUGUCCAGGAGGCGGCAGUAUCUCAAAA .......((((((((((.((((..((..(((((((((((........))))))))))).....))..))........((((......))))......)).)))))))))).......... ( -46.20) >DroWil_CAF1 38993 120 + 1 GCACCAUUUGCCGCCUCAUGGGUGACUCUGGCAUUCAGUUUUGACUUGUUUGGCGUCAAUACCGAUGCCACGCACUCGCCUUUACUCAGGCUUUGUCCAGGAGGCGGCAGUAUCUCAAAA .......((((((((((..((....))((((((.........((..(((.(((((((......))))))).))).))((((......))))..)).)))))))))))))).......... ( -46.80) >DroMoj_CAF1 58837 120 + 1 GCACCAUUUGCCGCCUCAUGGGUGACUCUGGCAUUCAGCUUUGACUUGUUUGGGGUCAAGACCGUUGCCACGCACUCGCCCUUGCUCAGGCUUUGUCCAGGAGGCGGCAGUAUCUCAAAA .......((((((((((..(((((....(((((..(.(..(((((((.....)))))))..).).)))))..)))))(((........))).........)))))))))).......... ( -44.00) >DroAna_CAF1 30657 120 + 1 GCACCAUUUGCCGCCUCGUGGGUGACUCUGGCGUUGAGCUUUGACUUACUGGGAGUCAAAACCGUGGCCACGCACUCGCCCUUGCUCAGGCUUUGUCCAGGAGGCGGCAGUAUCUCAAAA .......((((((((((.((((..(....(((.((((((((((((((.....))))))))...(((....)))..........))))))))))..)))).)))))))))).......... ( -50.10) >consensus GCACCAUUUGCCGCCUCAUGGGUGACUCUGGCAUUCAGCUUUGACUUGCUUGGAGUCAAAACCGUUGCCACGCACUCGCCCUUACUCAGGCUUUGUCCAGGAGGCGGCAGUAUCUCAAAA .......((((((((((.((((..(...(((((......((((((((.....)))))))).....))))).......(((........))).)..)))).)))))))))).......... (-43.81 = -44.00 + 0.19)
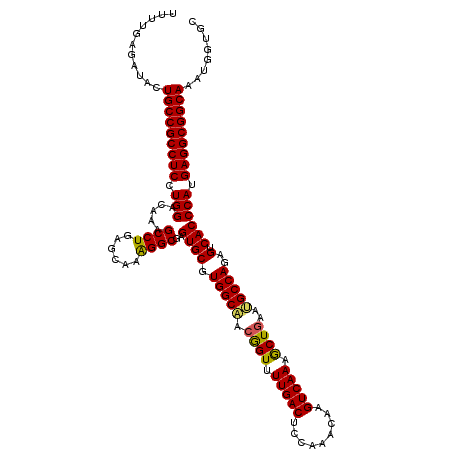
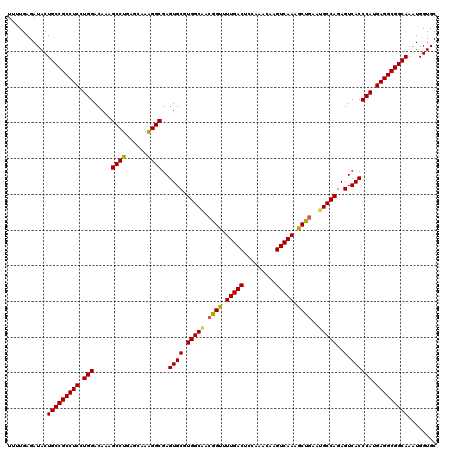
| Location | 18,232,913 – 18,233,033 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.83 |
| Mean single sequence MFE | -50.83 |
| Consensus MFE | -47.87 |
| Energy contribution | -47.45 |
| Covariance contribution | -0.42 |
| Combinations/Pair | 1.12 |
| Mean z-score | -3.75 |
| Structure conservation index | 0.94 |
| SVM decision value | 5.05 |
| SVM RNA-class probability | 0.999971 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>2R_DroMel_CAF1 18232913 120 - 20766785 UUUUGAGAUACUGCCGCCUCCUGGACAAAGCCUGAGUAAGGGCGAGUGCGUGGCGACAGUUUUGACUCCUAGCAAGUCAAAGCUGAACGCCAGAGUCACCCACGAGGCGGCAAAUGGUGC ...........(((((((((.(((.....((((......))))..((((.(((((.(((((((((((.......)))))))))))..)))))..).)))))).)))))))))........ ( -54.30) >DroVir_CAF1 59489 120 - 1 UUUUGAGAUACUGCCGCCUCCUGGACAAAGCCUGAGCAAAGGCGAGUGCGUGGCAACGGUCUUGACUCCAAGCAAGUCAAAGCUGAAUGCCAGAGUCACCCAUGAGGCGGCAAAUGGUGC ...........(((((((((.(((.....((((......))))..((((.(((((.(((..((((((.......))))))..)))..)))))..).)))))).)))))))))........ ( -50.30) >DroGri_CAF1 69451 120 - 1 UUUUGAGAUACUGCCGCCUCCUGGACAAAGCCUGAGUAAAGGCGAGUGCGUGGCAACGGUUUUGACACCCAACAAGUCAAAGCUGAAUGCCAGAGUCACCCAUGAGGCGGCAAAUGGUGC ...........(((((((((.(((.....((((......))))..((((.(((((.((((((((((.........))))))))))..)))))..).)))))).)))))))))........ ( -51.80) >DroWil_CAF1 38993 120 - 1 UUUUGAGAUACUGCCGCCUCCUGGACAAAGCCUGAGUAAAGGCGAGUGCGUGGCAUCGGUAUUGACGCCAAACAAGUCAAAACUGAAUGCCAGAGUCACCCAUGAGGCGGCAAAUGGUGC ...........(((((((((.(((.....((((......))))..((((.((((((((((.(((((.........))))).)))).))))))..).)))))).)))))))))........ ( -51.90) >DroMoj_CAF1 58837 120 - 1 UUUUGAGAUACUGCCGCCUCCUGGACAAAGCCUGAGCAAGGGCGAGUGCGUGGCAACGGUCUUGACCCCAAACAAGUCAAAGCUGAAUGCCAGAGUCACCCAUGAGGCGGCAAAUGGUGC ...........(((((((((.(((.....((((......))))..((((.(((((.(((..(((((.........)))))..)))..)))))..).)))))).)))))))))........ ( -47.90) >DroAna_CAF1 30657 120 - 1 UUUUGAGAUACUGCCGCCUCCUGGACAAAGCCUGAGCAAGGGCGAGUGCGUGGCCACGGUUUUGACUCCCAGUAAGUCAAAGCUCAACGCCAGAGUCACCCACGAGGCGGCAAAUGGUGC ...........(((((((((.(((.....((((......))))..((((.((((...((((((((((.......))))))))))....))))..).)))))).)))))))))........ ( -48.80) >consensus UUUUGAGAUACUGCCGCCUCCUGGACAAAGCCUGAGCAAAGGCGAGUGCGUGGCAACGGUUUUGACUCCAAACAAGUCAAAGCUGAAUGCCAGAGUCACCCAUGAGGCGGCAAAUGGUGC ...........(((((((((.(((.....((((......))))..((((.(((((.((((.(((((.........))))).))))..)))))..).)))))).)))))))))........ (-47.87 = -47.45 + -0.42)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:53:45 2006